In martscht/PsyBSc7: Übungen für die Praktika im Sommersemester 2020
source('setup.R')
Einleitung
Das Paket ggplot2 ist das umfangreichste und am weitesten verbreitete Paket zur Grafikerstellung in R. Seine Beliebtheit liegt vor allem an zwei Dingen: Es ist sehr eng mit der kommerziellen Seite von RStudio verwoben (Autor ist auch hier Hadley Wickham) und es folgt stringent einer "Grammatik der Grafikerstellung". Aus dem zweiten Punkt leitet sich auch sein Name ab: das "gg" steht für "Grammar of Graphics" und geht auf das gleichnamige Buch von Leland Wilkinson zurück, in dem auf 700 kurzen Seiten eine grammatikalische Grundstruktur für das Erstellen von Grafiken zur Datendarstellung hergeleitet und detailliert erklärt wird.
Weil ggplot2 so beliebt ist, gibt es online tausende von Quellen mit Tutorials, Beispielen und innovativen Ansätzen zur Datenvisualisierung. Vom Autor des Pakets selbst gibt es ein Überblickswerk über Data-Science als e-Book, in dem sich auch ein Kapitel mit ggplot2 befasst.
Beispieldaten {#Beispieldaten}
Welches Beispiel wäre zur Zeit naheliegender als die Ausbreitung von COVID-19? Auf GitHub stellen viele Nutzer und Organisationen tagesaktuelle Daten zu Infektionsraten, bestätigten Fällen und vielen anderen Aspekten zur Verfügung. Diese Daten können wir aus R mit einfachen Funktionen wie read.table oder read.csv abrufen.
Dafür nutzen wir die Daten, die von der Johns Hopkins Universität auf GitHub bereitgestellt werden. Für uns sind dabei die zwei Datensätze zu bestätigten Fällen und Todesopfern zentral.
Daten herunterladen
Die erste Möglichkeit die Daten zu beziehen, ist es sie als .csv Dateien herunterzuladen und lokal zu speichern. Dafür reichen diese beiden Links aus: r fontawesome::fa('download') Bestätigte Fälle und r fontawesome::fa('download') Todesfälle.
Statt den Umweg über externe Dateien zu gehen, können wir aber auch direkt aus R heraus die entsprechenden .csv öffnen und als Objekte anlegen. Das hat den Vorteil, dass die Datei jedes mal, wenn das R-Skript ausgeführt wird, auf den aktuellen Stand gebracht wird.
In R können wir dafür einfach die normalen Befehle zum Einlesen von Dateien nutzen. Diese sind auch dafür ausgelegt, dass der Dateipfad sich nicht auf lokale Daten, sondern auf URLs bezieht. Für die erste Datei:
confirmed <- read.csv('https://raw.githubusercontent.com/CSSEGISandData/COVID-19/master/csse_covid_19_data/csse_covid_19_time_series/time_series_covid19_confirmed_global.csv')
Im Environment-Tab sieht man in RStudio die Eigenschaften dieses Datensatzes:
ncol(confirmed)
nrow(confirmed)
Das Gleiche sollten wir jetzt noch für die anderen beiden Dateien machen, bevor wir uns gleich mit dem Format der Daten befassen.
deaths <- read.csv('https://raw.githubusercontent.com/CSSEGISandData/COVID-19/master/csse_covid_19_data/csse_covid_19_time_series/time_series_covid19_deaths_global.csv')
Daten aufbereiten (reshape)
Die Datensätze sind als Zeitreihen aufbereitet. Das bedeutet, dass jede Zeile des Datensatzes eine Region oder ein Land darstellt und jede Spalte des Datensatzes ein Tag ist. Was wir für unsere Visualisierung in ggplot2 benötigen ist ein Datensatz, der alle drei Kennzahlen enthält und dabei für jede Kombination aus Tag und Land eine Zeile enthält. Prinzipiell sollte der resultierende Datensatz also folgende Struktur haben:
| Provinz | Land | Tag | Bestätigt | Verstorben | Genesen |
|:-------:|:----:|:---:|:---------:|:----------:|:-------:|
| Ohio | USA | 21.3. | 248 | 3 | 0 |
| Ohio | USA | 22.3. | 355 | 3 | 0 |
| ... | ... | ... | ... | ... | ... |
| | Germany | 21.3. | 22213 | 84 | 233 |
| | Germany | 22.3. | 24873 | 94 | 266 |
| ... | ... | ... | ... | ... | ... |
Für genau solche Umstellungen gibt es in R die Funktion reshape. Wagen Sie ruhig einen Blick in die Hilfestellung zu der Funktion mit ?reshape. Die Bezeichnungen sind zunächst nicht besonders eingängig, also nutzen wir den Abschnitt mit Beispielen um uns zu orientieren.
Bei der Formatierung von sozialwissenschaftlichen Datensätzen können zwei generelle Typen unterschieden werden: das Long-Format und das Wide-Format. Die Bezeichnung bezieht sich dabei (meistens) auf die Anordnung von Messwiederholungen der gleichen Personen - in unserem Fall sind es keine Personen, sondern Regionen.
Was wir haben ist also ein Datensatz im "breiten" Format (eine Spalte pro Tag) und wir möchten ihn in einen Datensatz im "langen" Format (eine Zeile pro Tag) übertragen. Um die vorliegenden Daten ins lange Format zu überführen, benötigt die Funktion mindestens fünf Argumente:
data: Der Datensatzvarying: Die Variablen die wiederholt gemessen wurdenv.names: Der Name unter dem die Variablen zusammengefasst werden sollentimevar: Die Variable, die Wiederholungen kennzeichnetidvar: Die Variablen, die sich über Wiederholungen nicht änderndirection: Das Zielformat des neuen Datensatzes
Für den ersten Datensatz sollte die Syntax also wie folgt aussehen:
confirmed_long <- reshape(confirmed,
varying = names(confirmed)[-c(1:4)],
v.names = 'Confirmed',
timevar = 'Day',
idvar = names(confirmed)[1:4],
direction = 'long')
Wir nutzen den Datensatz confirmed. Wiederholt gemessen wurden alle Variablen außer die ersten 4 (Provinz, Land, Länge- und Breitengrad). Die neue Variable nennen wir Confirmed, damit wir gleich Verwirrung umgehen, wenn wir die drei Datensätze zusammenführen. Unsere Zeitvariable ist der Tag, also benennen wir sie mit Day. Als Identifikatoren halten unsere ersten 4 Variablen her (weil diese den Ort eindeutig bestimmen und sich nicht über die Zeit ändern) und die Zielausrichtung des Datensatzes ist "lang". Der Datensatz sollte also jetzt wie folgt aussehen:
head(confirmed_long)
Die Zeilennamen bewirken, dass der Datensatz etwas unübersichlich wirkt. Wir können diese ganz einfach entfernen:
rownames(confirmed_long) <- NULL
head(confirmed_long)
Bevor jetzt die beiden Datensätze zusammenführen können, muss auch deaths ins Long-Format übertragen werden. Sie können unten probieren, ob das Vorgehen zum gewünschten Ergebnis führt. Sie können auch Änderungen vornehmen, um zu sehen, wie sich das Ergebnis verändert.
deaths_long <- reshape(deaths,
varying = names(deaths)[-c(1:4)],
v.names = 'Deaths',
timevar = 'Day',
idvar = names(deaths)[1:4],
direction = 'long')
rownames(deaths_long) <- NULL
head(deaths_long)
Daten zusammenführen (merge)
Die beiden Datensätze, die wir jetzt erzeugt haben bestehen jeweils aus vier Variablen zur Region (Province.State, Country.Region, Lat und Long) einer Variable zur Zeit (Day) und der Anzahl der entsprechenden Fälle (Confirmed bzw. Deaths). Um die letzten beiden in einem Datensatz zusammenzuführen können wir die ersten fünf nutzen um eindeutig festzulegen, welche Daten zusammengehören. Daten vom gleichen Ort zur gleichen Zeit beziehen sich üblicherweise auf das Gleiche. Für das Zusammenführen von zwei Datensätzen gibt es in R den merge Befehl, der drei zentrale Argumente entgegennimmt:
x: Der erste Datensatzy: Der zweite Datensatzby: Die Variablen, anhand derer gleiche Fälle identifiziert werden können
In der Hilfe zur Funktion finden sich auch noch viele zusätzliche Argumente, aber diese Drei sind für uns zentral.
long <- merge(confirmed_long, deaths_long,
by = c('Province.State', 'Country.Region', 'Lat', 'Long', 'Day'))
head(long)
Daten zusammenfassen (aggregate)
Leider ist die Auflösung mit der die unterschiedlichen Länder erhoben werden nicht einheitlich. Für Australien liegen beispielsweise Daten aus 9 Bundesstaaten vor, für die USA sogar aus 247 Provinzen. Um die Daten einheitlich auf der Ebene von Nationen betrachten zu können, müssen sie aggregiert werden. In unserem Fall möchten wir also die Summe über z.B. die 9 Australischen Bundesstaaten bilden. Dafür gibt es in R sehr viele verschiedene Lösungsansätze. Einer davon ist der aggregate Befehl, den wir hier nutzen werden.
Um nun aggregieren zu können, können wir aggregate eine Formel geben, wie wir sie schon für Regressionen im letzten Semester verwendet haben. Die generelle Struktur solcher Formeln ist immer AV ~ UV, wobei mehrere UVs durch + vebunden werden können. In unserem Fall behandeln wir die zwei Spalten Confirmed und Deaths als AVs und Contry.Region plus Day als UVs. Zusätzlich benötigt der Befehl dann eine Auskunft darüber, auf welchen Datensatz wir uns beziehen und mit welcher Funktion wir aggregieren wollen:
covid <- aggregate(cbind(Confirmed, Deaths) ~ Country.Region + Day, data = long, FUN = 'sum')
head(covid)
Der Datensatz enthält jetzt bestätigte Fälle, Todesfälle und genesene Fälle pro Land und für jeden Tag und ist damit bereit für die Visualisierung. Sie können das Feld unten nutzen, um sich noch einen besseren Überblick über die Datenlage zu verschaffen. Der Beispielcode zeigt Ihnen die Daten der letzten 10 Tage für Deutschland:
covid[covid$Country.Region == 'Germany' & covid$Day > max(covid$Day)-10, ]
ggplot2 Grundprinzipien {#Grundprinzipien}
In ggplot2 werden immer Daten aus einem data.frame dargestellt. Das heißt, dass wir nicht, wie bei plot oder hist aus R selbst, Vektoren oder Matrizen nutzen können. Daten müssen immer so aufbereitet sein, dass der grundlegende Datensatz sinnvoll benannte Variablen enthält und in dem Format vorliegt, in dem wir die Daten visualisieren wollen. Das hat zwar den Nachteil, dass wir Datensätze umbauen müssen, wenn wir Dinge anders darstellen wollen, aber hat auch den Vorteil, dass wir alle Kenntnisse über Datenmanagement im Allgemeinen auf den Umgang mit ggplot2 übertragen können. Deswegen haben wir auch im vergangenen Abschnitt so viel Zeit damit verbracht die Daten in das korrekte Format zu überführen.
Bevor wir loslegen können, muss natürlich ggplot2 installiert sein und geladen werden:
library(ggplot2)
Im Kern bestehen Abbildungen in der Grammatik von ggplot2 immer aus drei Komponenten:
- Daten, die angezeigt werden sollen
- Geometrie, die vorgibt welche Arten von Grafiken (Säulendiagramme, Punktediagramme, usw.) genutzt werden
- Ästhetik, die vorgibt, wie die Geometrie und Daten aufbereitet werden (z.B. Farben)
In den folgenden Abschnitten werden wir versuchen diese drei Komponenten so zu nutzen, dass wir informative und eventuell auch ansehnliche Abbildungen generieren.
Schichten
In ggplot2 werden Grafiken nicht auf einmal mit einem Befehl erstellt, sondern bestehen aus verschiedenen Schichten. Diese Schichten werden meistens mit unterschiedlichen Befehlen erzeugt und dann so übereinandergelegt, dass sich am Ende eine Abbildung ergibt.
Die Grundschicht sind die Daten. Dafür haben wir im vorherigen Abschnitt covid als Datensatz aufbereitet. Benutzen wir zunächst nur die Daten für Deutschland um nicht unnötig die Perspektive zu verzerren:
covid_de <- covid[covid$Country.Region == 'Germany', ]
ggplot(covid_de)
Was entsteht ist eine leere Fläche. Wie bereits beschrieben, besteht eine Abbildung in ggplot2 immer aus den drei Komponenten Daten, Geometrie und Ästhetik. Bisher haben wir nur eine festgelegt. Als erste Ästhetik sollten wir festlegen, welche Variablen auf x- und y-Achse dargestellt werden sollen. Nehmen wir naheliegenderweise die Zeit (x-Achse) und die Zahl der bestätigten Fälle:
ggplot(covid_de, aes(x = Day, y = Confirmed))
Ästhetik wird in ggplot2 über den aes-Befehl erzeugt. Jetzt fehlt uns noch die geometrische Form, mit der die Daten abgebildet werden sollen. Für die Geometrie-Komponente stehen in ggplot2 sehr viele Funktionen zur Verfügung, die allesamt mit geom_ beginnen. Eine Übersicht über die Möglichkeiten finden Sie z.B. hier. Naheliegende Möglichkeiten für den Zeitverlauf sind eine Linie (geom_line) und mehrere Punkte (geom_point). Neue Schichten werden in ihrer eigenen Funktion erzeugt und mit dem einfachen + zu einem bestehenden Plot hinzugefügt.
Im Folgenden ist eine Linie die genutzte Geometrie - probieren Sie aus, wie das Punktediagramm aussieht oder eine andere Geometrie funktioniert, die Ihnen gefällt!
ggplot(covid_de, aes(x = Day, y = Confirmed)) + geom_line()
Der immense Vorteil des Schichtens besteht darin, dass wir gleichzeitig mehrere Visualisierungsformen nutzen können. Das Prinizp bleibt das gleiche wie vorher: wir fügen Schichten mit dem + hinzu. Probieren Sie die Kombination aus Punkten und Linie im Beispiel oben aus!
Plots als Objekte
Einer der Vorteile, die sich durch das Schichten der Abbildungen ergibt ist, dass wir Teile der Abbildung als Objekte definieren können und sie in verschiedenen Varianten wieder benutzen können. Das hilft besonders dann, wenn wir unterschiedliche Geometrie in einer gemeinsamen Abbildung darstellen wollen oder z.B. erst einmal eine Abbildung definieren wollen, bevor wir Feinheiten adjustieren.
basic <- ggplot(covid_de, aes(x = Day, y = Confirmed))
In basic wird jetzt die Anleitung für die Erstellung der Grafik gespeichert. Erstellt wird die Grafik aber erst, wenn wir das Objekt aufrufen. Dabei können wir das Objekt auch mit beliebigen anderen Komponenten über + kombinieren. Probieren Sie es unten aus, mit Objekt-Kombinationen zu arbeiten!
basic + geom_point()
Damit die Beispiele im weiteren Verlauf auch selbstständig funktionieren, wird unten immer der gesamte Plot aufgeschrieben. Aber für Ihre eigenen Übungen oder Notizen ist es durchaus praktischer mit dieser Objekt Funktionalität zu arbeiten, um so zu umgehen, dass man immer wieder die gleichen Abschnitte aufschreiben muss.
Farben und Ästhetik {#Farben}
Oben wurde erwähnt, dass Ästhetik die dritte Komponente ist und als Beispiel Farbe genannt. Das stimmt nicht immer: die Farbe der Darstellung muss nicht zwingend eine Ästhetik sein. Gucken wir uns zunächst an, wie es aussieht, wenn wir die Farbe der Darstellung ändern wollen:
ggplot(covid_de, aes(x = Day, y = Confirmed)) + geom_point(color = 'blue')
Alle Punkte haben die Farbe geändert. Eine Ästhetik im Sinne der ggplot-Grammatik ist immer abhängig von den Daten. Die globale Vergabe von Farbe ist also keine Ästhetik. Sie ist es nur, wenn wir sie von Ausprägungen der Daten abhängig machen. Das funktioniert z.B. so:
ggplot(covid_de, aes(x = Day, y = Confirmed)) + geom_point(aes(color = Confirmed))
Über den Befehl aes definieren wir eine Ästhetik und sagen ggplot, dass die Farbe der Punkte von der Ausprägung auf der Variable Confirmed abhängen soll. Die Farbe kann aber natürlich auch von jeder anderen Variable im Datensatz abhängen. Wie das aussehen kann gucken wir uns im kommenden Abschnitt an.
Gruppierte Abbildungen
Im letzten Abschnitt hatten wir die Daten auf Fälle aus Deutschland reduziert. In diesem Abschnitt wollen wir gleichzeitig mehrere Länder betrachten können. Dafür müssen wir wieder zunächst die Daten auswählen, die relevant sind. Wir beschränken uns in diesem Fall auf Deutschland, Italien, Frankreich, das Vereinigte Königreich und Spanien.
covid_sel <- covid[covid$Country.Region %in% c('France', 'Germany', 'Italy', 'Spain', 'United Kingdom'), ]
Wenn wir jetzt mit dem gleichen Vorgehen wie oben die Abbildung erstellen, wird es etwas chaotischer, weil unklar ist, welche Punkte sich auf welches Land beziehen:
ggplot(covid_sel, aes(x = Day, y = Confirmed)) + geom_point()
Um das zu umgehen, können wir natürlich die Ästhetik der Farben benutzen:
ggplot(covid_sel, aes(x = Day, y = Confirmed)) + geom_point(aes(color = Country.Region))
Wie Sie sehen ergibt sich automatisch eine Legende auf der rechten Seite, die jedem Land eine Farbe zuweist. Wir können auch hier wieder eine Kombination aus Punkten und Linien nutzen:
ggplot(covid_sel, aes(x = Day, y = Confirmed)) +
geom_point(aes(color = Country.Region)) +
geom_line(aes(color = Country.Region))
Das Problem ist hier, dass wir die Ästhetik für jede Geomtrie wiederholen müssen. Stattdessen können wir in ggplot auch allgemein eine Gruppierung vornehmen, die für alle Geometrien übernommen wird:
ggplot(covid_sel, aes(x = Day, y = Confirmed, color = Country.Region)) +
geom_point() + geom_line()
Wenn Sie das Ganze auch mit anderen Ländern ausprobieren möchten, können Sie das folgende Feld nutzen um erst Daten auszuwählen und dann einen Plot zu erstellen.
Mit "Hint" können Sie sich ein paar Hinweise ausgeben lassen.
# Ländernamen ausgeben lassen
levels(covid$Country.Region)
# 10 Länder mit den meisten Fällen auswählen
today <- covid[covid$Day == max(covid$Day), ]
filt <- today[rank(1/today$Confirmed) <= 10, 'Country.Region']
covid_10 <- covid[covid$Country.Region %in% filt, ]
Faceting
Alle Länder in der gleichen Abbildung darzustellen kann mitunter sehr unübersichtlich werden. Eine Möglichkeit, Übersichtlichkeit zu bewahren ist das sogenannte Faceting. Dabei wird eine Abbildung anhand von Ausprägungen auf einer oder mehr Variablen in verschiedene Abbildungen unterteilt. Mit dem gleichen Datensatz aus Deutschland, Italien, Frankreich, dem Vereinigten Königreich und Spanien können wir die Abbildung anhand des Landes einteilen:
ggplot(covid_sel, aes(x = Day, y = Confirmed)) +
geom_point() + geom_line() +
facet_wrap(~ Country.Region)
In facet_wrap wird wieder mit der R Gleichungsnotation gearbeitet: hier wird der Plot anhand der unabhängigen Variablen hinter der Tilde in Gruppen eingeteilt. Das gibt auch wieder die Möglichkeit mit + mehrere Variablen zu definieren, die zum Faceting benutzt werden sollen. Wenn Sie Gruppen anhand von zwei Variablen bilden bietet es sich außerdem an facet_grid zu benutzen.
Per Voreinstellung wird beim Faceting eine gemeinsame Skalierung der x- und y-Achsen für alle Teilabbildungen festgelegt. Das kann mit dem Argument scales in der facet_wrap Funktion umgangen werden. Bei ?facet_wrap finden Sie dafür genauere Informationen.
Mehrere Variablen
Bisher haben wir auf der x-Achse nur die Zeit und auf der y-Achse nur die bestätigten Fälle betrachtet. Durch das Schicht-System können wir auf unsere bisherigen Abbildungen aber auch zusätzliche Variablen - oder sogar komplett andere Datensätze - abbilden. Wenn wir uns z.B. die bestätigten und die Todesfälle in Deutschland angucken wollen, können wir wie folgt vorgehen:
ggplot(covid_de, aes(x = Day)) +
geom_line(aes(y = Confirmed), color = 'darkblue') +
geom_line(aes(y = Deaths), color = 'darkred')
Abbildungen anpassen {#Anpassen}
Die Abbildungen, die wir bisher erstellt haben nutzen alle das in ggplot2 voreingestellt Design. Auch wenn es sicherlich einen theoretisch sehr gut fundierten Grund gibt, dass der Hintergrund der Abbildung in einem demotivierenden Grauton gehalten sein sollte, gibt es Designs, die man schöner finden kann. Im folgenden gucken wir uns an, wie man seine Abbildungen nach seinen eigenen Vorlieben anpassen kann.
Themes {#Themes}
In ggplot2 werden die Grundeigenschaften von Abbildungen in "Themes" zusammengefasst. Mit ?theme_test erhalten Sie eine Auflistung aller Themes, die von ggplot2 direkt zur Verfügung gestellt werden. Noch mehr Themes gibt es im Paket ggthemes, welches Sie zusätzlich installieren können.
Um das Theme einer Abbildung zu verändern, können Sie es - wie Geometrie - mit dem + hinzufügen. Probieren Sie unten ein paar Themes aus und finden Sie Ihren Liebling!
ggplot(covid_sel, aes(x = Day, y = Confirmed, color = Country.Region)) +
geom_line() + geom_point() +
theme_light()
Sie können natürlich nicht nur vordefinierte Themes nutzen, sondern über theme jeden Aspekt der Aufbereitung der Abbildung selbst steuern. Mit ?theme sehen Sie die ausufernde Breite an Möglichkeiten, die Ihnen hier gegeben wird.
Beschriftung
Eine der wichtigsten Komponenten jeder Abbildung ist die Beschriftung. Nur wenn ausreichend gut gekennzeichnet ist, was wir darstellen, können wir darauf hoffen, dass die Information vermittelt wird, die wir vermitteln wollen. Die folgenden Befehle von ggplot2 beziehen sich spezifisch auf Beschriftungen, Sie können diese Änderungen aber auch allesamt mit der oben erwähnten theme-Funktion erreichen.
Zunächst ist es sinnvoll die Achsen ordentlich zu beschriften. Per Voreinstellung werden hierzu die Namen der Variablen genutzt. Wir können also eine nützliche Beschriftung schon früh dadurch erzwingen, dass wir die Variablen im Datensatz ordentlich benennen. Besonders wenn die Achsen aber Zusatzinformationen (wie z.B. "(in %)") enthalten sollen, ist es aber unumgänglich die Benennung hinterher zu ergänzen. Darüber hinaus kann es sinnvoll sein, einer Grafik Titel und Untertitel zu geben.
ggplot(covid_sel, aes(x = Day, y = Confirmed, color = Country.Region)) +
geom_line() + geom_point() +
theme_light() +
labs(x = 'Tage seit dem 22.1.', y = 'Bestätigte Fälle') +
ggtitle('COVID-19 Infektionen', paste('Stand:', Sys.Date()))
Im Untertitel ist dargestellt, dass wir auch für Beschriftungen wieder Objekte und Werte aus Objekten nutzen können, die in R vorliegen. Hier wird über Sys.Date() das aktuelle Datum abgefragt und durch paste mit "Stand:" zusammengeklebt.
Der Befehl labs bezieht sich auf die Beschriftung von jeder Ästhetik in der Abbildung. In der aktuellen Variante ist die Legende der Länder mit Country.Region nicht sonderlich schön benannt. Versuchen Sie unten diese Beschriftung über den labs Befehl in etwas Sinnvolleres zu ändern!
ggplot(covid_sel, aes(x = Day, y = Confirmed, color = Country.Region)) +
geom_line() + geom_point() +
theme_light() +
labs(x = 'Tage seit dem 22.1.', y = 'Bestätigte Fälle') +
ggtitle('COVID-19 Infektionen', paste('Stand:', Sys.Date()))
Farbpaletten
In ggplot2 wird die Vergabe von Farben in der Ästhetik anhand von zwei Dingen unterschieden: der Geometrie und dem Skalenniveau der Variable, die die Färbung vorgibt.
In den bisherigen Beispielen im Abschnitt Farben und Ästhetik haben wir die Unterschiede bezüglich des Skalenniveaus schon gesehen: Kontinuierliche Variablen (Variablen, die in R als numeric definiert sind) werden anhand eines Blau-Farbverlaufs dargestellt, diskrete Variablen (Variablen, die in R als factor definiert sind) anhand eines vordefinierten Schemas unterschiedlicher Farben. Dieses Schema ist das Brewer Farbschema, welches usprünglich für Kartendarstellungen entwickelt wurde.
Bezüglich Geometrie wird bei der Färbung zwischen fill und color unterschieden - also ob eine Geometrie mit einer Farbe gefüllt wird oder ihr Rand mit dieser Farbe gezeichnet wird. In den bisherigen Abbildungen haben wir noch kein Beispiel gehabt, in dem etwas gefüllt werden könnte, aber in den Aufgaben zu dieser Sitzung könnte das noch relevant werden.
Nehmen wir an, dass wir unsere Abbildung irgendwo drucken möchten - Farbdruck ist wahnsinnig teuer. Um mit Grautönen zu arbeiten, können wir z.B. scale_color_grey benutzen:
ggplot(covid_sel, aes(x = Day, y = Confirmed, color = Country.Region)) +
geom_line() + geom_point() +
theme_light() +
scale_color_grey()
Das bei den Themes erwähnte Paket ggthemes enthält auch weitere Farbpaletten, die Sie nutzen können, um Ihren Plot nach Ihren Vorlieben zu gestalten. Wichtig ist beispielsweise, dass es eine Palette namens colorblind hat, die Farben so auswählt, dass sie auch von Personen mit Farbblindheit differenziert werden können. In Fällen mit 6 oder weniger Gruppen bietet sich darüber hinaus in solchen Fällen an mit der Ästhetik pch (für plotcharacter). Darüber hinaus gibt es für Fans der Filme von Wes Anderson z.B. das Paket wesanderson, welches für jeden seiner Filme die Farbpalette parat hat. Darüber hinaus können wir aber natürlich auch unsere ganz eigene Farbpalette definieren - z.B. die offizielle Farbpalette des Corporate Designs der Goethe Universität, die Sie auf den Folien von PsyBSc 1 und 2 im letzten Semester kennen (und lieben!) gelernt haben.
Für diese Palette können wir zunächst in einem Objekt die Farben festhalten, die wir benötigen. In ggplot2 ist es dabei am gängigsten, Farben entweder über Worte auszuwählen oder via hexadezimaler Farbdefinition zu bestimmen. Für die fünf Farben, die von der Corporate Design Abteilung der Goethe Uni definiert werden ergibt sich folgendes Objekt:
gu_colors <- c('#00618f', '#e3ba0f', '#ad3b76', '#737c45', '#c96215')
Dieses Objekt können wir dann nutzen um mit scale_color_manual selbstständig Farben zuzuweisen:
ggplot(covid_sel, aes(x = Day, y = Confirmed, color = Country.Region)) +
geom_line() + geom_point() +
theme_light() +
scale_color_manual(values = gu_colors)
Die Zuordnung der Farben erfolgt anhand der Reihenfolge in gu_colors und der Reihenfolge der Ausprägungen von Country.Region. Letztere ist - wie sie bestimmt festgestellt haben - alphabetisch. Wie häufig in ggplot2 können Sie die Daten ändern (also mit relevel die Reihenfolge der Ausprägungen ändern) um Veränderungen in der Darstellung zu bewirken.
Verschiedene Plots
Bisher haben wir ausschließlich Punkte- und Liniendiagramme benutzt, um die Daten darzustellen. Im letzten Semester haben Sie darüber hinaus noch einige Arten von Diagrammen kennengelernt, die in der psychologischen Forschung extrem verbreitet sind. Ein paar davon werden auch in diesem Abschnitt erstellt.
Balkendiagramme
Häufigkeiten von nominalskalierten Variablen werden typischerweise in Balkendiagrammen dargestellt. Dazu benutzen wir in diesem Beispiel die Anzahl bestätigter Fälle zum jetzigen Zeitpunkt. Zunächst müssen wir also wieder den Datensatz so umstellen, dass er nur die heutigen Fallzahlen enthält. Weil in der bisherigen Variante von covid_sel die Tage fortlaufend nummeriert sind, können wir die heutigen Zahlen einfach anhand des Maximalwertes des Tages extrahieren:
covid_today <- covid_sel[covid_sel$Day == max(covid_sel$Day), ]
covid_today
Um diese Daten abzubilden könnten wir jetzt theoretisch wie bisher vorgehen, lediglich die Variable auf x-Achse und die genutzte Geometrie ändern sich:
ggplot(covid_today, aes(x = Country.Region, y = Confirmed)) +
geom_bar()
Der Fehler verrät uns, dass ggplot2 davon ausgeht, dass wir die Häufigkeit einer nominalskalierten Variablen (stat_count) auszählen lassen wollen. Das wäre dann z.B. der Fall, wenn wir einen Datensatz hätten, der Personen enthält und auf einer Variable das Geschlecht kodiert wird. In dem Fall würde ggplot2 die Häufigkeit der verschiedenen Ausprägungen auszählen und uns diese Plotten.
In unserem Fall haben wir allerdings eine Variable, die die Häufigkeiten enthält, sodass wir dieses Verhalten unterdrücken müssen. Das erreichen wir dadurch, dass wir als "zu berechnende Statistik" statt dem Auszählen (stat = 'count') die schon vorliegende Statistik (stat = 'identity') benutzen.
ggplot(covid_today, aes(x = Country.Region, y = Confirmed)) +
geom_bar(stat = 'identity')
Diese Art der Abbildung kann natürlich auch wieder den Möglichkeiten verbunden werden, die wir im Abschnitt Abbildungen Anpassen besprochen hatten. Probieren Sie es doch direkt aus! Sie können z.B. ein Theme mit einem weißen Hintergrund wählen und die Balken nach Land farbig füllen lassen.
ggplot(covid_today, aes(x = Country.Region, y = Confirmed)) +
geom_bar(stat = 'identity')
Histogramme
Die Übertragung der Balkendiagramme auf mindestens intervallskalierte Variablen hatten wir letztes Semester in Form von Histogrammen behandelt. Dafür wird eine intervallskalierte Variable in verschiedene Intervalle eingeteilt und die Häufigkeit angezeigt, mit der Ausprägungen auf der Variable in ein Intervall fallen. Wir können uns dafür die Anzahl der bis heute bestätigten Fälle über alle Länder hinweg ansehen. Wie üblich, müssen wir dafür zunächst die geeigneten Daten auswählen:
covid_global <- covid[covid$Day == max(covid$Day), ]
Dann können wir die Daten mit geom_histogram ganz einfach darstellen. Der einzige Unterschied ist, dass jetzt die uns interessierende Variable auf der x- und nicht mehr auf der y-Achse abgebildet wird.
ggplot(covid_global, aes(x = Confirmed)) +
geom_histogram()
ggplot2 macht uns darauf aufmerksam, dass wir keine Breite der Intervalle (binwidth) bzw. keine Anzahl der Intervalle (bins) angegeben haben und daher 30 Intervalle genutzt werden. Mit den Argumenten binwidth bzw. bins können wir hier händische Einstellungen vornehmen.
Boxplots
Um die Verteilung von Variablen zu untersuchen, werden in den Sozialwissenschaften häufig Boxplots genutzt. In ggplot2 werden diese entsprechend über geom_boxplot erstellt. Wir können also die gleichen Informationen wie im letzten Abschnitt auch in Form eines Boxplots darstellen:
ggplot(covid_global, aes(y = Confirmed)) +
geom_boxplot()
Plots mit Trendlinien
In den Abschnitten ggplot2 Grundprinzipien und Abbildungen anpassen haben wir uns mit dem sehr verbreiteten Scatterplot befasst. In diesen Plots werden zwei Variablen zueinander ins Verhältnis gestellt - bei uns war das die Zeit auf der x-Achse und die Anzahl bestätigter Fälle auf der y-Achse. Typischerweise wollen wir aus solchen Plots auch einen Trend erkennen können. Im Fall von COVID-19 ist dieser Trend auch in den Rohdaten sehr leicht erkennbar, aber das ist nicht immer der Fall. Mit geom_smooth können wir uns eine "Glättung" der Datenlagen anschauen, die den generellen Trend verdeutlicht. Per Voreinstellung wird hierzu die sogenannte LOESS-Glättung genutzt.
ggplot(covid_sel, aes(x = Day, y = Confirmed)) +
geom_point() +
geom_smooth()
Was eingezeichnet wird ist die globale Trendlinie. Die schattierte Region um diese Linie herum stellt den Standardschätzfehler dieser Kurve dar. Um die Trends länderspezifisch einzuzeichnen, können wir erneut mit der Ästhetik color arbeiten, um die Länder in allen Geometrien farblich voneinander abzugrenzen:
ggplot(covid_sel, aes(x = Day, y = Confirmed, color = Country.Region)) +
geom_point() +
geom_smooth()
Aufgaben
Zur Bearbeitung der Aufgaben öffnen Sie bitte ein neues RStudio-Fenster. Es werden die Daten benötigt, wie Sie in Beispieldaten erzeugt wurden. Sie können entweder die Syntax von dort kopieren und sie Ihrem Skript hinzufügen, oder den Befehl PsyBSc7::Aufgaben_2() ausführen um die Daten direkt im Workspace anlegen zu lassen.
In der Berichterstattung zur aktuellen COVID-19 Ausbreitung stehen vor allem europäische Länder, Süd-Korea, China und die USA im Zentrum. Um mal eine andere Gegend der Erde in den Fokus zu Rücken, betrachten wir in den Aufgaben die Ausbreitung in Südostasien. Untenstehende Tabelle stellt ein paar Informationen über die 11 Südostasiatischen Länder zusammen (Quelle):
| Land | Bevölkerung (in 100 000) | Human Development Index |
|:----:|:------------------------:|:-----------------------:|
| Brunei | 4 | .845 |
| Indonesien | 2677 | .707 |
| Kambodscha | 167 | .581 |
| Laos | 71 | .604 |
| Malaysia | 315 | .804 |
| Myanmar | 537 | .578 |
| Osttimor | 12 | .626 |
| Philippinen | 1067 | .712 |
| Singapur | 58 | .935 |
| Thailand | 694 | .765 |
| Vietnam | 955 | .693 |
- Erstellen Sie einen Datensatz mit dem Namen
covid_se, der nur die Daten dieser Länder enthält (Myanmar wird im englischsprachigen Raum weiterhin offiziell als "Burma" geführt).
- Die Darstellung absoluter Fallzahlen wird häufig als irreführend angesehen, weil Länder mit größerer Bevölkerung mehr mögliche Fälle habe. Nutzen Sie obenstehende Tabelle um die Anzahl bestätigter Fälle an der Bevölkerung zu relativieren und so die bestätigten Fälle pro 100 000 Einwohner zu erhalten. Nennen sie diese Variable
Per100k.
- Stellen Sie die Bestätigten Fälle pro 100 000 Einwohner in Abhängigkeit von der Zeit in einer Abbildung dar. Nutzen Sie Farben um zwischen den Ländern zu unterscheiden.
- Stellen Sie für jedes Land in einer Teilabbildung die bestätigten Fälle (
Confirmed), und die Todesfälle (Deaths) dar. Nutzen sie für die beiden Linien unterschiedliche Farben (aber über alle Länder hinweg die gleichen). Weil sich die Bevölkerungszahlen so drastisch unterscheiden, lassen sie die y-Achse über die Länder hinweg frei variieren.
- Nutzen Sie ein Balkendiagramm, um die relative Häufigkeit verstorbener Personen (
Deaths) and der Gesamtzahl besätigter Fälle (Confirmed) pro Land bis zum heutigen Tag darzustellen. Denken Sie daran, dass Sie dafür Daten aggregieren müssen. Füllen Sie die Balken dabei mit länderspezifischen Farben.
Bonusplots
Weil auch ich mich in dieser Phase der eingeschränkten Freizeitmöglichkeiten beschäftigen muss, habe ich für Sie im Folgenden ein paar zusätzliche Abbildungen erstellt, die auch Sie zum gleichen Datensatz mit ggplot2 und Erweiterungen erstellen können. Diese gehen über das Ausmaß dessen hinaus, was wir im Rahmen dieses Semesters besprechen, aber falls Sie die grafische Darstellung von Daten interessiert, sollten Sie aber von diesen Möglichkeiten gehört haben.
Animationen
Was sich bei Verlaufsdaten anbietet ist es, diese auch so zu animieren, dass der Verlauf deutlich wird. Für Animationen gibt es für ggplot2 das Erweiterungspaket gganimate, das Ihnen die Möglichkeit bietet, normale ggplots um eine Achse - die Zeit - zu erweitern. Zuerst aber müssen wir gganimate laden:
library(gganimate)
Nehmen wir wieder den Plot der Verläufe der bestätigten Fälle. Dieser stellt jetzt den "statischen" Anteil unserer Abbildung dar, daher können wir ihn einfach static nennen:
static <- ggplot(covid_sel, aes(x = Day, y = Confirmed, color = Country.Region)) +
geom_line() + geom_point() +
theme_light() + scale_color_discrete('Country')
Im Gegensatz zu den bisherigen Abbildungen ändern wir außerdem den Titel der Legende zu "Country", um das Ganze ein wenig aufzuhübschen. In static ist jetzt zunächst der gleiche Plot enthalten, wie bisher:
static
Um Animationen zu generieren, können wir mit den Funktionen von gganimate eine neue Schicht - die der Übergänge - erzeugen. Alle Funktionen, mit denen man durch das Pakte Animationen erzeugen kann beginnen mit transition_ - ähnlich wie im Kern-ggplot2 alle Geometrien mit geom_ beginnen.
Um in einer Animation nach und nach die Werte der einzelnen Tage anzuzeigen, können wir die Daten schrittweise "aufdecken" - der dazugehörige Befehl heißt also transition_reveal. Um das mit unserem bisher statistischen Plot zu verbinden, benutzen wir in ggplot Tradition weiterhin das +:
fluid <- static + transition_reveal(Day)
Die Funktion transition_reveal möchte als Argument wissen, anhand welche Variable des Datensatzes wir die Zeitachse kodieren. Bei uns ist das der Tag, also Day. Wenn Sie jetzt fluid aufrufen würden, würde nach ca. 20 Sekunden renderzeit eine Animation in RStudio erscheinen. Wir fügen der Abbildung aber zunächst noch einen Titel und Untertitel hinzu. Dabei soll der Untertitel den jeweiligen Tag angeben, sich also über die Zeit ebenfalls verändern. In der Hilfe zu transition_reveal finden Sie den Abschnitt Label variables, der Ihnen verrät, welche Werte von der Animation ausgegeben werden und z.B. in die Benennung mit eingebunden werden können. In diesem Fall heißt diese Variable frame_along. Diese können wir in den Untertitel einbinden:
fluid <- static + transition_reveal(Day) +
ggtitle('Confirmed COVID-19 Cases', subtitle = 'Day {frame_along}')
Somit wird in der Animation im Untertitel immer "Day X" stehen, wobei X sich im Verlauf der Animation ändert. Um dann die Abbildung zu animieren gibt es den Befehl animate, der ein paar, für uns relevante, Argumente entgegennimmt:
plot: Welche Plot soll animiert werden? Hier fluidnframes: Wieviele Frames sollen insgesamt animiert werden?fps: Wieviele Frames per Second soll die finale Animation haben?duration: Wie lang soll die Animation (in Sekunden) sein?
Von den letzten drei sind immer nur zwei nötig, weil die dritte sich dann aus den beiden Angaben ergibt. In unserem Fall mach es Sinn jedem Tag einen eigenen Frame zuzuweisen und 10 Frames pro Sekunde zu benutzen:
animate(fluid,
nframes = max(covid_sel$Day),
fps = 10)
Die Animation sollte erneute ca. 20 Sekunden dauern und in folgendem gif resultieren (ich habe die Animation am 19. April durchgeführt, ihre Animation wird sich also dementsprechend unterscheiden):
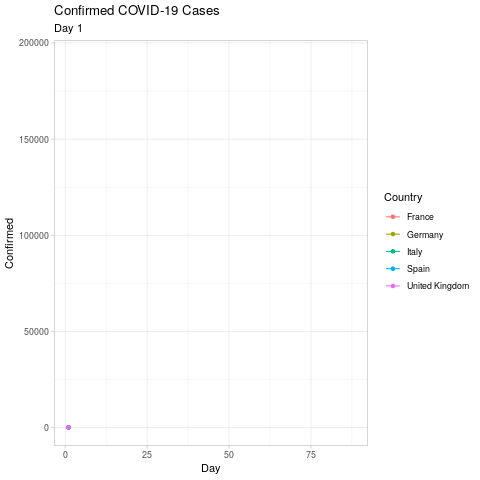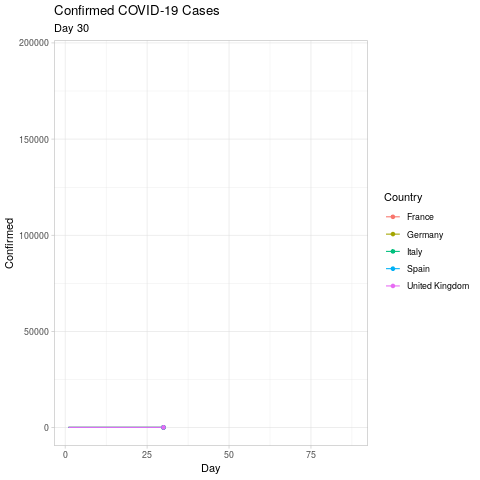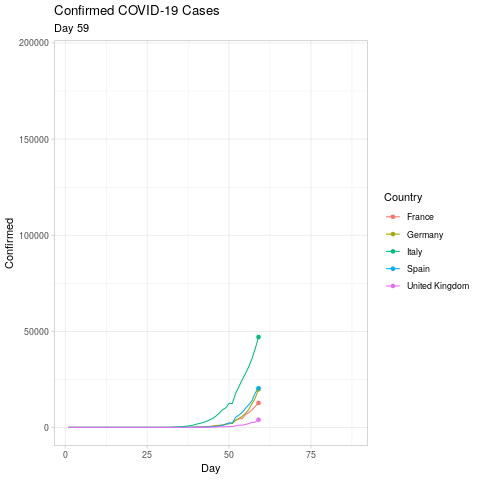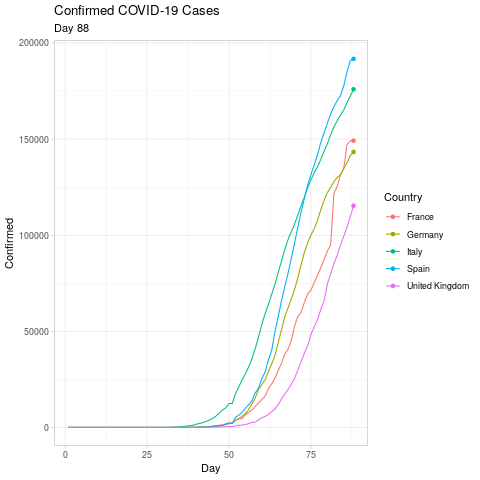
Karten
Bei psychologischen Daten eher selten, aber mit dem COVID-19 Datensatz natürlich sehr naheliegend, ist die Datenvisualisierung auf Karten. Für komplexere Karten (z.B. mit Google Maps) gibt es das ggmap Paket. Für unsere Zwecke reichen allerdings die von ggplot2 mitgelieferten Karten aus.
Karten benötigen eine sehr eigene Art der Datenaufbereitung, die häufig nicht gerade platzsparend ist. Daher sind die meisten Karten in R nicht als Datensätze vorhanden, sondern müssen erst einmal in solche überführt werden. Dafür gibt es die map_data Funktion. Um die Weltkarte in einen Datensatz zu übertragen, z.B.:
welt <- map_data('world')
head(welt)
Was Sie in den Daten sehen sind Länge- und Breitengrade von Landesgrenzen. Außerdem bestimmt die Variable group das Land (anhand dessen die Landesgrenzen gruppiert werden sollten). Damit Linie der Grenzen nicht hin und her springt gibt es außerdem die Variable order die angibt, welcher Punkt in der Grenze als nächstes kommt. Anhand dieser Punkte werden in ggplot2 mit der allgemeinen geom_polygon Funktion Karten gezeichnet. Um eine leere Weltkarte zu erzeugen reicht Folgendes aus:
ggplot(welt, aes(x = long, y = lat, group = group)) +
geom_polygon()
Wie Sie sehen, hat dieser Plot die gleichen Eigenschaften wie normale ggplots - weil es ein ganz normaler Plot ist. Um einzelne Länder erkennen zu können, sollten wir z.B. die Länder weiß und nicht schwarz füllen. Außerdem brauchen wir nicht unbedingt x- und y-Achse, sodass wir das komplett leere Theme theme_void nutzen können:
ggplot(welt, aes(x = long, y = lat, group = group)) +
geom_polygon(fill = 'white', color = 'black', lwd = .25) +
theme_void()
Um die Karten-Daten mit den COVID Daten in Verbindung zu bringen steht uns leider - wie so häufig - im Weg, dass die Daten nicht einheitlich kodiert wurden. In diesem Fall sind es die Benennungen der Länder, die uneinheitlich sind. Um herauszufinden, wo Unterschiede bestehen, können wir die normalen Operatoren der Mengenvergleiche in R nutzen:
setdiff(unique(welt$region), unique(covid$Country.Region))
setdiff(unique(covid$Country.Region), unique(welt$region))
Im Folgenden werden die Namen der Länder mit dem recode Befehl des car-Pakets umkodiert. Diesen hatten wir im letzten Semester zum Rekodieren negativ formulierter Items genutzt.
# Recodes
covid$Country.Region <- car::recode(covid$Country.Region,
"'Burma' = 'Myanmar';
'Cabo Verde' = 'Cape Verde';
'Congo (Brazzaville)' = 'Republic of Congo';
'Congo (Kinshasa)' = 'Democratic Republic of the Congo';
'Czechia' = 'Czech Republic';
'Eswatini' = 'Swaziland';
'Holy See' = 'Vatican';
'Korea, South' = 'South Korea';
'North Macedonia' = 'Macedonia';
'Saint Kitts and Nevis' = 'Saint Kitts';
'Saint Vincent and the Grenadines' = 'Saint Vincent';
'Taiwan*' = 'Taiwan';
'United Kingdom' = 'UK';
'US' = 'USA';
'West Bank and Gaza' = 'Palestine'")
Darüber hinaus brauchen zwei Inselstaaten eine gesonderte Behandlung, weil sie auf der Karte als separate Inseln, im COVID Datensatz aber als ein Land geführt werden:
# Antigua & Barbuda
covid$Country.Region <- car::recode(covid$Country.Region, "'Antigua and Barbuda'='Antigua'")
tmp <- covid[covid$Country.Region == 'Antigua', ]
tmp$Country.Region <- 'Barbuda'
covid <- rbind(covid, tmp)
# Trinidad & Tobago
covid$Country.Region <- car::recode(covid$Country.Region, "'Trinidad and Tobago'='Trinidad'")
tmp <- covid[covid$Country.Region == 'Trinidad', ]
tmp$Country.Region <- 'Tobago'
covid <- rbind(covid, tmp)
Zu guter Letzt muss die Elfenbeinküste einzeln umkodiert werden, weil der Apostroph im französischen Namen ein Problem bereitet:
levels(covid$Country.Region) <- c(levels(covid$Country.Region), 'Ivory Coast')
covid$Country.Region[covid$Country.Region == "Cote d'Ivoire"] <- 'Ivory Coast'
Diese Umkodierung ist nicht auf andere Datensätze übertragbar - Sie müssen immer in den Daten, die Sie vorliegen haben nachgucken, welche Schritte zum Angleichen verschiedener Datensätze notwendig sind.
Um die Daten für heute anzuzeigen, können wir den Datensatz wieder erst einmal auf heute beschränken:
covid_today <- covid[covid$Day == max(covid$Day), ]
Anschließend können wir den Datensatz mit der Weltkarte zusammenführen. Dafür verwenden wir wieder den merge Befehl. Damit nach dem merge die Grenzen richtig gezeichnet werden, müssen wieder die Reihenfolge der Daten wiederherstellen. Dazu wird mit order nach Land (group) und dann nach Reihenfolge der Grenzpunkte (order) sortiert.
covid_map <- merge(welt, covid_today, by.x = 'region', by.y = 'Country.Region', all.x = TRUE, all.y = FALSE)
covid_map <- covid_map[order(covid_map$group, covid_map$order), ]
Mit den neuen Daten können wir unsere vorherige Karte jetzt so ergänzen, dass wir die Länder nach der Anzahl der Fälle einfärben:
ggplot(covid_map, aes(x = long, y = lat, group = group)) +
geom_polygon(color = 'black', lwd = .25, aes(fill = Confirmed)) +
theme_void()
Wegen der exponentiellen Art und Weise mit der die Anzahl der Fälle in betroffenen Ländern zunimmt, sind mit dieser Skalierung nur wenige Länder überhaupt unterscheidbar. Das liegt besonders an der exorbitanten Anzahl bestätigter Fälle in den USA. Um solche Situationen zu umgehen wird in der Datenvisualisierung häufig mit logarithmischen Skalen gearbeitet. Das Gleiche können wir hier auch mit dem Argument trans für Funktionen tun, die mit scale_ beginnen. Darüber hinaus gefällt mir die Farbgebung nicht, sodass ich mit scale_fill_gradient2 drei Farben aussuchen werden, die Untergrenze, Mittelpunkt und Obergrenze der bestätigten Fälle kodieren:
ggplot(covid_map, aes(x = long, y = lat, group = group)) +
geom_polygon(color = 'black', lwd = .25, aes(fill = Confirmed)) +
theme_void() +
scale_fill_gradient2(low = '#737c45', mid = '#e3ba0f', high = '#ad3b76',
trans = 'log2',
midpoint = log(median(covid_map$Confirmed, na.rm = TRUE), 2))
Per Voreinstellung ist der Mittelpunkt der Skala bei 0 - was in unserem Fall nicht sonderlich sinnvoll ist. Stattdessen wird hier über midpoint der aktuelle Median als Mittelpunkt der Skala definiert.
Im letzten Schritt werden die Farbe, die bei fehlenden Werten vergeben wird, etwas angepasst und die Skala wird von der etwas befremdlich wirkenden Beschriftung in 10, 1000 und 100000 überführt. Darüber hinaus wird die Legende nach unten Verschoben, um der Karte genug Platz in die Breite zu geben. Mit scipen wird außerdem vorübergehend die wissenschaftliche Notation der Zahlen ausgesetzt:
options(scipen = 3)
ggplot(covid_map, aes(x = long, y = lat, group = group)) +
geom_polygon(color = 'black', lwd = .25, aes(fill = Confirmed)) +
theme_void() +
scale_fill_gradient2(trans = 'log2', low = '#737c45', mid = '#e3ba0f', high = '#ad3b76', na.value = 'grey95', midpoint = log(median(covid_map$Confirmed, na.rm = TRUE), 2),
breaks = c(10, 1000, 100000), name = 'Confirmed\n(Log-Scale)') +
theme(legend.position = 'bottom')
options(scipen = 1)
martscht/PsyBSc7 documentation built on Sept. 1, 2020, 10:50 p.m.
source('setup.R')
Einleitung
Das Paket ggplot2 ist das umfangreichste und am weitesten verbreitete Paket zur Grafikerstellung in R. Seine Beliebtheit liegt vor allem an zwei Dingen: Es ist sehr eng mit der kommerziellen Seite von RStudio verwoben (Autor ist auch hier Hadley Wickham) und es folgt stringent einer "Grammatik der Grafikerstellung". Aus dem zweiten Punkt leitet sich auch sein Name ab: das "gg" steht für "Grammar of Graphics" und geht auf das gleichnamige Buch von Leland Wilkinson zurück, in dem auf 700 kurzen Seiten eine grammatikalische Grundstruktur für das Erstellen von Grafiken zur Datendarstellung hergeleitet und detailliert erklärt wird.
Weil ggplot2 so beliebt ist, gibt es online tausende von Quellen mit Tutorials, Beispielen und innovativen Ansätzen zur Datenvisualisierung. Vom Autor des Pakets selbst gibt es ein Überblickswerk über Data-Science als e-Book, in dem sich auch ein Kapitel mit ggplot2 befasst.
Beispieldaten {#Beispieldaten}
Welches Beispiel wäre zur Zeit naheliegender als die Ausbreitung von COVID-19? Auf GitHub stellen viele Nutzer und Organisationen tagesaktuelle Daten zu Infektionsraten, bestätigten Fällen und vielen anderen Aspekten zur Verfügung. Diese Daten können wir aus R mit einfachen Funktionen wie read.table oder read.csv abrufen.
Dafür nutzen wir die Daten, die von der Johns Hopkins Universität auf GitHub bereitgestellt werden. Für uns sind dabei die zwei Datensätze zu bestätigten Fällen und Todesopfern zentral.
Daten herunterladen
Die erste Möglichkeit die Daten zu beziehen, ist es sie als .csv Dateien herunterzuladen und lokal zu speichern. Dafür reichen diese beiden Links aus: r fontawesome::fa('download') Bestätigte Fälle und r fontawesome::fa('download') Todesfälle.
Statt den Umweg über externe Dateien zu gehen, können wir aber auch direkt aus R heraus die entsprechenden .csv öffnen und als Objekte anlegen. Das hat den Vorteil, dass die Datei jedes mal, wenn das R-Skript ausgeführt wird, auf den aktuellen Stand gebracht wird.
In R können wir dafür einfach die normalen Befehle zum Einlesen von Dateien nutzen. Diese sind auch dafür ausgelegt, dass der Dateipfad sich nicht auf lokale Daten, sondern auf URLs bezieht. Für die erste Datei:
confirmed <- read.csv('https://raw.githubusercontent.com/CSSEGISandData/COVID-19/master/csse_covid_19_data/csse_covid_19_time_series/time_series_covid19_confirmed_global.csv')
Im Environment-Tab sieht man in RStudio die Eigenschaften dieses Datensatzes:
ncol(confirmed) nrow(confirmed)
Das Gleiche sollten wir jetzt noch für die anderen beiden Dateien machen, bevor wir uns gleich mit dem Format der Daten befassen.
deaths <- read.csv('https://raw.githubusercontent.com/CSSEGISandData/COVID-19/master/csse_covid_19_data/csse_covid_19_time_series/time_series_covid19_deaths_global.csv')
Daten aufbereiten (reshape)
Die Datensätze sind als Zeitreihen aufbereitet. Das bedeutet, dass jede Zeile des Datensatzes eine Region oder ein Land darstellt und jede Spalte des Datensatzes ein Tag ist. Was wir für unsere Visualisierung in ggplot2 benötigen ist ein Datensatz, der alle drei Kennzahlen enthält und dabei für jede Kombination aus Tag und Land eine Zeile enthält. Prinzipiell sollte der resultierende Datensatz also folgende Struktur haben:
| Provinz | Land | Tag | Bestätigt | Verstorben | Genesen | |:-------:|:----:|:---:|:---------:|:----------:|:-------:| | Ohio | USA | 21.3. | 248 | 3 | 0 | | Ohio | USA | 22.3. | 355 | 3 | 0 | | ... | ... | ... | ... | ... | ... | | | Germany | 21.3. | 22213 | 84 | 233 | | | Germany | 22.3. | 24873 | 94 | 266 | | ... | ... | ... | ... | ... | ... |
Für genau solche Umstellungen gibt es in R die Funktion reshape. Wagen Sie ruhig einen Blick in die Hilfestellung zu der Funktion mit ?reshape. Die Bezeichnungen sind zunächst nicht besonders eingängig, also nutzen wir den Abschnitt mit Beispielen um uns zu orientieren.
Bei der Formatierung von sozialwissenschaftlichen Datensätzen können zwei generelle Typen unterschieden werden: das Long-Format und das Wide-Format. Die Bezeichnung bezieht sich dabei (meistens) auf die Anordnung von Messwiederholungen der gleichen Personen - in unserem Fall sind es keine Personen, sondern Regionen.
Was wir haben ist also ein Datensatz im "breiten" Format (eine Spalte pro Tag) und wir möchten ihn in einen Datensatz im "langen" Format (eine Zeile pro Tag) übertragen. Um die vorliegenden Daten ins lange Format zu überführen, benötigt die Funktion mindestens fünf Argumente:
data: Der Datensatzvarying: Die Variablen die wiederholt gemessen wurdenv.names: Der Name unter dem die Variablen zusammengefasst werden sollentimevar: Die Variable, die Wiederholungen kennzeichnetidvar: Die Variablen, die sich über Wiederholungen nicht änderndirection: Das Zielformat des neuen Datensatzes
Für den ersten Datensatz sollte die Syntax also wie folgt aussehen:
confirmed_long <- reshape(confirmed, varying = names(confirmed)[-c(1:4)], v.names = 'Confirmed', timevar = 'Day', idvar = names(confirmed)[1:4], direction = 'long')
Wir nutzen den Datensatz confirmed. Wiederholt gemessen wurden alle Variablen außer die ersten 4 (Provinz, Land, Länge- und Breitengrad). Die neue Variable nennen wir Confirmed, damit wir gleich Verwirrung umgehen, wenn wir die drei Datensätze zusammenführen. Unsere Zeitvariable ist der Tag, also benennen wir sie mit Day. Als Identifikatoren halten unsere ersten 4 Variablen her (weil diese den Ort eindeutig bestimmen und sich nicht über die Zeit ändern) und die Zielausrichtung des Datensatzes ist "lang". Der Datensatz sollte also jetzt wie folgt aussehen:
head(confirmed_long)
Die Zeilennamen bewirken, dass der Datensatz etwas unübersichlich wirkt. Wir können diese ganz einfach entfernen:
rownames(confirmed_long) <- NULL head(confirmed_long)
Bevor jetzt die beiden Datensätze zusammenführen können, muss auch deaths ins Long-Format übertragen werden. Sie können unten probieren, ob das Vorgehen zum gewünschten Ergebnis führt. Sie können auch Änderungen vornehmen, um zu sehen, wie sich das Ergebnis verändert.
deaths_long <- reshape(deaths, varying = names(deaths)[-c(1:4)], v.names = 'Deaths', timevar = 'Day', idvar = names(deaths)[1:4], direction = 'long') rownames(deaths_long) <- NULL head(deaths_long)
Daten zusammenführen (merge)
Die beiden Datensätze, die wir jetzt erzeugt haben bestehen jeweils aus vier Variablen zur Region (Province.State, Country.Region, Lat und Long) einer Variable zur Zeit (Day) und der Anzahl der entsprechenden Fälle (Confirmed bzw. Deaths). Um die letzten beiden in einem Datensatz zusammenzuführen können wir die ersten fünf nutzen um eindeutig festzulegen, welche Daten zusammengehören. Daten vom gleichen Ort zur gleichen Zeit beziehen sich üblicherweise auf das Gleiche. Für das Zusammenführen von zwei Datensätzen gibt es in R den merge Befehl, der drei zentrale Argumente entgegennimmt:
x: Der erste Datensatzy: Der zweite Datensatzby: Die Variablen, anhand derer gleiche Fälle identifiziert werden können
In der Hilfe zur Funktion finden sich auch noch viele zusätzliche Argumente, aber diese Drei sind für uns zentral.
long <- merge(confirmed_long, deaths_long, by = c('Province.State', 'Country.Region', 'Lat', 'Long', 'Day')) head(long)
Daten zusammenfassen (aggregate)
Leider ist die Auflösung mit der die unterschiedlichen Länder erhoben werden nicht einheitlich. Für Australien liegen beispielsweise Daten aus 9 Bundesstaaten vor, für die USA sogar aus 247 Provinzen. Um die Daten einheitlich auf der Ebene von Nationen betrachten zu können, müssen sie aggregiert werden. In unserem Fall möchten wir also die Summe über z.B. die 9 Australischen Bundesstaaten bilden. Dafür gibt es in R sehr viele verschiedene Lösungsansätze. Einer davon ist der aggregate Befehl, den wir hier nutzen werden.
Um nun aggregieren zu können, können wir aggregate eine Formel geben, wie wir sie schon für Regressionen im letzten Semester verwendet haben. Die generelle Struktur solcher Formeln ist immer AV ~ UV, wobei mehrere UVs durch + vebunden werden können. In unserem Fall behandeln wir die zwei Spalten Confirmed und Deaths als AVs und Contry.Region plus Day als UVs. Zusätzlich benötigt der Befehl dann eine Auskunft darüber, auf welchen Datensatz wir uns beziehen und mit welcher Funktion wir aggregieren wollen:
covid <- aggregate(cbind(Confirmed, Deaths) ~ Country.Region + Day, data = long, FUN = 'sum') head(covid)
Der Datensatz enthält jetzt bestätigte Fälle, Todesfälle und genesene Fälle pro Land und für jeden Tag und ist damit bereit für die Visualisierung. Sie können das Feld unten nutzen, um sich noch einen besseren Überblick über die Datenlage zu verschaffen. Der Beispielcode zeigt Ihnen die Daten der letzten 10 Tage für Deutschland:
covid[covid$Country.Region == 'Germany' & covid$Day > max(covid$Day)-10, ]
ggplot2 Grundprinzipien {#Grundprinzipien}
In ggplot2 werden immer Daten aus einem data.frame dargestellt. Das heißt, dass wir nicht, wie bei plot oder hist aus R selbst, Vektoren oder Matrizen nutzen können. Daten müssen immer so aufbereitet sein, dass der grundlegende Datensatz sinnvoll benannte Variablen enthält und in dem Format vorliegt, in dem wir die Daten visualisieren wollen. Das hat zwar den Nachteil, dass wir Datensätze umbauen müssen, wenn wir Dinge anders darstellen wollen, aber hat auch den Vorteil, dass wir alle Kenntnisse über Datenmanagement im Allgemeinen auf den Umgang mit ggplot2 übertragen können. Deswegen haben wir auch im vergangenen Abschnitt so viel Zeit damit verbracht die Daten in das korrekte Format zu überführen.
Bevor wir loslegen können, muss natürlich ggplot2 installiert sein und geladen werden:
library(ggplot2)
Im Kern bestehen Abbildungen in der Grammatik von ggplot2 immer aus drei Komponenten:
- Daten, die angezeigt werden sollen
- Geometrie, die vorgibt welche Arten von Grafiken (Säulendiagramme, Punktediagramme, usw.) genutzt werden
- Ästhetik, die vorgibt, wie die Geometrie und Daten aufbereitet werden (z.B. Farben)
In den folgenden Abschnitten werden wir versuchen diese drei Komponenten so zu nutzen, dass wir informative und eventuell auch ansehnliche Abbildungen generieren.
Schichten
In ggplot2 werden Grafiken nicht auf einmal mit einem Befehl erstellt, sondern bestehen aus verschiedenen Schichten. Diese Schichten werden meistens mit unterschiedlichen Befehlen erzeugt und dann so übereinandergelegt, dass sich am Ende eine Abbildung ergibt.
Die Grundschicht sind die Daten. Dafür haben wir im vorherigen Abschnitt covid als Datensatz aufbereitet. Benutzen wir zunächst nur die Daten für Deutschland um nicht unnötig die Perspektive zu verzerren:
covid_de <- covid[covid$Country.Region == 'Germany', ]
ggplot(covid_de)
Was entsteht ist eine leere Fläche. Wie bereits beschrieben, besteht eine Abbildung in ggplot2 immer aus den drei Komponenten Daten, Geometrie und Ästhetik. Bisher haben wir nur eine festgelegt. Als erste Ästhetik sollten wir festlegen, welche Variablen auf x- und y-Achse dargestellt werden sollen. Nehmen wir naheliegenderweise die Zeit (x-Achse) und die Zahl der bestätigten Fälle:
ggplot(covid_de, aes(x = Day, y = Confirmed))
Ästhetik wird in ggplot2 über den aes-Befehl erzeugt. Jetzt fehlt uns noch die geometrische Form, mit der die Daten abgebildet werden sollen. Für die Geometrie-Komponente stehen in ggplot2 sehr viele Funktionen zur Verfügung, die allesamt mit geom_ beginnen. Eine Übersicht über die Möglichkeiten finden Sie z.B. hier. Naheliegende Möglichkeiten für den Zeitverlauf sind eine Linie (geom_line) und mehrere Punkte (geom_point). Neue Schichten werden in ihrer eigenen Funktion erzeugt und mit dem einfachen + zu einem bestehenden Plot hinzugefügt.
Im Folgenden ist eine Linie die genutzte Geometrie - probieren Sie aus, wie das Punktediagramm aussieht oder eine andere Geometrie funktioniert, die Ihnen gefällt!
ggplot(covid_de, aes(x = Day, y = Confirmed)) + geom_line()
Der immense Vorteil des Schichtens besteht darin, dass wir gleichzeitig mehrere Visualisierungsformen nutzen können. Das Prinizp bleibt das gleiche wie vorher: wir fügen Schichten mit dem + hinzu. Probieren Sie die Kombination aus Punkten und Linie im Beispiel oben aus!
Plots als Objekte
Einer der Vorteile, die sich durch das Schichten der Abbildungen ergibt ist, dass wir Teile der Abbildung als Objekte definieren können und sie in verschiedenen Varianten wieder benutzen können. Das hilft besonders dann, wenn wir unterschiedliche Geometrie in einer gemeinsamen Abbildung darstellen wollen oder z.B. erst einmal eine Abbildung definieren wollen, bevor wir Feinheiten adjustieren.
basic <- ggplot(covid_de, aes(x = Day, y = Confirmed))
In basic wird jetzt die Anleitung für die Erstellung der Grafik gespeichert. Erstellt wird die Grafik aber erst, wenn wir das Objekt aufrufen. Dabei können wir das Objekt auch mit beliebigen anderen Komponenten über + kombinieren. Probieren Sie es unten aus, mit Objekt-Kombinationen zu arbeiten!
basic + geom_point()
Damit die Beispiele im weiteren Verlauf auch selbstständig funktionieren, wird unten immer der gesamte Plot aufgeschrieben. Aber für Ihre eigenen Übungen oder Notizen ist es durchaus praktischer mit dieser Objekt Funktionalität zu arbeiten, um so zu umgehen, dass man immer wieder die gleichen Abschnitte aufschreiben muss.
Farben und Ästhetik {#Farben}
Oben wurde erwähnt, dass Ästhetik die dritte Komponente ist und als Beispiel Farbe genannt. Das stimmt nicht immer: die Farbe der Darstellung muss nicht zwingend eine Ästhetik sein. Gucken wir uns zunächst an, wie es aussieht, wenn wir die Farbe der Darstellung ändern wollen:
ggplot(covid_de, aes(x = Day, y = Confirmed)) + geom_point(color = 'blue')
Alle Punkte haben die Farbe geändert. Eine Ästhetik im Sinne der ggplot-Grammatik ist immer abhängig von den Daten. Die globale Vergabe von Farbe ist also keine Ästhetik. Sie ist es nur, wenn wir sie von Ausprägungen der Daten abhängig machen. Das funktioniert z.B. so:
ggplot(covid_de, aes(x = Day, y = Confirmed)) + geom_point(aes(color = Confirmed))
Über den Befehl aes definieren wir eine Ästhetik und sagen ggplot, dass die Farbe der Punkte von der Ausprägung auf der Variable Confirmed abhängen soll. Die Farbe kann aber natürlich auch von jeder anderen Variable im Datensatz abhängen. Wie das aussehen kann gucken wir uns im kommenden Abschnitt an.
Gruppierte Abbildungen
Im letzten Abschnitt hatten wir die Daten auf Fälle aus Deutschland reduziert. In diesem Abschnitt wollen wir gleichzeitig mehrere Länder betrachten können. Dafür müssen wir wieder zunächst die Daten auswählen, die relevant sind. Wir beschränken uns in diesem Fall auf Deutschland, Italien, Frankreich, das Vereinigte Königreich und Spanien.
covid_sel <- covid[covid$Country.Region %in% c('France', 'Germany', 'Italy', 'Spain', 'United Kingdom'), ]
Wenn wir jetzt mit dem gleichen Vorgehen wie oben die Abbildung erstellen, wird es etwas chaotischer, weil unklar ist, welche Punkte sich auf welches Land beziehen:
ggplot(covid_sel, aes(x = Day, y = Confirmed)) + geom_point()
Um das zu umgehen, können wir natürlich die Ästhetik der Farben benutzen:
ggplot(covid_sel, aes(x = Day, y = Confirmed)) + geom_point(aes(color = Country.Region))
Wie Sie sehen ergibt sich automatisch eine Legende auf der rechten Seite, die jedem Land eine Farbe zuweist. Wir können auch hier wieder eine Kombination aus Punkten und Linien nutzen:
ggplot(covid_sel, aes(x = Day, y = Confirmed)) + geom_point(aes(color = Country.Region)) + geom_line(aes(color = Country.Region))
Das Problem ist hier, dass wir die Ästhetik für jede Geomtrie wiederholen müssen. Stattdessen können wir in ggplot auch allgemein eine Gruppierung vornehmen, die für alle Geometrien übernommen wird:
ggplot(covid_sel, aes(x = Day, y = Confirmed, color = Country.Region)) + geom_point() + geom_line()
Wenn Sie das Ganze auch mit anderen Ländern ausprobieren möchten, können Sie das folgende Feld nutzen um erst Daten auszuwählen und dann einen Plot zu erstellen.
Mit "Hint" können Sie sich ein paar Hinweise ausgeben lassen.
# Ländernamen ausgeben lassen levels(covid$Country.Region)
# 10 Länder mit den meisten Fällen auswählen today <- covid[covid$Day == max(covid$Day), ] filt <- today[rank(1/today$Confirmed) <= 10, 'Country.Region'] covid_10 <- covid[covid$Country.Region %in% filt, ]
Faceting
Alle Länder in der gleichen Abbildung darzustellen kann mitunter sehr unübersichtlich werden. Eine Möglichkeit, Übersichtlichkeit zu bewahren ist das sogenannte Faceting. Dabei wird eine Abbildung anhand von Ausprägungen auf einer oder mehr Variablen in verschiedene Abbildungen unterteilt. Mit dem gleichen Datensatz aus Deutschland, Italien, Frankreich, dem Vereinigten Königreich und Spanien können wir die Abbildung anhand des Landes einteilen:
ggplot(covid_sel, aes(x = Day, y = Confirmed)) + geom_point() + geom_line() + facet_wrap(~ Country.Region)
In facet_wrap wird wieder mit der R Gleichungsnotation gearbeitet: hier wird der Plot anhand der unabhängigen Variablen hinter der Tilde in Gruppen eingeteilt. Das gibt auch wieder die Möglichkeit mit + mehrere Variablen zu definieren, die zum Faceting benutzt werden sollen. Wenn Sie Gruppen anhand von zwei Variablen bilden bietet es sich außerdem an facet_grid zu benutzen.
Per Voreinstellung wird beim Faceting eine gemeinsame Skalierung der x- und y-Achsen für alle Teilabbildungen festgelegt. Das kann mit dem Argument scales in der facet_wrap Funktion umgangen werden. Bei ?facet_wrap finden Sie dafür genauere Informationen.
Mehrere Variablen
Bisher haben wir auf der x-Achse nur die Zeit und auf der y-Achse nur die bestätigten Fälle betrachtet. Durch das Schicht-System können wir auf unsere bisherigen Abbildungen aber auch zusätzliche Variablen - oder sogar komplett andere Datensätze - abbilden. Wenn wir uns z.B. die bestätigten und die Todesfälle in Deutschland angucken wollen, können wir wie folgt vorgehen:
ggplot(covid_de, aes(x = Day)) + geom_line(aes(y = Confirmed), color = 'darkblue') + geom_line(aes(y = Deaths), color = 'darkred')
Abbildungen anpassen {#Anpassen}
Die Abbildungen, die wir bisher erstellt haben nutzen alle das in ggplot2 voreingestellt Design. Auch wenn es sicherlich einen theoretisch sehr gut fundierten Grund gibt, dass der Hintergrund der Abbildung in einem demotivierenden Grauton gehalten sein sollte, gibt es Designs, die man schöner finden kann. Im folgenden gucken wir uns an, wie man seine Abbildungen nach seinen eigenen Vorlieben anpassen kann.
Themes {#Themes}
In ggplot2 werden die Grundeigenschaften von Abbildungen in "Themes" zusammengefasst. Mit ?theme_test erhalten Sie eine Auflistung aller Themes, die von ggplot2 direkt zur Verfügung gestellt werden. Noch mehr Themes gibt es im Paket ggthemes, welches Sie zusätzlich installieren können.
Um das Theme einer Abbildung zu verändern, können Sie es - wie Geometrie - mit dem + hinzufügen. Probieren Sie unten ein paar Themes aus und finden Sie Ihren Liebling!
ggplot(covid_sel, aes(x = Day, y = Confirmed, color = Country.Region)) + geom_line() + geom_point() + theme_light()
Sie können natürlich nicht nur vordefinierte Themes nutzen, sondern über theme jeden Aspekt der Aufbereitung der Abbildung selbst steuern. Mit ?theme sehen Sie die ausufernde Breite an Möglichkeiten, die Ihnen hier gegeben wird.
Beschriftung
Eine der wichtigsten Komponenten jeder Abbildung ist die Beschriftung. Nur wenn ausreichend gut gekennzeichnet ist, was wir darstellen, können wir darauf hoffen, dass die Information vermittelt wird, die wir vermitteln wollen. Die folgenden Befehle von ggplot2 beziehen sich spezifisch auf Beschriftungen, Sie können diese Änderungen aber auch allesamt mit der oben erwähnten theme-Funktion erreichen.
Zunächst ist es sinnvoll die Achsen ordentlich zu beschriften. Per Voreinstellung werden hierzu die Namen der Variablen genutzt. Wir können also eine nützliche Beschriftung schon früh dadurch erzwingen, dass wir die Variablen im Datensatz ordentlich benennen. Besonders wenn die Achsen aber Zusatzinformationen (wie z.B. "(in %)") enthalten sollen, ist es aber unumgänglich die Benennung hinterher zu ergänzen. Darüber hinaus kann es sinnvoll sein, einer Grafik Titel und Untertitel zu geben.
ggplot(covid_sel, aes(x = Day, y = Confirmed, color = Country.Region)) + geom_line() + geom_point() + theme_light() + labs(x = 'Tage seit dem 22.1.', y = 'Bestätigte Fälle') + ggtitle('COVID-19 Infektionen', paste('Stand:', Sys.Date()))
Im Untertitel ist dargestellt, dass wir auch für Beschriftungen wieder Objekte und Werte aus Objekten nutzen können, die in R vorliegen. Hier wird über Sys.Date() das aktuelle Datum abgefragt und durch paste mit "Stand:" zusammengeklebt.
Der Befehl labs bezieht sich auf die Beschriftung von jeder Ästhetik in der Abbildung. In der aktuellen Variante ist die Legende der Länder mit Country.Region nicht sonderlich schön benannt. Versuchen Sie unten diese Beschriftung über den labs Befehl in etwas Sinnvolleres zu ändern!
ggplot(covid_sel, aes(x = Day, y = Confirmed, color = Country.Region)) + geom_line() + geom_point() + theme_light() + labs(x = 'Tage seit dem 22.1.', y = 'Bestätigte Fälle') + ggtitle('COVID-19 Infektionen', paste('Stand:', Sys.Date()))
Farbpaletten
In ggplot2 wird die Vergabe von Farben in der Ästhetik anhand von zwei Dingen unterschieden: der Geometrie und dem Skalenniveau der Variable, die die Färbung vorgibt.
In den bisherigen Beispielen im Abschnitt Farben und Ästhetik haben wir die Unterschiede bezüglich des Skalenniveaus schon gesehen: Kontinuierliche Variablen (Variablen, die in R als numeric definiert sind) werden anhand eines Blau-Farbverlaufs dargestellt, diskrete Variablen (Variablen, die in R als factor definiert sind) anhand eines vordefinierten Schemas unterschiedlicher Farben. Dieses Schema ist das Brewer Farbschema, welches usprünglich für Kartendarstellungen entwickelt wurde.
Bezüglich Geometrie wird bei der Färbung zwischen fill und color unterschieden - also ob eine Geometrie mit einer Farbe gefüllt wird oder ihr Rand mit dieser Farbe gezeichnet wird. In den bisherigen Abbildungen haben wir noch kein Beispiel gehabt, in dem etwas gefüllt werden könnte, aber in den Aufgaben zu dieser Sitzung könnte das noch relevant werden.
Nehmen wir an, dass wir unsere Abbildung irgendwo drucken möchten - Farbdruck ist wahnsinnig teuer. Um mit Grautönen zu arbeiten, können wir z.B. scale_color_grey benutzen:
ggplot(covid_sel, aes(x = Day, y = Confirmed, color = Country.Region)) + geom_line() + geom_point() + theme_light() + scale_color_grey()
Das bei den Themes erwähnte Paket ggthemes enthält auch weitere Farbpaletten, die Sie nutzen können, um Ihren Plot nach Ihren Vorlieben zu gestalten. Wichtig ist beispielsweise, dass es eine Palette namens colorblind hat, die Farben so auswählt, dass sie auch von Personen mit Farbblindheit differenziert werden können. In Fällen mit 6 oder weniger Gruppen bietet sich darüber hinaus in solchen Fällen an mit der Ästhetik pch (für plotcharacter). Darüber hinaus gibt es für Fans der Filme von Wes Anderson z.B. das Paket wesanderson, welches für jeden seiner Filme die Farbpalette parat hat. Darüber hinaus können wir aber natürlich auch unsere ganz eigene Farbpalette definieren - z.B. die offizielle Farbpalette des Corporate Designs der Goethe Universität, die Sie auf den Folien von PsyBSc 1 und 2 im letzten Semester kennen (und lieben!) gelernt haben.
Für diese Palette können wir zunächst in einem Objekt die Farben festhalten, die wir benötigen. In ggplot2 ist es dabei am gängigsten, Farben entweder über Worte auszuwählen oder via hexadezimaler Farbdefinition zu bestimmen. Für die fünf Farben, die von der Corporate Design Abteilung der Goethe Uni definiert werden ergibt sich folgendes Objekt:
gu_colors <- c('#00618f', '#e3ba0f', '#ad3b76', '#737c45', '#c96215')
Dieses Objekt können wir dann nutzen um mit scale_color_manual selbstständig Farben zuzuweisen:
ggplot(covid_sel, aes(x = Day, y = Confirmed, color = Country.Region)) + geom_line() + geom_point() + theme_light() + scale_color_manual(values = gu_colors)
Die Zuordnung der Farben erfolgt anhand der Reihenfolge in gu_colors und der Reihenfolge der Ausprägungen von Country.Region. Letztere ist - wie sie bestimmt festgestellt haben - alphabetisch. Wie häufig in ggplot2 können Sie die Daten ändern (also mit relevel die Reihenfolge der Ausprägungen ändern) um Veränderungen in der Darstellung zu bewirken.
Verschiedene Plots
Bisher haben wir ausschließlich Punkte- und Liniendiagramme benutzt, um die Daten darzustellen. Im letzten Semester haben Sie darüber hinaus noch einige Arten von Diagrammen kennengelernt, die in der psychologischen Forschung extrem verbreitet sind. Ein paar davon werden auch in diesem Abschnitt erstellt.
Balkendiagramme
Häufigkeiten von nominalskalierten Variablen werden typischerweise in Balkendiagrammen dargestellt. Dazu benutzen wir in diesem Beispiel die Anzahl bestätigter Fälle zum jetzigen Zeitpunkt. Zunächst müssen wir also wieder den Datensatz so umstellen, dass er nur die heutigen Fallzahlen enthält. Weil in der bisherigen Variante von covid_sel die Tage fortlaufend nummeriert sind, können wir die heutigen Zahlen einfach anhand des Maximalwertes des Tages extrahieren:
covid_today <- covid_sel[covid_sel$Day == max(covid_sel$Day), ] covid_today
Um diese Daten abzubilden könnten wir jetzt theoretisch wie bisher vorgehen, lediglich die Variable auf x-Achse und die genutzte Geometrie ändern sich:
ggplot(covid_today, aes(x = Country.Region, y = Confirmed)) + geom_bar()
Der Fehler verrät uns, dass ggplot2 davon ausgeht, dass wir die Häufigkeit einer nominalskalierten Variablen (stat_count) auszählen lassen wollen. Das wäre dann z.B. der Fall, wenn wir einen Datensatz hätten, der Personen enthält und auf einer Variable das Geschlecht kodiert wird. In dem Fall würde ggplot2 die Häufigkeit der verschiedenen Ausprägungen auszählen und uns diese Plotten.
In unserem Fall haben wir allerdings eine Variable, die die Häufigkeiten enthält, sodass wir dieses Verhalten unterdrücken müssen. Das erreichen wir dadurch, dass wir als "zu berechnende Statistik" statt dem Auszählen (stat = 'count') die schon vorliegende Statistik (stat = 'identity') benutzen.
ggplot(covid_today, aes(x = Country.Region, y = Confirmed)) + geom_bar(stat = 'identity')
Diese Art der Abbildung kann natürlich auch wieder den Möglichkeiten verbunden werden, die wir im Abschnitt Abbildungen Anpassen besprochen hatten. Probieren Sie es doch direkt aus! Sie können z.B. ein Theme mit einem weißen Hintergrund wählen und die Balken nach Land farbig füllen lassen.
ggplot(covid_today, aes(x = Country.Region, y = Confirmed)) + geom_bar(stat = 'identity')
Histogramme
Die Übertragung der Balkendiagramme auf mindestens intervallskalierte Variablen hatten wir letztes Semester in Form von Histogrammen behandelt. Dafür wird eine intervallskalierte Variable in verschiedene Intervalle eingeteilt und die Häufigkeit angezeigt, mit der Ausprägungen auf der Variable in ein Intervall fallen. Wir können uns dafür die Anzahl der bis heute bestätigten Fälle über alle Länder hinweg ansehen. Wie üblich, müssen wir dafür zunächst die geeigneten Daten auswählen:
covid_global <- covid[covid$Day == max(covid$Day), ]
Dann können wir die Daten mit geom_histogram ganz einfach darstellen. Der einzige Unterschied ist, dass jetzt die uns interessierende Variable auf der x- und nicht mehr auf der y-Achse abgebildet wird.
ggplot(covid_global, aes(x = Confirmed)) + geom_histogram()
ggplot2 macht uns darauf aufmerksam, dass wir keine Breite der Intervalle (binwidth) bzw. keine Anzahl der Intervalle (bins) angegeben haben und daher 30 Intervalle genutzt werden. Mit den Argumenten binwidth bzw. bins können wir hier händische Einstellungen vornehmen.
Boxplots
Um die Verteilung von Variablen zu untersuchen, werden in den Sozialwissenschaften häufig Boxplots genutzt. In ggplot2 werden diese entsprechend über geom_boxplot erstellt. Wir können also die gleichen Informationen wie im letzten Abschnitt auch in Form eines Boxplots darstellen:
ggplot(covid_global, aes(y = Confirmed)) + geom_boxplot()
Plots mit Trendlinien
In den Abschnitten ggplot2 Grundprinzipien und Abbildungen anpassen haben wir uns mit dem sehr verbreiteten Scatterplot befasst. In diesen Plots werden zwei Variablen zueinander ins Verhältnis gestellt - bei uns war das die Zeit auf der x-Achse und die Anzahl bestätigter Fälle auf der y-Achse. Typischerweise wollen wir aus solchen Plots auch einen Trend erkennen können. Im Fall von COVID-19 ist dieser Trend auch in den Rohdaten sehr leicht erkennbar, aber das ist nicht immer der Fall. Mit geom_smooth können wir uns eine "Glättung" der Datenlagen anschauen, die den generellen Trend verdeutlicht. Per Voreinstellung wird hierzu die sogenannte LOESS-Glättung genutzt.
ggplot(covid_sel, aes(x = Day, y = Confirmed)) + geom_point() + geom_smooth()
Was eingezeichnet wird ist die globale Trendlinie. Die schattierte Region um diese Linie herum stellt den Standardschätzfehler dieser Kurve dar. Um die Trends länderspezifisch einzuzeichnen, können wir erneut mit der Ästhetik color arbeiten, um die Länder in allen Geometrien farblich voneinander abzugrenzen:
ggplot(covid_sel, aes(x = Day, y = Confirmed, color = Country.Region)) + geom_point() + geom_smooth()
Aufgaben
Zur Bearbeitung der Aufgaben öffnen Sie bitte ein neues RStudio-Fenster. Es werden die Daten benötigt, wie Sie in Beispieldaten erzeugt wurden. Sie können entweder die Syntax von dort kopieren und sie Ihrem Skript hinzufügen, oder den Befehl PsyBSc7::Aufgaben_2() ausführen um die Daten direkt im Workspace anlegen zu lassen.
In der Berichterstattung zur aktuellen COVID-19 Ausbreitung stehen vor allem europäische Länder, Süd-Korea, China und die USA im Zentrum. Um mal eine andere Gegend der Erde in den Fokus zu Rücken, betrachten wir in den Aufgaben die Ausbreitung in Südostasien. Untenstehende Tabelle stellt ein paar Informationen über die 11 Südostasiatischen Länder zusammen (Quelle):
| Land | Bevölkerung (in 100 000) | Human Development Index | |:----:|:------------------------:|:-----------------------:| | Brunei | 4 | .845 | | Indonesien | 2677 | .707 | | Kambodscha | 167 | .581 | | Laos | 71 | .604 | | Malaysia | 315 | .804 | | Myanmar | 537 | .578 | | Osttimor | 12 | .626 | | Philippinen | 1067 | .712 | | Singapur | 58 | .935 | | Thailand | 694 | .765 | | Vietnam | 955 | .693 |
- Erstellen Sie einen Datensatz mit dem Namen
covid_se, der nur die Daten dieser Länder enthält (Myanmar wird im englischsprachigen Raum weiterhin offiziell als "Burma" geführt). - Die Darstellung absoluter Fallzahlen wird häufig als irreführend angesehen, weil Länder mit größerer Bevölkerung mehr mögliche Fälle habe. Nutzen Sie obenstehende Tabelle um die Anzahl bestätigter Fälle an der Bevölkerung zu relativieren und so die bestätigten Fälle pro 100 000 Einwohner zu erhalten. Nennen sie diese Variable
Per100k. - Stellen Sie die Bestätigten Fälle pro 100 000 Einwohner in Abhängigkeit von der Zeit in einer Abbildung dar. Nutzen Sie Farben um zwischen den Ländern zu unterscheiden.
- Stellen Sie für jedes Land in einer Teilabbildung die bestätigten Fälle (
Confirmed), und die Todesfälle (Deaths) dar. Nutzen sie für die beiden Linien unterschiedliche Farben (aber über alle Länder hinweg die gleichen). Weil sich die Bevölkerungszahlen so drastisch unterscheiden, lassen sie die y-Achse über die Länder hinweg frei variieren. - Nutzen Sie ein Balkendiagramm, um die relative Häufigkeit verstorbener Personen (
Deaths) and der Gesamtzahl besätigter Fälle (Confirmed) pro Land bis zum heutigen Tag darzustellen. Denken Sie daran, dass Sie dafür Daten aggregieren müssen. Füllen Sie die Balken dabei mit länderspezifischen Farben.
Bonusplots
Weil auch ich mich in dieser Phase der eingeschränkten Freizeitmöglichkeiten beschäftigen muss, habe ich für Sie im Folgenden ein paar zusätzliche Abbildungen erstellt, die auch Sie zum gleichen Datensatz mit ggplot2 und Erweiterungen erstellen können. Diese gehen über das Ausmaß dessen hinaus, was wir im Rahmen dieses Semesters besprechen, aber falls Sie die grafische Darstellung von Daten interessiert, sollten Sie aber von diesen Möglichkeiten gehört haben.
Animationen
Was sich bei Verlaufsdaten anbietet ist es, diese auch so zu animieren, dass der Verlauf deutlich wird. Für Animationen gibt es für ggplot2 das Erweiterungspaket gganimate, das Ihnen die Möglichkeit bietet, normale ggplots um eine Achse - die Zeit - zu erweitern. Zuerst aber müssen wir gganimate laden:
library(gganimate)
Nehmen wir wieder den Plot der Verläufe der bestätigten Fälle. Dieser stellt jetzt den "statischen" Anteil unserer Abbildung dar, daher können wir ihn einfach static nennen:
static <- ggplot(covid_sel, aes(x = Day, y = Confirmed, color = Country.Region)) + geom_line() + geom_point() + theme_light() + scale_color_discrete('Country')
Im Gegensatz zu den bisherigen Abbildungen ändern wir außerdem den Titel der Legende zu "Country", um das Ganze ein wenig aufzuhübschen. In static ist jetzt zunächst der gleiche Plot enthalten, wie bisher:
static
Um Animationen zu generieren, können wir mit den Funktionen von gganimate eine neue Schicht - die der Übergänge - erzeugen. Alle Funktionen, mit denen man durch das Pakte Animationen erzeugen kann beginnen mit transition_ - ähnlich wie im Kern-ggplot2 alle Geometrien mit geom_ beginnen.
Um in einer Animation nach und nach die Werte der einzelnen Tage anzuzeigen, können wir die Daten schrittweise "aufdecken" - der dazugehörige Befehl heißt also transition_reveal. Um das mit unserem bisher statistischen Plot zu verbinden, benutzen wir in ggplot Tradition weiterhin das +:
fluid <- static + transition_reveal(Day)
Die Funktion transition_reveal möchte als Argument wissen, anhand welche Variable des Datensatzes wir die Zeitachse kodieren. Bei uns ist das der Tag, also Day. Wenn Sie jetzt fluid aufrufen würden, würde nach ca. 20 Sekunden renderzeit eine Animation in RStudio erscheinen. Wir fügen der Abbildung aber zunächst noch einen Titel und Untertitel hinzu. Dabei soll der Untertitel den jeweiligen Tag angeben, sich also über die Zeit ebenfalls verändern. In der Hilfe zu transition_reveal finden Sie den Abschnitt Label variables, der Ihnen verrät, welche Werte von der Animation ausgegeben werden und z.B. in die Benennung mit eingebunden werden können. In diesem Fall heißt diese Variable frame_along. Diese können wir in den Untertitel einbinden:
fluid <- static + transition_reveal(Day) + ggtitle('Confirmed COVID-19 Cases', subtitle = 'Day {frame_along}')
Somit wird in der Animation im Untertitel immer "Day X" stehen, wobei X sich im Verlauf der Animation ändert. Um dann die Abbildung zu animieren gibt es den Befehl animate, der ein paar, für uns relevante, Argumente entgegennimmt:
plot: Welche Plot soll animiert werden? Hierfluidnframes: Wieviele Frames sollen insgesamt animiert werden?fps: Wieviele Frames per Second soll die finale Animation haben?duration: Wie lang soll die Animation (in Sekunden) sein?
Von den letzten drei sind immer nur zwei nötig, weil die dritte sich dann aus den beiden Angaben ergibt. In unserem Fall mach es Sinn jedem Tag einen eigenen Frame zuzuweisen und 10 Frames pro Sekunde zu benutzen:
animate(fluid, nframes = max(covid_sel$Day), fps = 10)
Die Animation sollte erneute ca. 20 Sekunden dauern und in folgendem gif resultieren (ich habe die Animation am 19. April durchgeführt, ihre Animation wird sich also dementsprechend unterscheiden):

Karten
Bei psychologischen Daten eher selten, aber mit dem COVID-19 Datensatz natürlich sehr naheliegend, ist die Datenvisualisierung auf Karten. Für komplexere Karten (z.B. mit Google Maps) gibt es das ggmap Paket. Für unsere Zwecke reichen allerdings die von ggplot2 mitgelieferten Karten aus.
Karten benötigen eine sehr eigene Art der Datenaufbereitung, die häufig nicht gerade platzsparend ist. Daher sind die meisten Karten in R nicht als Datensätze vorhanden, sondern müssen erst einmal in solche überführt werden. Dafür gibt es die map_data Funktion. Um die Weltkarte in einen Datensatz zu übertragen, z.B.:
welt <- map_data('world') head(welt)
Was Sie in den Daten sehen sind Länge- und Breitengrade von Landesgrenzen. Außerdem bestimmt die Variable group das Land (anhand dessen die Landesgrenzen gruppiert werden sollten). Damit Linie der Grenzen nicht hin und her springt gibt es außerdem die Variable order die angibt, welcher Punkt in der Grenze als nächstes kommt. Anhand dieser Punkte werden in ggplot2 mit der allgemeinen geom_polygon Funktion Karten gezeichnet. Um eine leere Weltkarte zu erzeugen reicht Folgendes aus:
ggplot(welt, aes(x = long, y = lat, group = group)) + geom_polygon()
Wie Sie sehen, hat dieser Plot die gleichen Eigenschaften wie normale ggplots - weil es ein ganz normaler Plot ist. Um einzelne Länder erkennen zu können, sollten wir z.B. die Länder weiß und nicht schwarz füllen. Außerdem brauchen wir nicht unbedingt x- und y-Achse, sodass wir das komplett leere Theme theme_void nutzen können:
ggplot(welt, aes(x = long, y = lat, group = group)) + geom_polygon(fill = 'white', color = 'black', lwd = .25) + theme_void()
Um die Karten-Daten mit den COVID Daten in Verbindung zu bringen steht uns leider - wie so häufig - im Weg, dass die Daten nicht einheitlich kodiert wurden. In diesem Fall sind es die Benennungen der Länder, die uneinheitlich sind. Um herauszufinden, wo Unterschiede bestehen, können wir die normalen Operatoren der Mengenvergleiche in R nutzen:
setdiff(unique(welt$region), unique(covid$Country.Region)) setdiff(unique(covid$Country.Region), unique(welt$region))
Im Folgenden werden die Namen der Länder mit dem recode Befehl des car-Pakets umkodiert. Diesen hatten wir im letzten Semester zum Rekodieren negativ formulierter Items genutzt.
# Recodes covid$Country.Region <- car::recode(covid$Country.Region, "'Burma' = 'Myanmar'; 'Cabo Verde' = 'Cape Verde'; 'Congo (Brazzaville)' = 'Republic of Congo'; 'Congo (Kinshasa)' = 'Democratic Republic of the Congo'; 'Czechia' = 'Czech Republic'; 'Eswatini' = 'Swaziland'; 'Holy See' = 'Vatican'; 'Korea, South' = 'South Korea'; 'North Macedonia' = 'Macedonia'; 'Saint Kitts and Nevis' = 'Saint Kitts'; 'Saint Vincent and the Grenadines' = 'Saint Vincent'; 'Taiwan*' = 'Taiwan'; 'United Kingdom' = 'UK'; 'US' = 'USA'; 'West Bank and Gaza' = 'Palestine'")
Darüber hinaus brauchen zwei Inselstaaten eine gesonderte Behandlung, weil sie auf der Karte als separate Inseln, im COVID Datensatz aber als ein Land geführt werden:
# Antigua & Barbuda covid$Country.Region <- car::recode(covid$Country.Region, "'Antigua and Barbuda'='Antigua'") tmp <- covid[covid$Country.Region == 'Antigua', ] tmp$Country.Region <- 'Barbuda' covid <- rbind(covid, tmp) # Trinidad & Tobago covid$Country.Region <- car::recode(covid$Country.Region, "'Trinidad and Tobago'='Trinidad'") tmp <- covid[covid$Country.Region == 'Trinidad', ] tmp$Country.Region <- 'Tobago' covid <- rbind(covid, tmp)
Zu guter Letzt muss die Elfenbeinküste einzeln umkodiert werden, weil der Apostroph im französischen Namen ein Problem bereitet:
levels(covid$Country.Region) <- c(levels(covid$Country.Region), 'Ivory Coast') covid$Country.Region[covid$Country.Region == "Cote d'Ivoire"] <- 'Ivory Coast'
Diese Umkodierung ist nicht auf andere Datensätze übertragbar - Sie müssen immer in den Daten, die Sie vorliegen haben nachgucken, welche Schritte zum Angleichen verschiedener Datensätze notwendig sind.
Um die Daten für heute anzuzeigen, können wir den Datensatz wieder erst einmal auf heute beschränken:
covid_today <- covid[covid$Day == max(covid$Day), ]
Anschließend können wir den Datensatz mit der Weltkarte zusammenführen. Dafür verwenden wir wieder den merge Befehl. Damit nach dem merge die Grenzen richtig gezeichnet werden, müssen wieder die Reihenfolge der Daten wiederherstellen. Dazu wird mit order nach Land (group) und dann nach Reihenfolge der Grenzpunkte (order) sortiert.
covid_map <- merge(welt, covid_today, by.x = 'region', by.y = 'Country.Region', all.x = TRUE, all.y = FALSE) covid_map <- covid_map[order(covid_map$group, covid_map$order), ]
Mit den neuen Daten können wir unsere vorherige Karte jetzt so ergänzen, dass wir die Länder nach der Anzahl der Fälle einfärben:
ggplot(covid_map, aes(x = long, y = lat, group = group)) + geom_polygon(color = 'black', lwd = .25, aes(fill = Confirmed)) + theme_void()
Wegen der exponentiellen Art und Weise mit der die Anzahl der Fälle in betroffenen Ländern zunimmt, sind mit dieser Skalierung nur wenige Länder überhaupt unterscheidbar. Das liegt besonders an der exorbitanten Anzahl bestätigter Fälle in den USA. Um solche Situationen zu umgehen wird in der Datenvisualisierung häufig mit logarithmischen Skalen gearbeitet. Das Gleiche können wir hier auch mit dem Argument trans für Funktionen tun, die mit scale_ beginnen. Darüber hinaus gefällt mir die Farbgebung nicht, sodass ich mit scale_fill_gradient2 drei Farben aussuchen werden, die Untergrenze, Mittelpunkt und Obergrenze der bestätigten Fälle kodieren:
ggplot(covid_map, aes(x = long, y = lat, group = group)) + geom_polygon(color = 'black', lwd = .25, aes(fill = Confirmed)) + theme_void() + scale_fill_gradient2(low = '#737c45', mid = '#e3ba0f', high = '#ad3b76', trans = 'log2', midpoint = log(median(covid_map$Confirmed, na.rm = TRUE), 2))
Per Voreinstellung ist der Mittelpunkt der Skala bei 0 - was in unserem Fall nicht sonderlich sinnvoll ist. Stattdessen wird hier über midpoint der aktuelle Median als Mittelpunkt der Skala definiert.
Im letzten Schritt werden die Farbe, die bei fehlenden Werten vergeben wird, etwas angepasst und die Skala wird von der etwas befremdlich wirkenden Beschriftung in 10, 1000 und 100000 überführt. Darüber hinaus wird die Legende nach unten Verschoben, um der Karte genug Platz in die Breite zu geben. Mit scipen wird außerdem vorübergehend die wissenschaftliche Notation der Zahlen ausgesetzt:
options(scipen = 3) ggplot(covid_map, aes(x = long, y = lat, group = group)) + geom_polygon(color = 'black', lwd = .25, aes(fill = Confirmed)) + theme_void() + scale_fill_gradient2(trans = 'log2', low = '#737c45', mid = '#e3ba0f', high = '#ad3b76', na.value = 'grey95', midpoint = log(median(covid_map$Confirmed, na.rm = TRUE), 2), breaks = c(10, 1000, 100000), name = 'Confirmed\n(Log-Scale)') + theme(legend.position = 'bottom') options(scipen = 1)
Add the following code to your website.
For more information on customizing the embed code, read Embedding Snippets.
