In DHLab-TSENG/dxpr: An analysis tool for diagnosis and procedure records
診斷前處理與整合方法
cat(utils::packageDescription("dxpr")$Description)
安裝套件
install.packages("remotes")
# Install development version from GitHub
remotes::install_github("DHLab-TSENG/dxpr")
library(dxpr)
knitr::opts_chunk$set(collapse = T, comment = "#>")
options(tibble.print_min = 4L, tibble.print_max = 4L)
#remotes::install_github("DHLab-TSENG/dxpr")
library(dxpr)
ICD-CM-CODE 格式對照
本套件使用的診斷編碼依照 CMS (Centers for Medicare & Medicaid Services)所提供的診斷編碼做為標準依據,並依照WHO訂定的診斷編碼規則,分別產生 ICD-9-CM and ICD-10-CM 的兩種格式的表格: ICD9DxwithTwoFormat 和 ICD10DxwithTwoFormat 供後續診斷編碼格式轉換的功能使用
ICD-9-CM
# Short
head(ICD9DxwithTwoFormat$Short)
# Decimal
head(ICD9DxwithTwoFormat$Decimal)
ICD-10-CM
# Short
head(ICD10DxwithTwoFormat$Short)
# Decimal
head(ICD10DxwithTwoFormat$Decimal)
範例資料
以下功能介紹皆以sampleDxFile做為範例,共有38位病患,300筆診斷紀錄
head(sampleDxFile)
I. Data Integration
一、格式轉換
ICD-CM code兩種格式的轉換:Short <-> Decimal
將醫療大數據中的診斷編碼進行編碼一致格式的轉換,以便後續診斷編碼的標準化分群。依據 Clinical Classifications Software (CCS)、共病症 (Comorbidity) 及 Phenome-wide association studies (PheWAS) 的分組表,其分別使用之 ICD 診斷碼格式如表一。
表一 標準化分組之診斷編碼格式
| |ICD format|
|--------|----|
|Clinical Classifications Software|short format|
|Comorbidity |short format|
|PheWAS|decimal format|
舉例來說,當使用者欲進行 CCS 的標準化分組,在分組前必須先將資料的診斷編碼統一轉換為 short 的格式
疾病分組時,為能區別疾病分類編碼版本 ICD-9/ICD-10,本套件提供兩種方式區分編碼的版本,一者為資料中 記錄 ICD 版本之欄位,另一者則依據 診斷日期,其時間切割點可依使用者需求設定。
如使用者分析台灣的醫療資料,ICD-10 使用日期設定為 2016 年 1 月 1 日;
如使用者分析美國的醫療資料,ICD-10 使用日期設定為 2015 年 10 月 1 日。
如範例,區分診斷編碼版本的日期為:2015年10月1日
將 ICD-CM code 的格式統一轉成 Decimal
# Short to decimal
decimal <- icdDxShortToDecimal(dxDataFile = sampleDxFile,
icdColName = ICD,
dateColName = Date,
icd10usingDate = "2015/10/01")
decimal$ICD[6:10]
將 ICD-CM code 的格式統一轉成 Short
# Decimal to short
short <- icdDxDecimalToShort(dxDataFile = sampleDxFile,
icdColName = ICD,
dateColName = Date,
icd10usingDate = "2015/10/01")
short$ICD[6:10]
進行格式轉換後,會得到兩項回傳的結果:轉換成統一格式之 ICD、診斷編碼錯誤列表 Error。
warning message
醫療數據的診斷編碼有可能誤植導致診斷編碼錯誤,而錯誤的診斷編碼將影響後續臨床疾病分組錯誤或是無法分組的情形。
為方便使用者修改錯誤的診斷編碼,dxpr 套件提供 warning message 提醒使用者錯誤的診斷編碼。
編碼錯誤的種類:格式錯誤 (wrong format)及版本分類錯誤 (wrong version)
sample <- icdDxShortToDecimal(dxDataFile = sampleDxFile,
icdColName = ICD,
dateColName = Date,
icd10usingDate = "2015/10/01")
從上述例子中,可見診斷編碼錯誤相關訊息。
1) 診斷編碼格式錯誤:共 9 種診斷編碼(括號內為該診斷編碼出現次數)
c("A0.11 (20)", "E114 (8)", "Z9.90 (6)", "F42 (6)", "001 (5)", "75.52 (4)", "755.2 (3)", "123.45 (3)", "7552 (2)")
2) 診斷編碼版本錯誤:共 7 種診斷編碼(括號內為診斷編碼出現次數)
c("V27.0 (18)", "A01.05 (8)", "42761 (7)", "V24.1 (6)", "A0105 (5)", "E03.0 (4)", "650 (4)")
函式回傳之Error會顯示資料中全部的錯誤,顯示錯誤的診斷編碼、出現次數、在檔案中編碼被判斷為何種版本、錯誤的類別及建議該如何修正
sample$Error
除了錯誤編碼表格供使用者了解錯誤的種類數量;本套件另提供圖表功能(plotICDError)供使用者觀察錯誤編碼的分布情形。
Warning message判斷流程
以下為判斷診斷編碼錯誤流程圖,以診斷編碼轉為 decimal 作為範例
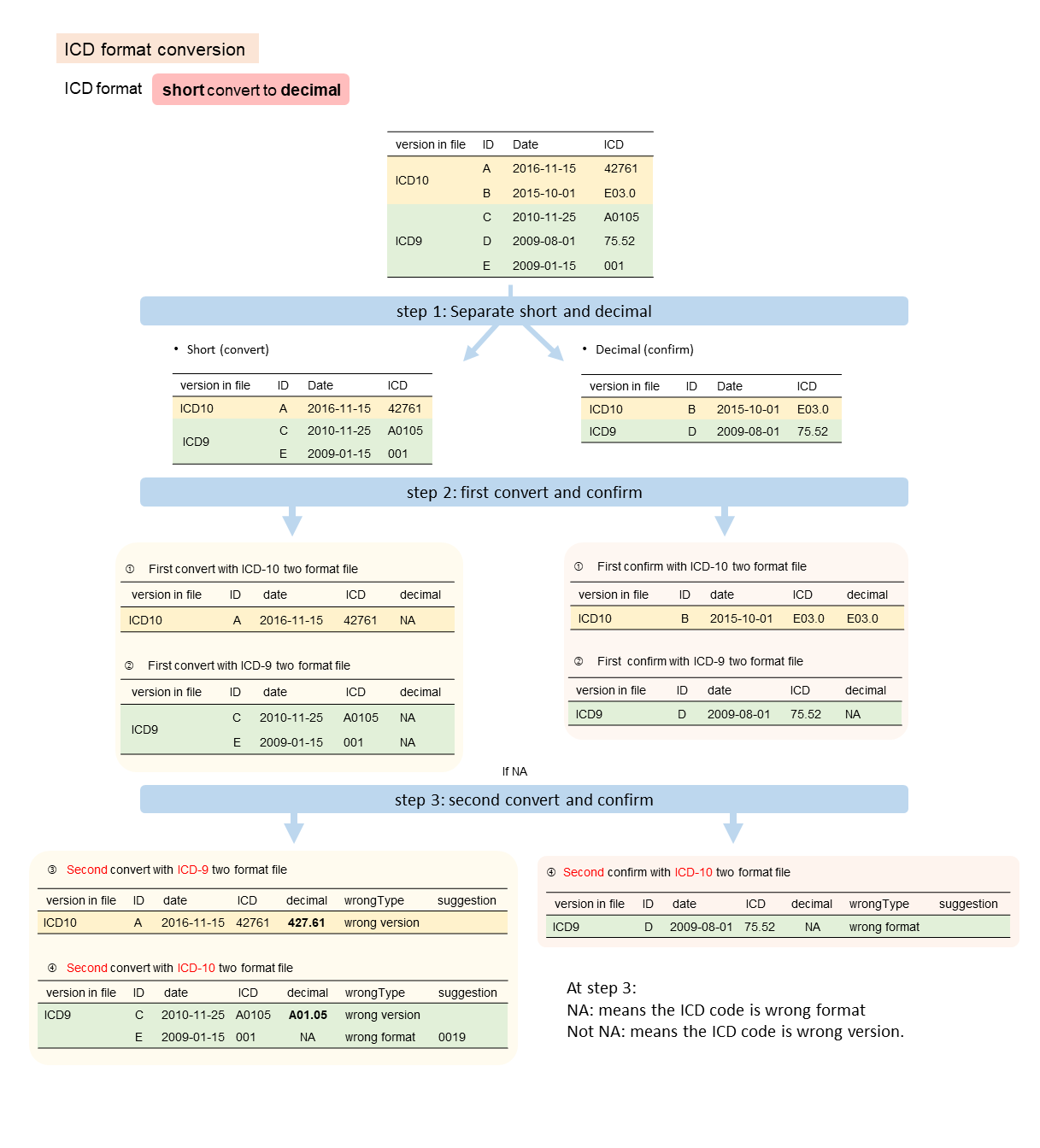
- 將資料依照格式依診斷日期分為ICD-9及ICD-10兩部分
- Step 1: 將資料分為 short 及 decimal
- Step 2: 第一次格式轉換及確認格式正確性
Short 的組別: 依照診斷編碼版本分組進行第一次格式轉換
ICD-9: C, E
ICD-10: A
Decimal 的組別: 依診斷編碼版本分組進行第一次確認格式正確性
ICD-9: D
ICD-10: B
- Step 3: 挑出有 NA 的個案 (A, C, D, E),進行第二次的格式轉換和確認正確性。
A為ICD-10 code,因此將與ICD9DxwithTwoFormat進行第二次的確認及轉換
C, D, E為ICD-9 code,因此將與ICD10DxwithTwoFormat進行第二次的確認及轉換
若結果為NA,則表示該診斷編碼格式錯誤;反之則表示該診斷編碼版本錯誤
如圖
A和C 轉換格式的結果不是NA,表示診斷編碼版本錯誤(42761應為ICD 9的診斷編碼,A0105應為ICD 10的診斷編碼)
D和E 轉換格式的結果為NA,表示診斷編格式錯誤 (應為755.2和001.9)
二、疾病分組
將分散的診斷編碼基於臨床意義整合成較大群組的疾病分組功能,以供後續分析使用。
目前套件提供四種標準化分組的種類,包括CCS階層式編碼轉換、PheWAS分群編碼、共病症(AHRQ、Charlson、Elixhauser Comorbidity)分組以及自定義診斷分組,以下將分別介紹之。
經過四種標準化處理後,可得三項回傳的結果:
1) groupedDT:如表二,將診斷編碼依國際標準轉換為有臨床意義的疾病分組。groupedDT將顯示完整的分組結果,如範例為300筆資料,groupedDT就會顯示300筆
用途:計算疾病世代 (condition era)、選取合適的case組(selectCase)以及後續將資料轉為適合進行統計分析之寬表型態。
表二 標準化分組後回傳的資料型態
Short/Decimal|ID|ICD|Date|GroupType
----|--|-----|---|----
ICD short/Decimal格式|病患編號|使用者輸入的診斷編碼 |診斷日期|標準化分組組別
2) summarised_groupedDT:如表三,將病患被歸類在同一個組別的多筆資料會統整成一筆資料。
表三 標準化分組後回傳的長表資料型態
ID|GroupType|FirstCaseDate|EndCaseDate|Count|Period
--|----|---|---|---|-----
病患編號|標準化分組|第一筆資料時間|最後一筆資料時間|個數|第一筆資料與最後一筆資料相隔週期
取診斷資料中最早診斷的時間為first case date,診斷資料中最後記錄的時間為end case date,並計算其週期(period)內相差的天數,以及週期內共有幾筆診斷紀錄(count)。
其中CCS單階層、多階層、PheWAS及共病症的分組類型另可選擇為"分類"或"分類敘述" (isDescription = TRUE or FALSE)
舉例來說CCS單階層中 CCS分組編號為 1 者,其組別敘述為"Tuberculosis";使用者可自行選擇呈現方式。
3) Error:若 ICD 格式仍未在格式轉換階段進行修正,則進行標準化分組時,會遇到 ICD 格式錯誤的情況,將回傳錯誤 ICD 列表。
1. CCS 臨床分類軟體
美國Healthcare Cost and Utilization Project (HCUP)發展出一套診斷與處置的臨床分組標準CCS。基於此機制,將分散的診斷碼依照其臨床意義進行單層式或階層式的標準化診斷整合分類,其階層式診斷分類可依使用需求調整分類標準。
- 單一階層:較通用的分類
- 多階層:將診斷碼分為較精確的分組
1) CCS單一階層分類
icdDxToCCS 可取得診斷編碼相對應的 CCS 分類 (CCS category) 及分類之敘述,ICD-9-CM 和 ICD-10-CM 的 CCS 單一階層一致:共260種。
## ICD to CCS category's description
CCS_description <- icdDxToCCS(dxDataFile = sampleDxFile,
idColName = ID,
icdColName = ICD,
dateColName = Date,
icd10usingDate = "2015-10-01",
isDescription = TRUE)
head(CCS_description$groupedDT, 5)
head(CCS_description$summarised_groupedDT, 5)
## ICD to CCS category
CCS_category <- icdDxToCCS(dxDataFile = sampleDxFile,
idColName = ID,
icdColName = ICD,
dateColName = Date,
icd10usingDate = "2015-10-01",
isDescription = FALSE)
2) CCS多階層分類
icdDxToCCSLvl 可取得診斷編碼相對應的 CCS 多階層分類 (Multiple CCS level) 及分類之敘述。
- ICD-9-CM 共 4 個階層 (Level 1~4)
- ICD-10-CM 共 2 個階層 (Level 1~2)
## ICD to CCS multiple level 2 description
CCSlvl_description <- icdDxToCCSLvl(dxDataFile = sampleDxFile,
idColName = ID,
icdColName = ICD,
dateColName = Date,
icd10usingDate = "2015-10-01",
CCSLevel = 2,
isDescription = TRUE)
head(CCSlvl_description$groupedDT, 5)
head(CCSlvl_description$summarised_groupedDT, 5)
## ICD to CCS multiple level 3 category
CCSLvl_category <- icdDxToCCSLvl(dxDataFile = sampleDxFile,
idColName = ID,
icdColName = ICD,
dateColName = Date,
icd10usingDate = "2015-10-01",
CCSLevel = 3,
isDescription = FALSE)
2. PheWAS
由 Vanderbilt University Medical Center 開發的 PheWAS 工具,將分散的診斷碼依照其臨床意義進行標準化分組,ICD和PheWAS為多對多的配對關係,ICD-9-CM共1,866種的PheWAS分類,ICD-10-CM則有1,755種的PheWAS分類。
icdDxToPheWAS可取得診斷碼相對應的 PheWAS 分類及分類敘述。
dxpr套件使用 version 1.2 (phecode_icd9_2) 以及 beta 版本之 version 1.2 (phecode_icd10) 進行標準化分組。
## ICD to PheWAS
PheWAS <- icdDxToPheWAS(dxDataFile = sampleDxFile,
idColName = ID,
icdColName = ICD,
dateColName = Date,
icd10usingDate = "2015-10-01",
isDescription = FALSE)
PheWAS$groupedDT[7:11]
PheWAS$summarised_groupedDT[7:11]
3. 共病症
共病症(Comorbidities)為病患於主診斷之外,其他已存在且會對主診斷疾病產生影響的疾病症狀,共病症分群基於診斷編碼進行標準化分組, 根據不同測量方法對共病症的定義分別介紹之:AHRQ、Charlson及Elixhauser。
1) Elixhauser
Elixhauser Comorbidity為 Dr. Elixhauser 與其團隊在1998年提出基於標準ICD診斷碼的共病症分類分組標準,Elixhauser database共病症測量指標為 HCUP 發展的共病症資料庫之一,ICD-9-CM 及 ICD-10-CM 的 Elixhauser 共病症種類一致,共 29 種。
欲將診斷編碼依照 Elixhauser 共病症分類,在 function icdDxToComorbid 的comorbidMethod 輸入 elix
ELIX <- icdDxToComorbid(dxDataFile = sampleDxFile,
idColName = ID,
icdColName = ICD,
dateColName = Date,
icd10usingDate = "2015-10-01",
comorbidMethod = elix)
ELIX$groupedDT[160:164]
head(ELIX$summarised_groupedDT, 5)
2) AHRQ
於2017年基於 Elixhauser Comorbidity 更進一步設計 AHRQ Elixhauser Comorbidity Index,此index為單一的風險評估分數,可用於再入院與死亡的風險評估。
ICD-9-CM 及 ICD-10-CM 的 AHRQ 共病症種類一致,共30種。
欲將診斷編碼依照 AHRQ 共病症分類,在 function icdDxToComorbid 的 comorbidMethod 輸入 AHRQ
AHRQ <- icdDxToComorbid(dxDataFile = sampleDxFile,
idColName = ID,
icdColName = ICD,
dateColName = Date,
icd10usingDate = "2015-10-01",
comorbidMethod = AHRQ)
AHRQ$groupedDT[160:164]
head(AHRQ$summarised_groupedDT, 5)
3) Charlson
Charlson database 共病症測量指標的是基於 Quan 的 Charlson Comorbidity Index 版本,依診斷編碼其相關的共病症做分類。
ICD-9-CM 及 ICD-10-CM 的 Charlson 共病症種類一致,共17種。
欲將診斷編碼依照 Charlson 共病症分類,在 function icdDxToComorbid 的comorbidMethod 輸入 Charlson
Charlson <- icdDxToComorbid(dxDataFile = sampleDxFile,
idColName = ID,
icdColName = ICD,
dateColName = Date,
icd10usingDate = "2015-10-01",
comorbidMethod = charlson)
Charlson$groupedDT[160:164]
head(Charlson$summarised_groupedDT, 5)
4. 自定義診斷碼分組
因應臨床研究需求,本套件採多樣的分組方式以維持診斷醫療大數據分析的彈性,提供進階使用者自行定義診斷分組類別及類別項目,使用者可選擇精確比對或是模糊比對的方式訂定篩選的分組類別及類別項目進行診斷碼的分群。
範例如下,使用者欲定義符合慢性腎臟疾病(Chronic kidney disease)的疾病編碼,相關診斷編碼如下:
- ICD-9:5851, 5852,..., 5859
- ICD-10:N181, N182,..., N189
自定義診斷分組提供兩種分組方法供使用者選擇,將符合條件的診斷編碼分類為心律異常的組別。
1) icdDxToCustom:精確比對
使用者須定義精確分組的表格 (groupingTable),輸入詳細的診斷編碼 (N181, 5853, 5854…),
進行分組的資料須要完全符合,才會歸類為 Cardiac dysrhythmias,另外不符合條件的組別則顯示 NA。
# CustomGroupingTable
groupingTable <- data.frame(Group = rep("Chronic kidney disease",6),
ICD = c("N181","5853","5854","5855","5856","5859"),
stringsAsFactors = FALSE)
CustomGroup <- icdDxToCustom(dxDataFile = sampleDxFile,
idColName = ID,
icdColName = ICD,
dateColName = Date,
customGroupingTable = groupingTable)
CustomGroup$groupedDT[10:14]
head(CustomGroup$summarised_groupedDT, 5)
2) icdDxToCustomGrep:模糊比對
使用者須定義模糊分組的表格 (grepTable),以字串比對之正規表示式 (585*\ , N18*\…),當資料開頭為585*\ (ICD-9-CM) 或是N18*\ (ICD-10-CM) 即符合使用者定義之組別條件 Chronic kidney disease,另外不符合條件的組別則顯示 NA。
# CustomGroupingTable
grepTable <- data.frame(Group = "Chronic kidney disease",
grepIcd = "^585|^N18",
stringsAsFactors = FALSE)
CustomGrepGroup <- icdDxToCustomGrep(dxDataFile = sampleDxFile,
idColName = ID,
icdColName = ICD,
dateColName = Date,
customGroupingTable = grepTable)
CustomGrepGroup$groupedDT[10:14]
head(CustomGrepGroup$summarised_groupedDT, 5)
II. Data Wrangling
一、篩選符合條件的個案
將醫療大數據診斷資訊進行標準化處理與整合後,將依據使用者所設立的條件篩選符合條件的個案。篩選個案的功能和疾病世代的概念類似,適用於一般疾病(限制搜尋範圍)或慢性病(沒有限制搜尋範圍)的計算。
為選取符合特定條件之診斷資料,目前有三種條件進行篩選:
1) 設立篩選條件
`groupDataType`:設定篩選的分組依據,可用 CCS、PheWAS、comorbidity、自定義組別或 ICD 診斷編碼
`caseCondition`:設定篩選的條件,如:以 CCS 為分組依據,則條件可為特定 CCS 編號 (`isDescription` = FALSE) 或特定 CCS 分類敘述 (`isDescription` = TRUE) ,條件字串為精確比對,使用者亦可輸入正規表達式進行模糊比對
2) 設定搜尋範圍
periodRange:限制每筆診斷資料之間的間隔範圍,預設為間隔30天至365天時間內的資料
3) ICD次數
caseCount:符合篩選方式的最小出現次數,預設至少出現兩次
最後回傳符合條件的個案資料如表四。
表四 選取特定個案診斷紀錄後回傳的資料型態
|ID|Count|FirstCaseDate|EndCaseDate|Period|MostCommonICD|MostCommonICDCount|
|--|--|---|---|--|-----|-----|
|病患編碼|個數|第一筆資料時間|最後一筆資料時間|第一筆資料與最後一筆資料相隔週期| 出現最多次數的診斷編碼|最常出現診斷編碼的次數 |
符合條件的個案將整合為一筆資料,內容為符合條件的資料總數(count),第一筆診斷資料(first case date)及最後一筆資料時間(end case date)及資料時間相隔週期(period),出現最多次的ICD及次數(most common ICD 和 most common ICD count)。
符合篩選條件會在欄位 selectedCase 顯示 "Selected",使用者也可自行定義組別名稱 (在 caseName 引數項中設定)
Case <- selectCases(dxDataFile = sampleDxFile,
idColName = ID,
icdColName = ICD,
dateColName = Date,
groupDataType = ccslvl2,
icd10usingDate = "2015/10/01",
isDescription = TRUE,
caseCondition = "Diseases of the urinary system",
caseCount = 1,
caseName = "Selected")
head(Case)
以上述例子結果之第一筆做說明,個案 A3 符合篩選條件的診斷資料共 5 筆,第一筆診斷日期為 "2008-07-08",最後一筆診斷紀錄為 "2014-02-24"。其中 V420 出現次數最高,共 3 筆。整個診斷紀錄週期為 2057 天。
二、就診紀錄第一筆資料及最後一筆資料
取得病患的就診紀錄第一筆資料及最後一筆資料,可用來做為個案的指標日期供 splitDataByDate 使用。
admissionDate <- getEligiblePeriod(dxDataFile = sampleDxFile,
idColName = ID,
dateColName = Date)
head(admissionDate)
三、以事件(index date)區分事件前後的醫療紀錄
醫學研究常需要觀察特定事件發生前後的改變,因此需要一個指標時間(index date)來記錄事件的發生。timeTag 欄位可顯示此紀錄為事件前或是事件後(Before or After),使用者可依此結果再行篩選事件發生前/後的資料。
指標日期 (indexDateFile) 由使用者定義,可依據個案之醫療紀錄個別定義,如使用 getEligiblePeriod函數所得之結果。
另外本套見也提供週期(window)計算功能,使用者可自行定義週期長度 (gap),預設週期長度為30天。
indexDateTable <- data.frame(ID = c("A0","B0","C0","D0"),
indexDate = c("2009-07-25", "2015-12-26",
"2015-12-05", "2017-01-29"),
stringsAsFactors = FALSE)
Data <- splitDataByDate(dxDataFile = sampleDxFile,
idColName = ID,
icdColName = ICD,
dateColName = Date,
indexDateFile = indexDateTable,
gap = 30)
head(Data, 5)
舉例來說,個案 A0 之指標日期為 2009-07-25,則個案 A0 的第一筆診斷資料時間為 2009-07-25 視為指標日期之後發生的紀錄,TimeTag 為 A (after)。兩筆資料間隔天數為 0 天,故為第 1 個週期;個案 A0 的第二筆診斷資料時間為 2010-03-28 ,亦為指標日期後的紀錄,TimeTag 為 A。資料間隔天數為 246 天,為第 9 個週期。
四、 疾病世代
在診斷紀錄整合機制方面,Dr. Ryan 提出疾病世代(Condition era)概念整合處理連續性的醫療行為, 將分散的醫療紀錄整合為單一病程發展紀錄,以利後續醫療大數據分析使用。
疾病世代的概念是針對疾病定義持續期(gap)長度,若任兩個連續診斷之間的間隔小於 gap,則此兩個診斷視為同一個疾病世代。
疾病世代的診斷分類可選擇 ICD 診斷編碼或四種標準化分組,計算病患之疾病世代,其持續期長度預設值為30天(gapDate)。
Era <- getConditionEra(dxDataFile = sampleDxFile,
idColName = ID,
icdColName = ICD,
dateColName = Date,
icd10usingDate = "2015-10-01",
gapDate = 30,
groupDataType = ccs,
isDescription = FALSE)
head(Era)
以範例的第一個結果做說明:個案 A0 被分組到 CCS 類別 158 共有 5 筆資料,第一筆診斷日期為 "2009-07-25" 、最後一筆診斷紀錄為 "2013-12-20",其中任意兩個連續的診斷,相差間隔皆不小於 30 天,所以有 5 個世代。
五、 探索性資料寬表轉換
診斷碼整合分組後,為能進行後續的探索性資料分析(EDA preparation),本套件提供資料型態轉換的功能,以便轉換為更利於進行統計分析的寬表型態。
寬表提供兩種表示方式,以二分法(binary)或是數量(numeric)的方式呈現(numericOrBinary = N 或 B)。當病患沒有相關的疾病診斷,或診斷次數低於使用者設定之閾值(引數 count,預設為 1),則二分法表示為 FALSE,反之,超過閾值的診斷紀錄數目時,二分法表示為 TRUE;數量表示法則計算該病患在相關疾病的分組上共有幾筆疾病診斷資料。
以下範例以共病症 Elixhauser為範例進行兩種表示法回傳的資料型態。
#binary
ELIX <- icdDxToComorbid(dxDataFile = sampleDxFile,
idColName = ID,
icdColName = ICD,
dateColName = Date,
icd10usingDate = "2015-10-01",
comorbidMethod = elix)
groupedData_Wide <- groupedDataLongToWide(dxDataFile = ELIX$groupedDT,
idColName = ID,
categoryColName = Comorbidity,
dateColName = Date,
reDup = TRUE,
numericOrBinary = B)
head(groupedData_Wide, 5)
# numeric
groupedData_Wide <- groupedDataLongToWide(dxDataFile = ELIX$groupedDT,
idColName = ID,
categoryColName = Comorbidity,
dateColName = Date,
reDup = TRUE,
numericOrBinary = N)
head(groupedData_Wide, 5)
III. Visualization
第三部份視覺化:套件將前述功能之結果整合,以圖表形式呈現。
一、 Pareto plot:錯誤編碼之柏拉圖圖表
將第一階段- ICD 診斷編碼格式轉換(Code transformation)過程中產生之錯誤編碼訊息列表換成圖表。
以柏拉圖圖表(Pareto plot)的方式呈現,使用者可得知錯誤編碼的種類及出現比例,並依照錯誤數量依序排名。
errorFile:診斷編碼進行統一格式轉換時產生的錯誤編碼 - 顯示診斷編碼的版本:顯示全部的編碼版本(
ICDVersion = 9, 10 or all)
- 顯示錯誤診斷編碼的種類:顯示全部的錯誤編碼種類(
wrongICDType = version, format or all)
- 預設為顯示前 10 名的錯誤編碼(
TopN = 10)
- 第 10 名以後的錯誤編碼設為"others"(
Others = TRUE)
如欲觀察前十名的錯誤編碼種類及分布比例,則範例如下:
error <- icdDxDecimalToShort(dxDataFile = sampleDxFile,
icdColName = ICD,
dateColName = Date,
icd10usingDate = "2015/10/01")
Plot_error1 <- plotICDError(errorFile = error$Error,
icdVersion = all,
wrongICDType = all,
others = TRUE)
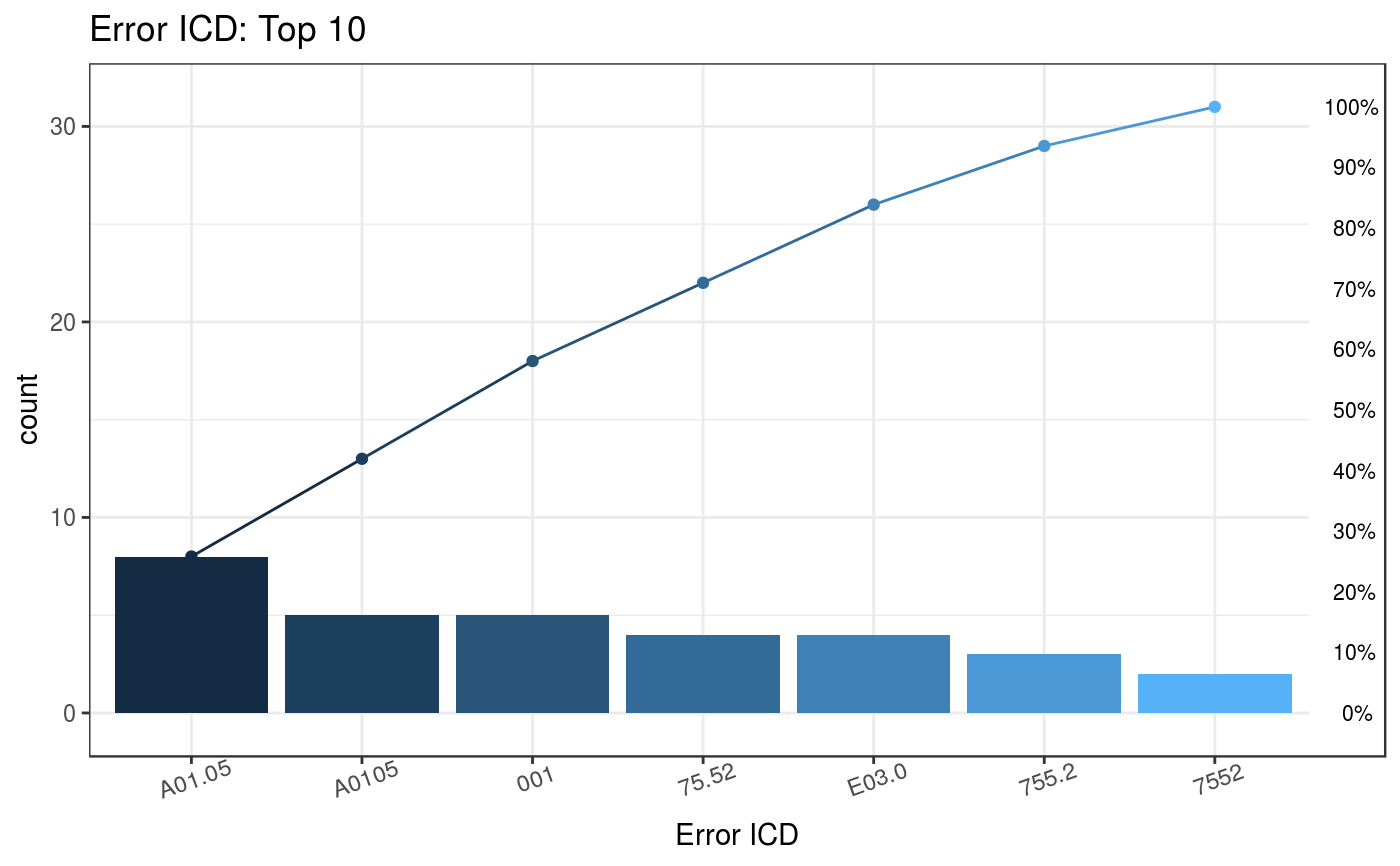 上圖顯示前10名錯誤的診斷編碼,其中第一名的錯誤A0.11共有20筆,佔全部錯誤18.35% (20/109),其餘名次的錯誤編碼則分類至 "Others"。
上圖顯示前10名錯誤的診斷編碼,其中第一名的錯誤A0.11共有20筆,佔全部錯誤18.35% (20/109),其餘名次的錯誤編碼則分類至 "Others"。
Plot_error1$ICD
另外使用者可選擇將 ICD-9 依照章節分組 (groupICD = TRUE);僅 ICD9 有此功能, ICD10 本身已有前綴之字母編號故無須額外編組。
ICD-9 共 19 章節疾病分類:
001-139:傳染病或寄生蟲相關疾病
140-239:腫瘤相關疾病
...等。
plotICDError依照編碼開頭分類,大致分為 12 組:0, 1, 2,..., 9, V 和 E
如欲觀察 ICD9 的前三名錯誤編碼種類及分布比例,則範例如下。
Plot_error2 <- plotICDError(errorFile = error$Error,
icdVersion = 9,
wrongICDType = all,
groupICD = TRUE,
others = TRUE,
topN = 3)
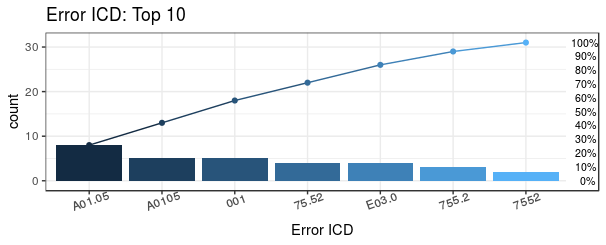
icdVersion設為 9,緊顯示 ICD9 版本之錯誤編碼,並設 groupICD 為 TRUE,使 ICD9 依編碼開頭分組,
顯示前三名的錯誤編碼
第3名以後的錯誤編碼設為"others"
由下表格得知第一名的錯誤編碼為A01.05佔組別A共61.54%,總共有13筆錯誤,佔全部的錯誤的比例為41.94%
Plot_error2$ICD
#> ICDGroup groupCount CumCountPerc MostICDInGroup ICDPercInGroup WrongType
#> 1: A 13 41.94% A01.05 61.54% Wrong version
#> 2: 7 9 70.97% 75.52 44.44% Wrong format
#> 3: 0 5 87.1% 001 100% Wrong format
#> 4: Others 4 100% E03.0 100% Wrong version
二、 histogram plot:標準化分組之長條圖表
將第二階段-探索性資料轉換之寬表資料圖形化,以便觀察診斷資料在各分群(如 comorbidity)的分布情形及數量。
使用者可選擇顯示前幾名的分組資料(Top N),且可限制每個分群的病患至少有一定比例(limit Frequency)。
- 預設為顯示前 10 名的錯誤編碼(
TopN = 10)
- 限制最低比例預設為 1% (
limitFreq = 0.01);假設使用者的資料共 1000 個病患,則每種組別至少要有 10 個病患
範例使用步驟如下:
ELIX <- icdDxToComorbid(dxDataFile = sampleDxFile,
idColName = ID,
icdColName = ICD,
dateColName = Date,
icd10usingDate = "2015-10-01",
comorbidMethod = elix)
groupedDataWide <- groupedDataLongToWide(dxDataFile = ELIX$groupedDT,
idColName = ID,
categoryColName = Comorbidity,
dateColName = Date,
reDup = TRUE,
numericOrBinary = B)
plot1 <- plotDiagCat(groupedDataWide = groupedDataWide,
idColName = ID,
topN = 10,
limitFreq = 0.01)
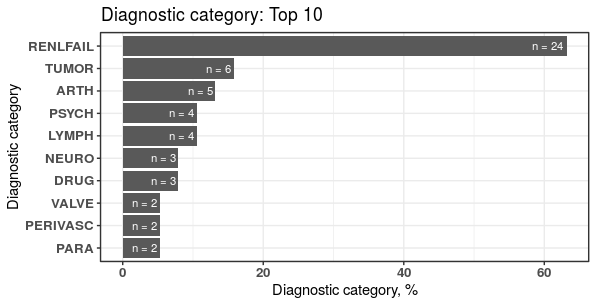
上述程式碼先將資料以共病症 Elixhauser 做標準化分組並轉成寬表,最後製圖:顯示前 10 名、至少 1% 比例的組別。
下表格為符合條件設定的共病症前十名,每組分別有多少人,且該共病症在資料中的比例。
本範例共有 38 個病患,以第一組為例,共有 24 人被分為 RENLFAIL 共病症,比例為 63.16% (24/38)
plot1$sigCate
若資料有進行病患分組 Case/Control,於每個類別(如comorbidity)將進行兩組(Case and Control)的卡方獨立性檢定,圖表將顯示達統計上顯著差異的分組類別。
若樣本數過小以致不適用卡方檢定時,則採用費雪爾精確檢定(Fisher’s Exact Test)
顯著差異 p-value 預設門檻為 0.05,預設顯示前 10 名的類別,且每群種類的病患在兩組的比率至少有一組達 1%
舉例來說,假設 Case 和 Control 組各 500 人,在某特定組別至少有一組的人數有 5 個病患 (1%)。
範例如下:
selectedCaseFile <- selectCases(dxDataFile = sampleDxFile,
idColName = ID,
icdColName = ICD,
dateColName = Date,
icd10usingDate = "2015/10/01",
groupDataType = ccslvl2,
caseCondition = "Diseases of the urinary system",
caseCount = 1)
ELIX <- icdDxToComorbid(dxDataFile = sampleDxFile,
idColName = ID,
icdColName = ICD,
dateColName = Date,
icd10usingDate = "2015-10-01",
comorbidMethod = elix)
groupedData_Wide <- groupedDataLongToWide(dxDataFile = ELIX$groupedDT,
idColName = ID,
categoryColName = Comorbidity,
dateColName = Date,
reDup = TRUE,
selectedCaseFile = selectedCaseFile)
plot2 <- plotDiagCat(groupedDataWide = groupedData_Wide,
idColName = ID,
groupColName = selectedCase,
topN = 10,
limitFreq = 0.01,
pvalue = 0.05)
上述範例之步驟為:
- 定義 case/control 組,採 CCS 多階層的標準化分組,在 CCS level 2 被分組為 "Diseases of the urinary system" 者視為 case 組,詳細設定條件請參照函式
selectCases 之範例。
- 將資料以共病症 Elixhauser 做標準化分組並轉成寬表
- 設定作圖標準:顯示前幾名組別、限制比例、p 值(p-value)。
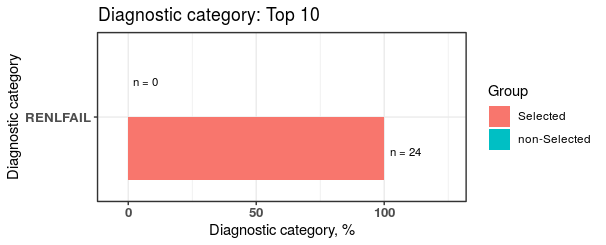
下表格為在 selected / nonSelected 兩組間達顯著差異的共病症組別 RENLFAIL
在 selected 組佔了 100% (24/24),在 nonSelected 組佔了0% (0/14)
plot2$sigCate
Reference
I. Code tramsformation
ICD-9-CM code (2014): https://www.cms.gov/Medicare/Coding/ICD9ProviderDiagnosticCodes/codes.html
ICD-10-CM code (2019): https://www.cms.gov/Medicare/Coding/ICD10/2019-ICD-10-CM.html
https://www.cms.gov/Medicare/Coding/ICD10/2019-ICD-10-CM.html
https://www.findacode.com/search/search.php
https://www.cms.gov/Medicare/Quality-Initiatives-Patient-Assessment-Instruments/HospitalQualityInits/Downloads/HospitalAppendix_F.pdf
II. Code grouping
本套件使用之CCS版本依照HCUP提供之最新版本進行年度的更新,目前ICD-9-CM與ICD-10-CM的CCS定義版本分別為2015年及2019年版。
PheWAS依照PheWAS Resources網站 進行更新,目前版本為第二版(2015年)。
Elixhauser共病症依照HCUP提供之版本進行更新,目前ICD-9-CM與ICD-10的Elixhauser共病症定義版本分別為2015年及2019年版。
CCS (Clinical Classifications Software)
ICD-9-CM (2015): https://www.hcup-us.ahrq.gov/toolssoftware/ccs/Single_Level_CCS_2015.zip
https://www.hcup-us.ahrq.gov/toolssoftware/ccs/Multi_Level_CCS_2015.zip
ICD-10-CM (2019): https://www.hcup-us.ahrq.gov/toolssoftware/ccs10/ccs_dx_icd10cm_2019_1.zip
PheWAS
ICD-9-Phecode (version 1.2, 2015): https://phewascatalog.org/phecodes
ICD-10 Phecode (version 1.2 beta, 2019): https://phewascatalog.org/phecodes_icd10cm
Comorbidities
ICD-9-AHRQ (2012-2015): https://www.hcup-us.ahrq.gov/toolssoftware/comorbidity/comorbidity.jsp#references
ICD-10-AHRQ (2019): https://www.hcup-us.ahrq.gov/toolssoftware/comorbidityicd10/comformat_icd10cm_2019_1.txt
ICD-9-Charlson: http://mchp-appserv.cpe.umanitoba.ca/Upload/SAS/ICD9_E_Charlson.sas.txt
ICD-10-Charlson: http://mchp-appserv.cpe.umanitoba.ca/Upload/SAS/ICD10_Charlson.sas.txt
ICD-9-Elixhauser (2012-2015): https://www.hcup-us.ahrq.gov/toolssoftware/comorbidity/comorbidity.jsp#references
ICD-10-Elixhauser (2019): https://www.hcup-us.ahrq.gov/toolssoftware/comorbidityicd10/comformat_icd10cm_2019_1.txt
DHLab-TSENG/dxpr documentation built on Sept. 1, 2023, 8:12 a.m.
診斷前處理與整合方法
cat(utils::packageDescription("dxpr")$Description)
安裝套件
install.packages("remotes") # Install development version from GitHub remotes::install_github("DHLab-TSENG/dxpr") library(dxpr)
knitr::opts_chunk$set(collapse = T, comment = "#>") options(tibble.print_min = 4L, tibble.print_max = 4L) #remotes::install_github("DHLab-TSENG/dxpr") library(dxpr)
ICD-CM-CODE 格式對照
本套件使用的診斷編碼依照 CMS (Centers for Medicare & Medicaid Services)所提供的診斷編碼做為標準依據,並依照WHO訂定的診斷編碼規則,分別產生 ICD-9-CM and ICD-10-CM 的兩種格式的表格: ICD9DxwithTwoFormat 和 ICD10DxwithTwoFormat 供後續診斷編碼格式轉換的功能使用
ICD-9-CM
# Short head(ICD9DxwithTwoFormat$Short) # Decimal head(ICD9DxwithTwoFormat$Decimal)
ICD-10-CM
# Short head(ICD10DxwithTwoFormat$Short) # Decimal head(ICD10DxwithTwoFormat$Decimal)
範例資料
以下功能介紹皆以sampleDxFile做為範例,共有38位病患,300筆診斷紀錄
head(sampleDxFile)
I. Data Integration
一、格式轉換
ICD-CM code兩種格式的轉換:Short <-> Decimal
將醫療大數據中的診斷編碼進行編碼一致格式的轉換,以便後續診斷編碼的標準化分群。依據 Clinical Classifications Software (CCS)、共病症 (Comorbidity) 及 Phenome-wide association studies (PheWAS) 的分組表,其分別使用之 ICD 診斷碼格式如表一。
表一 標準化分組之診斷編碼格式
| |ICD format| |--------|----| |Clinical Classifications Software|short format| |Comorbidity |short format| |PheWAS|decimal format|
舉例來說,當使用者欲進行 CCS 的標準化分組,在分組前必須先將資料的診斷編碼統一轉換為 short 的格式
疾病分組時,為能區別疾病分類編碼版本 ICD-9/ICD-10,本套件提供兩種方式區分編碼的版本,一者為資料中 記錄 ICD 版本之欄位,另一者則依據 診斷日期,其時間切割點可依使用者需求設定。
如使用者分析台灣的醫療資料,ICD-10 使用日期設定為 2016 年 1 月 1 日;
如使用者分析美國的醫療資料,ICD-10 使用日期設定為 2015 年 10 月 1 日。
如範例,區分診斷編碼版本的日期為:2015年10月1日
將 ICD-CM code 的格式統一轉成 Decimal
# Short to decimal decimal <- icdDxShortToDecimal(dxDataFile = sampleDxFile, icdColName = ICD, dateColName = Date, icd10usingDate = "2015/10/01") decimal$ICD[6:10]
將 ICD-CM code 的格式統一轉成 Short
# Decimal to short short <- icdDxDecimalToShort(dxDataFile = sampleDxFile, icdColName = ICD, dateColName = Date, icd10usingDate = "2015/10/01") short$ICD[6:10]
進行格式轉換後,會得到兩項回傳的結果:轉換成統一格式之 ICD、診斷編碼錯誤列表 Error。
warning message
醫療數據的診斷編碼有可能誤植導致診斷編碼錯誤,而錯誤的診斷編碼將影響後續臨床疾病分組錯誤或是無法分組的情形。
為方便使用者修改錯誤的診斷編碼,dxpr 套件提供 warning message 提醒使用者錯誤的診斷編碼。
編碼錯誤的種類:格式錯誤 (wrong format)及版本分類錯誤 (wrong version)
sample <- icdDxShortToDecimal(dxDataFile = sampleDxFile, icdColName = ICD, dateColName = Date, icd10usingDate = "2015/10/01")
從上述例子中,可見診斷編碼錯誤相關訊息。
1) 診斷編碼格式錯誤:共 9 種診斷編碼(括號內為該診斷編碼出現次數)
c("A0.11 (20)", "E114 (8)", "Z9.90 (6)", "F42 (6)", "001 (5)", "75.52 (4)", "755.2 (3)", "123.45 (3)", "7552 (2)")
2) 診斷編碼版本錯誤:共 7 種診斷編碼(括號內為診斷編碼出現次數)
c("V27.0 (18)", "A01.05 (8)", "42761 (7)", "V24.1 (6)", "A0105 (5)", "E03.0 (4)", "650 (4)")
函式回傳之Error會顯示資料中全部的錯誤,顯示錯誤的診斷編碼、出現次數、在檔案中編碼被判斷為何種版本、錯誤的類別及建議該如何修正
sample$Error
除了錯誤編碼表格供使用者了解錯誤的種類數量;本套件另提供圖表功能(plotICDError)供使用者觀察錯誤編碼的分布情形。
Warning message判斷流程
以下為判斷診斷編碼錯誤流程圖,以診斷編碼轉為 decimal 作為範例

- 將資料依照格式依診斷日期分為ICD-9及ICD-10兩部分
- Step 1: 將資料分為 short 及 decimal
- Step 2: 第一次格式轉換及確認格式正確性
Short 的組別: 依照診斷編碼版本分組進行第一次格式轉換ICD-9: C, E
ICD-10: A
Decimal 的組別: 依診斷編碼版本分組進行第一次確認格式正確性
ICD-9: D
ICD-10: B - Step 3: 挑出有 NA 的個案 (A, C, D, E),進行第二次的格式轉換和確認正確性。
A為ICD-10 code,因此將與
ICD9DxwithTwoFormat進行第二次的確認及轉換
C, D, E為ICD-9 code,因此將與ICD10DxwithTwoFormat進行第二次的確認及轉換
若結果為NA,則表示該診斷編碼格式錯誤;反之則表示該診斷編碼版本錯誤
如圖
A和C 轉換格式的結果不是NA,表示診斷編碼版本錯誤(42761應為ICD 9的診斷編碼,A0105應為ICD 10的診斷編碼)
D和E 轉換格式的結果為NA,表示診斷編格式錯誤 (應為755.2和001.9)
二、疾病分組
將分散的診斷編碼基於臨床意義整合成較大群組的疾病分組功能,以供後續分析使用。
目前套件提供四種標準化分組的種類,包括CCS階層式編碼轉換、PheWAS分群編碼、共病症(AHRQ、Charlson、Elixhauser Comorbidity)分組以及自定義診斷分組,以下將分別介紹之。
經過四種標準化處理後,可得三項回傳的結果:
1) groupedDT:如表二,將診斷編碼依國際標準轉換為有臨床意義的疾病分組。groupedDT將顯示完整的分組結果,如範例為300筆資料,groupedDT就會顯示300筆
用途:計算疾病世代 (condition era)、選取合適的case組(selectCase)以及後續將資料轉為適合進行統計分析之寬表型態。
表二 標準化分組後回傳的資料型態
Short/Decimal|ID|ICD|Date|GroupType ----|--|-----|---|---- ICD short/Decimal格式|病患編號|使用者輸入的診斷編碼 |診斷日期|標準化分組組別
2) summarised_groupedDT:如表三,將病患被歸類在同一個組別的多筆資料會統整成一筆資料。
表三 標準化分組後回傳的長表資料型態
ID|GroupType|FirstCaseDate|EndCaseDate|Count|Period --|----|---|---|---|----- 病患編號|標準化分組|第一筆資料時間|最後一筆資料時間|個數|第一筆資料與最後一筆資料相隔週期
取診斷資料中最早診斷的時間為first case date,診斷資料中最後記錄的時間為end case date,並計算其週期(period)內相差的天數,以及週期內共有幾筆診斷紀錄(count)。
其中CCS單階層、多階層、PheWAS及共病症的分組類型另可選擇為"分類"或"分類敘述" (isDescription = TRUE or FALSE)
舉例來說CCS單階層中 CCS分組編號為 1 者,其組別敘述為"Tuberculosis";使用者可自行選擇呈現方式。
3) Error:若 ICD 格式仍未在格式轉換階段進行修正,則進行標準化分組時,會遇到 ICD 格式錯誤的情況,將回傳錯誤 ICD 列表。
1. CCS 臨床分類軟體
美國Healthcare Cost and Utilization Project (HCUP)發展出一套診斷與處置的臨床分組標準CCS。基於此機制,將分散的診斷碼依照其臨床意義進行單層式或階層式的標準化診斷整合分類,其階層式診斷分類可依使用需求調整分類標準。
- 單一階層:較通用的分類
- 多階層:將診斷碼分為較精確的分組
1) CCS單一階層分類
icdDxToCCS 可取得診斷編碼相對應的 CCS 分類 (CCS category) 及分類之敘述,ICD-9-CM 和 ICD-10-CM 的 CCS 單一階層一致:共260種。
## ICD to CCS category's description CCS_description <- icdDxToCCS(dxDataFile = sampleDxFile, idColName = ID, icdColName = ICD, dateColName = Date, icd10usingDate = "2015-10-01", isDescription = TRUE) head(CCS_description$groupedDT, 5) head(CCS_description$summarised_groupedDT, 5) ## ICD to CCS category CCS_category <- icdDxToCCS(dxDataFile = sampleDxFile, idColName = ID, icdColName = ICD, dateColName = Date, icd10usingDate = "2015-10-01", isDescription = FALSE)
2) CCS多階層分類
icdDxToCCSLvl 可取得診斷編碼相對應的 CCS 多階層分類 (Multiple CCS level) 及分類之敘述。
- ICD-9-CM 共 4 個階層 (Level 1~4)
- ICD-10-CM 共 2 個階層 (Level 1~2)
## ICD to CCS multiple level 2 description CCSlvl_description <- icdDxToCCSLvl(dxDataFile = sampleDxFile, idColName = ID, icdColName = ICD, dateColName = Date, icd10usingDate = "2015-10-01", CCSLevel = 2, isDescription = TRUE) head(CCSlvl_description$groupedDT, 5) head(CCSlvl_description$summarised_groupedDT, 5) ## ICD to CCS multiple level 3 category CCSLvl_category <- icdDxToCCSLvl(dxDataFile = sampleDxFile, idColName = ID, icdColName = ICD, dateColName = Date, icd10usingDate = "2015-10-01", CCSLevel = 3, isDescription = FALSE)
2. PheWAS
由 Vanderbilt University Medical Center 開發的 PheWAS 工具,將分散的診斷碼依照其臨床意義進行標準化分組,ICD和PheWAS為多對多的配對關係,ICD-9-CM共1,866種的PheWAS分類,ICD-10-CM則有1,755種的PheWAS分類。
icdDxToPheWAS可取得診斷碼相對應的 PheWAS 分類及分類敘述。
dxpr套件使用 version 1.2 (phecode_icd9_2) 以及 beta 版本之 version 1.2 (phecode_icd10) 進行標準化分組。
## ICD to PheWAS PheWAS <- icdDxToPheWAS(dxDataFile = sampleDxFile, idColName = ID, icdColName = ICD, dateColName = Date, icd10usingDate = "2015-10-01", isDescription = FALSE) PheWAS$groupedDT[7:11] PheWAS$summarised_groupedDT[7:11]
3. 共病症
共病症(Comorbidities)為病患於主診斷之外,其他已存在且會對主診斷疾病產生影響的疾病症狀,共病症分群基於診斷編碼進行標準化分組, 根據不同測量方法對共病症的定義分別介紹之:AHRQ、Charlson及Elixhauser。
1) Elixhauser
Elixhauser Comorbidity為 Dr. Elixhauser 與其團隊在1998年提出基於標準ICD診斷碼的共病症分類分組標準,Elixhauser database共病症測量指標為 HCUP 發展的共病症資料庫之一,ICD-9-CM 及 ICD-10-CM 的 Elixhauser 共病症種類一致,共 29 種。
欲將診斷編碼依照 Elixhauser 共病症分類,在 function icdDxToComorbid 的comorbidMethod 輸入 elix
ELIX <- icdDxToComorbid(dxDataFile = sampleDxFile, idColName = ID, icdColName = ICD, dateColName = Date, icd10usingDate = "2015-10-01", comorbidMethod = elix) ELIX$groupedDT[160:164] head(ELIX$summarised_groupedDT, 5)
2) AHRQ
於2017年基於 Elixhauser Comorbidity 更進一步設計 AHRQ Elixhauser Comorbidity Index,此index為單一的風險評估分數,可用於再入院與死亡的風險評估。
ICD-9-CM 及 ICD-10-CM 的 AHRQ 共病症種類一致,共30種。
欲將診斷編碼依照 AHRQ 共病症分類,在 function icdDxToComorbid 的 comorbidMethod 輸入 AHRQ
AHRQ <- icdDxToComorbid(dxDataFile = sampleDxFile, idColName = ID, icdColName = ICD, dateColName = Date, icd10usingDate = "2015-10-01", comorbidMethod = AHRQ) AHRQ$groupedDT[160:164] head(AHRQ$summarised_groupedDT, 5)
3) Charlson
Charlson database 共病症測量指標的是基於 Quan 的 Charlson Comorbidity Index 版本,依診斷編碼其相關的共病症做分類。
ICD-9-CM 及 ICD-10-CM 的 Charlson 共病症種類一致,共17種。
欲將診斷編碼依照 Charlson 共病症分類,在 function icdDxToComorbid 的comorbidMethod 輸入 Charlson
Charlson <- icdDxToComorbid(dxDataFile = sampleDxFile, idColName = ID, icdColName = ICD, dateColName = Date, icd10usingDate = "2015-10-01", comorbidMethod = charlson) Charlson$groupedDT[160:164] head(Charlson$summarised_groupedDT, 5)
4. 自定義診斷碼分組
因應臨床研究需求,本套件採多樣的分組方式以維持診斷醫療大數據分析的彈性,提供進階使用者自行定義診斷分組類別及類別項目,使用者可選擇精確比對或是模糊比對的方式訂定篩選的分組類別及類別項目進行診斷碼的分群。
範例如下,使用者欲定義符合慢性腎臟疾病(Chronic kidney disease)的疾病編碼,相關診斷編碼如下:
- ICD-9:5851, 5852,..., 5859
- ICD-10:N181, N182,..., N189
自定義診斷分組提供兩種分組方法供使用者選擇,將符合條件的診斷編碼分類為心律異常的組別。
1) icdDxToCustom:精確比對
使用者須定義精確分組的表格 (groupingTable),輸入詳細的診斷編碼 (N181, 5853, 5854…),
進行分組的資料須要完全符合,才會歸類為 Cardiac dysrhythmias,另外不符合條件的組別則顯示 NA。
# CustomGroupingTable groupingTable <- data.frame(Group = rep("Chronic kidney disease",6), ICD = c("N181","5853","5854","5855","5856","5859"), stringsAsFactors = FALSE) CustomGroup <- icdDxToCustom(dxDataFile = sampleDxFile, idColName = ID, icdColName = ICD, dateColName = Date, customGroupingTable = groupingTable) CustomGroup$groupedDT[10:14] head(CustomGroup$summarised_groupedDT, 5)
2) icdDxToCustomGrep:模糊比對
使用者須定義模糊分組的表格 (grepTable),以字串比對之正規表示式 (585*\ , N18*\…),當資料開頭為585*\ (ICD-9-CM) 或是N18*\ (ICD-10-CM) 即符合使用者定義之組別條件 Chronic kidney disease,另外不符合條件的組別則顯示 NA。
# CustomGroupingTable grepTable <- data.frame(Group = "Chronic kidney disease", grepIcd = "^585|^N18", stringsAsFactors = FALSE) CustomGrepGroup <- icdDxToCustomGrep(dxDataFile = sampleDxFile, idColName = ID, icdColName = ICD, dateColName = Date, customGroupingTable = grepTable) CustomGrepGroup$groupedDT[10:14] head(CustomGrepGroup$summarised_groupedDT, 5)
II. Data Wrangling
一、篩選符合條件的個案
將醫療大數據診斷資訊進行標準化處理與整合後,將依據使用者所設立的條件篩選符合條件的個案。篩選個案的功能和疾病世代的概念類似,適用於一般疾病(限制搜尋範圍)或慢性病(沒有限制搜尋範圍)的計算。
為選取符合特定條件之診斷資料,目前有三種條件進行篩選:
1) 設立篩選條件
`groupDataType`:設定篩選的分組依據,可用 CCS、PheWAS、comorbidity、自定義組別或 ICD 診斷編碼 `caseCondition`:設定篩選的條件,如:以 CCS 為分組依據,則條件可為特定 CCS 編號 (`isDescription` = FALSE) 或特定 CCS 分類敘述 (`isDescription` = TRUE) ,條件字串為精確比對,使用者亦可輸入正規表達式進行模糊比對
2) 設定搜尋範圍
periodRange:限制每筆診斷資料之間的間隔範圍,預設為間隔30天至365天時間內的資料
3) ICD次數
caseCount:符合篩選方式的最小出現次數,預設至少出現兩次
最後回傳符合條件的個案資料如表四。
表四 選取特定個案診斷紀錄後回傳的資料型態
|ID|Count|FirstCaseDate|EndCaseDate|Period|MostCommonICD|MostCommonICDCount| |--|--|---|---|--|-----|-----| |病患編碼|個數|第一筆資料時間|最後一筆資料時間|第一筆資料與最後一筆資料相隔週期| 出現最多次數的診斷編碼|最常出現診斷編碼的次數 |
符合條件的個案將整合為一筆資料,內容為符合條件的資料總數(count),第一筆診斷資料(first case date)及最後一筆資料時間(end case date)及資料時間相隔週期(period),出現最多次的ICD及次數(most common ICD 和 most common ICD count)。
符合篩選條件會在欄位 selectedCase 顯示 "Selected",使用者也可自行定義組別名稱 (在 caseName 引數項中設定)
Case <- selectCases(dxDataFile = sampleDxFile, idColName = ID, icdColName = ICD, dateColName = Date, groupDataType = ccslvl2, icd10usingDate = "2015/10/01", isDescription = TRUE, caseCondition = "Diseases of the urinary system", caseCount = 1, caseName = "Selected") head(Case)
以上述例子結果之第一筆做說明,個案 A3 符合篩選條件的診斷資料共 5 筆,第一筆診斷日期為 "2008-07-08",最後一筆診斷紀錄為 "2014-02-24"。其中 V420 出現次數最高,共 3 筆。整個診斷紀錄週期為 2057 天。
二、就診紀錄第一筆資料及最後一筆資料
取得病患的就診紀錄第一筆資料及最後一筆資料,可用來做為個案的指標日期供 splitDataByDate 使用。
admissionDate <- getEligiblePeriod(dxDataFile = sampleDxFile, idColName = ID, dateColName = Date) head(admissionDate)
三、以事件(index date)區分事件前後的醫療紀錄
醫學研究常需要觀察特定事件發生前後的改變,因此需要一個指標時間(index date)來記錄事件的發生。timeTag 欄位可顯示此紀錄為事件前或是事件後(Before or After),使用者可依此結果再行篩選事件發生前/後的資料。
指標日期 (indexDateFile) 由使用者定義,可依據個案之醫療紀錄個別定義,如使用 getEligiblePeriod函數所得之結果。
另外本套見也提供週期(window)計算功能,使用者可自行定義週期長度 (gap),預設週期長度為30天。
indexDateTable <- data.frame(ID = c("A0","B0","C0","D0"), indexDate = c("2009-07-25", "2015-12-26", "2015-12-05", "2017-01-29"), stringsAsFactors = FALSE) Data <- splitDataByDate(dxDataFile = sampleDxFile, idColName = ID, icdColName = ICD, dateColName = Date, indexDateFile = indexDateTable, gap = 30) head(Data, 5)
舉例來說,個案 A0 之指標日期為 2009-07-25,則個案 A0 的第一筆診斷資料時間為 2009-07-25 視為指標日期之後發生的紀錄,TimeTag 為 A (after)。兩筆資料間隔天數為 0 天,故為第 1 個週期;個案 A0 的第二筆診斷資料時間為 2010-03-28 ,亦為指標日期後的紀錄,TimeTag 為 A。資料間隔天數為 246 天,為第 9 個週期。
四、 疾病世代
在診斷紀錄整合機制方面,Dr. Ryan 提出疾病世代(Condition era)概念整合處理連續性的醫療行為, 將分散的醫療紀錄整合為單一病程發展紀錄,以利後續醫療大數據分析使用。
疾病世代的概念是針對疾病定義持續期(gap)長度,若任兩個連續診斷之間的間隔小於 gap,則此兩個診斷視為同一個疾病世代。
疾病世代的診斷分類可選擇 ICD 診斷編碼或四種標準化分組,計算病患之疾病世代,其持續期長度預設值為30天(gapDate)。
Era <- getConditionEra(dxDataFile = sampleDxFile, idColName = ID, icdColName = ICD, dateColName = Date, icd10usingDate = "2015-10-01", gapDate = 30, groupDataType = ccs, isDescription = FALSE) head(Era)
以範例的第一個結果做說明:個案 A0 被分組到 CCS 類別 158 共有 5 筆資料,第一筆診斷日期為 "2009-07-25" 、最後一筆診斷紀錄為 "2013-12-20",其中任意兩個連續的診斷,相差間隔皆不小於 30 天,所以有 5 個世代。
五、 探索性資料寬表轉換
診斷碼整合分組後,為能進行後續的探索性資料分析(EDA preparation),本套件提供資料型態轉換的功能,以便轉換為更利於進行統計分析的寬表型態。
寬表提供兩種表示方式,以二分法(binary)或是數量(numeric)的方式呈現(numericOrBinary = N 或 B)。當病患沒有相關的疾病診斷,或診斷次數低於使用者設定之閾值(引數 count,預設為 1),則二分法表示為 FALSE,反之,超過閾值的診斷紀錄數目時,二分法表示為 TRUE;數量表示法則計算該病患在相關疾病的分組上共有幾筆疾病診斷資料。
以下範例以共病症 Elixhauser為範例進行兩種表示法回傳的資料型態。
#binary ELIX <- icdDxToComorbid(dxDataFile = sampleDxFile, idColName = ID, icdColName = ICD, dateColName = Date, icd10usingDate = "2015-10-01", comorbidMethod = elix) groupedData_Wide <- groupedDataLongToWide(dxDataFile = ELIX$groupedDT, idColName = ID, categoryColName = Comorbidity, dateColName = Date, reDup = TRUE, numericOrBinary = B) head(groupedData_Wide, 5)
# numeric groupedData_Wide <- groupedDataLongToWide(dxDataFile = ELIX$groupedDT, idColName = ID, categoryColName = Comorbidity, dateColName = Date, reDup = TRUE, numericOrBinary = N) head(groupedData_Wide, 5)
III. Visualization
第三部份視覺化:套件將前述功能之結果整合,以圖表形式呈現。
一、 Pareto plot:錯誤編碼之柏拉圖圖表
將第一階段- ICD 診斷編碼格式轉換(Code transformation)過程中產生之錯誤編碼訊息列表換成圖表。
以柏拉圖圖表(Pareto plot)的方式呈現,使用者可得知錯誤編碼的種類及出現比例,並依照錯誤數量依序排名。
errorFile:診斷編碼進行統一格式轉換時產生的錯誤編碼- 顯示診斷編碼的版本:顯示全部的編碼版本(
ICDVersion=9,10orall) - 顯示錯誤診斷編碼的種類:顯示全部的錯誤編碼種類(
wrongICDType=version,formatorall) - 預設為顯示前 10 名的錯誤編碼(
TopN=10) - 第 10 名以後的錯誤編碼設為"others"(
Others=TRUE)
如欲觀察前十名的錯誤編碼種類及分布比例,則範例如下:
error <- icdDxDecimalToShort(dxDataFile = sampleDxFile, icdColName = ICD, dateColName = Date, icd10usingDate = "2015/10/01") Plot_error1 <- plotICDError(errorFile = error$Error, icdVersion = all, wrongICDType = all, others = TRUE)
 上圖顯示前10名錯誤的診斷編碼,其中第一名的錯誤A0.11共有20筆,佔全部錯誤18.35% (20/109),其餘名次的錯誤編碼則分類至 "Others"。
上圖顯示前10名錯誤的診斷編碼,其中第一名的錯誤A0.11共有20筆,佔全部錯誤18.35% (20/109),其餘名次的錯誤編碼則分類至 "Others"。
Plot_error1$ICD
另外使用者可選擇將 ICD-9 依照章節分組 (groupICD = TRUE);僅 ICD9 有此功能, ICD10 本身已有前綴之字母編號故無須額外編組。
ICD-9 共 19 章節疾病分類:
001-139:傳染病或寄生蟲相關疾病
140-239:腫瘤相關疾病
...等。
plotICDError依照編碼開頭分類,大致分為 12 組:0, 1, 2,..., 9, V 和 E
如欲觀察 ICD9 的前三名錯誤編碼種類及分布比例,則範例如下。
Plot_error2 <- plotICDError(errorFile = error$Error, icdVersion = 9, wrongICDType = all, groupICD = TRUE, others = TRUE, topN = 3)

icdVersion設為 9,緊顯示 ICD9 版本之錯誤編碼,並設 groupICD 為 TRUE,使 ICD9 依編碼開頭分組,
顯示前三名的錯誤編碼
第3名以後的錯誤編碼設為"others"
由下表格得知第一名的錯誤編碼為A01.05佔組別A共61.54%,總共有13筆錯誤,佔全部的錯誤的比例為41.94%
Plot_error2$ICD #> ICDGroup groupCount CumCountPerc MostICDInGroup ICDPercInGroup WrongType #> 1: A 13 41.94% A01.05 61.54% Wrong version #> 2: 7 9 70.97% 75.52 44.44% Wrong format #> 3: 0 5 87.1% 001 100% Wrong format #> 4: Others 4 100% E03.0 100% Wrong version
二、 histogram plot:標準化分組之長條圖表
將第二階段-探索性資料轉換之寬表資料圖形化,以便觀察診斷資料在各分群(如 comorbidity)的分布情形及數量。
使用者可選擇顯示前幾名的分組資料(Top N),且可限制每個分群的病患至少有一定比例(limit Frequency)。
- 預設為顯示前 10 名的錯誤編碼(
TopN=10) - 限制最低比例預設為 1% (
limitFreq=0.01);假設使用者的資料共 1000 個病患,則每種組別至少要有 10 個病患
範例使用步驟如下:
ELIX <- icdDxToComorbid(dxDataFile = sampleDxFile, idColName = ID, icdColName = ICD, dateColName = Date, icd10usingDate = "2015-10-01", comorbidMethod = elix) groupedDataWide <- groupedDataLongToWide(dxDataFile = ELIX$groupedDT, idColName = ID, categoryColName = Comorbidity, dateColName = Date, reDup = TRUE, numericOrBinary = B) plot1 <- plotDiagCat(groupedDataWide = groupedDataWide, idColName = ID, topN = 10, limitFreq = 0.01)

上述程式碼先將資料以共病症 Elixhauser 做標準化分組並轉成寬表,最後製圖:顯示前 10 名、至少 1% 比例的組別。
下表格為符合條件設定的共病症前十名,每組分別有多少人,且該共病症在資料中的比例。 本範例共有 38 個病患,以第一組為例,共有 24 人被分為 RENLFAIL 共病症,比例為 63.16% (24/38)
plot1$sigCate
若資料有進行病患分組 Case/Control,於每個類別(如comorbidity)將進行兩組(Case and Control)的卡方獨立性檢定,圖表將顯示達統計上顯著差異的分組類別。
若樣本數過小以致不適用卡方檢定時,則採用費雪爾精確檢定(Fisher’s Exact Test)
顯著差異 p-value 預設門檻為 0.05,預設顯示前 10 名的類別,且每群種類的病患在兩組的比率至少有一組達 1%
舉例來說,假設 Case 和 Control 組各 500 人,在某特定組別至少有一組的人數有 5 個病患 (1%)。
範例如下:
selectedCaseFile <- selectCases(dxDataFile = sampleDxFile, idColName = ID, icdColName = ICD, dateColName = Date, icd10usingDate = "2015/10/01", groupDataType = ccslvl2, caseCondition = "Diseases of the urinary system", caseCount = 1) ELIX <- icdDxToComorbid(dxDataFile = sampleDxFile, idColName = ID, icdColName = ICD, dateColName = Date, icd10usingDate = "2015-10-01", comorbidMethod = elix) groupedData_Wide <- groupedDataLongToWide(dxDataFile = ELIX$groupedDT, idColName = ID, categoryColName = Comorbidity, dateColName = Date, reDup = TRUE, selectedCaseFile = selectedCaseFile) plot2 <- plotDiagCat(groupedDataWide = groupedData_Wide, idColName = ID, groupColName = selectedCase, topN = 10, limitFreq = 0.01, pvalue = 0.05)

上述範例之步驟為:
- 定義 case/control 組,採 CCS 多階層的標準化分組,在 CCS level 2 被分組為 "Diseases of the urinary system" 者視為 case 組,詳細設定條件請參照函式
selectCases之範例。 - 將資料以共病症 Elixhauser 做標準化分組並轉成寬表
- 設定作圖標準:顯示前幾名組別、限制比例、p 值(p-value)。
下表格為在 selected / nonSelected 兩組間達顯著差異的共病症組別 RENLFAIL
在 selected 組佔了 100% (24/24),在 nonSelected 組佔了0% (0/14)
plot2$sigCate
Reference
I. Code tramsformation
ICD-9-CM code (2014): https://www.cms.gov/Medicare/Coding/ICD9ProviderDiagnosticCodes/codes.html
ICD-10-CM code (2019): https://www.cms.gov/Medicare/Coding/ICD10/2019-ICD-10-CM.html
https://www.cms.gov/Medicare/Coding/ICD10/2019-ICD-10-CM.html
https://www.findacode.com/search/search.php
https://www.cms.gov/Medicare/Quality-Initiatives-Patient-Assessment-Instruments/HospitalQualityInits/Downloads/HospitalAppendix_F.pdf
II. Code grouping
本套件使用之CCS版本依照HCUP提供之最新版本進行年度的更新,目前ICD-9-CM與ICD-10-CM的CCS定義版本分別為2015年及2019年版。
PheWAS依照PheWAS Resources網站 進行更新,目前版本為第二版(2015年)。
Elixhauser共病症依照HCUP提供之版本進行更新,目前ICD-9-CM與ICD-10的Elixhauser共病症定義版本分別為2015年及2019年版。
CCS (Clinical Classifications Software)
ICD-9-CM (2015): https://www.hcup-us.ahrq.gov/toolssoftware/ccs/Single_Level_CCS_2015.zip
https://www.hcup-us.ahrq.gov/toolssoftware/ccs/Multi_Level_CCS_2015.zip
ICD-10-CM (2019): https://www.hcup-us.ahrq.gov/toolssoftware/ccs10/ccs_dx_icd10cm_2019_1.zip
PheWAS
ICD-9-Phecode (version 1.2, 2015): https://phewascatalog.org/phecodes
ICD-10 Phecode (version 1.2 beta, 2019): https://phewascatalog.org/phecodes_icd10cm
Comorbidities
ICD-9-AHRQ (2012-2015): https://www.hcup-us.ahrq.gov/toolssoftware/comorbidity/comorbidity.jsp#references
ICD-10-AHRQ (2019): https://www.hcup-us.ahrq.gov/toolssoftware/comorbidityicd10/comformat_icd10cm_2019_1.txt
ICD-9-Charlson: http://mchp-appserv.cpe.umanitoba.ca/Upload/SAS/ICD9_E_Charlson.sas.txt
ICD-10-Charlson: http://mchp-appserv.cpe.umanitoba.ca/Upload/SAS/ICD10_Charlson.sas.txt
ICD-9-Elixhauser (2012-2015): https://www.hcup-us.ahrq.gov/toolssoftware/comorbidity/comorbidity.jsp#references
ICD-10-Elixhauser (2019): https://www.hcup-us.ahrq.gov/toolssoftware/comorbidityicd10/comformat_icd10cm_2019_1.txt
Add the following code to your website.
For more information on customizing the embed code, read Embedding Snippets.
