In paithiov909/gibasa: An Alternative 'Rcpp' Wrapper of 'MeCab'
knitr::opts_chunk$set(
tidy = "styler",
collapse = TRUE,
comment = "#>"
)
gibasaパッケージについて
gibasaは、RからMeCabを利用して形態素解析をおこなうためのパッケージです。
モチベーションとしては、tidytext::unnest_tokensを意識した処理をMeCabを利用しつつできるようにしたいということで開発しています。また、gibasaはv0.5.0からMeCabのソースコードをソースパッケージ内に含んでいるため、ビルドするのにMeCabのバイナリファイルを必要としないという長所があります。
インストールの仕方
CRANのほか、r-universeからもインストールできます。
# Install gibasa from r-universe repository
install.packages("gibasa", repos = c("https://paithiov909.r-universe.dev", "https://cloud.r-project.org"))
バイナリパッケージが用意されていない環境では、ソースパッケージをビルドしてインストールします。ビルド時にMECAB_DEFAULT_RCという環境変数を内部的に指定するため、正しく動作させるにはmecabrcファイルとMeCabの辞書があらかじめ適切な位置に配置されている必要があります。使っているOSのパッケージマネージャなどからインストールしておいてください。
# Sys.setenv(MECAB_DEFAULT_RC = "/fullpath/to/your/mecabrc") # if necessary
remotes::install_github("paithiov909/gibasa")
Windowsの場合、ソースビルドにはRtoolsが必要です。また、インストールの方法によらず、gibasaを使用するにはMeCabの辞書が必要なため、これなどを使ってMeCabをインストールしておいてください。
なお、v0.9.4から、オリジナルのMeCabと同様にmecabrcファイルを参照できるようになったため、辞書とmecabrcファイルが適切に配置されていれば、MeCabのバイナリががなくても形態素解析が可能になっています。mecabrcファイルは、環境変数MECABRCで指定されたファイルか、~/.mecabrcというファイルが参照されます。
たとえば、IPA辞書(ipadic)をPyPIからダウンロードして利用するには、ターミナルから次のコマンドを実行します。
$ python3 -m pip install ipadic
$ python3 -c "import ipadic; print('dicdir=' + ipadic.DICDIR);" > ~/.mecabrc
また、gibasaはMeCabのシステム辞書をビルドする機能もラップしているため、辞書のソースファイルを用意できれば、パッケージのインストール後に辞書を配置することによっても使用可能になります。たとえば、kelpbedsというパッケージを利用してIPA辞書を配置して使えるようにするには、次のようにします。
install.packages("kelpbeds", repos = c("https://paithiov909.r-universe.dev", "https://cran.r-project.org"))
dic_dir <- fs::dir_create(file.path(Sys.getenv("HOME"), "ipadic-utf8"))
kelpbeds::prep_ipadic(dic_dir)
gibasa::build_sys_dic(
dic_dir = dic_dir,
out_dir = dic_dir,
encoding = "utf8"
)
readr::write_lines(
paste0("dicdir=", dic_dir),
file.path(Sys.getenv("HOME"), ".mecabrc")
)
基本的な使い方
gibasaは、次にあげる関数を使って、CJKテキストの分かち書きをすることができるというパッケージです。
gibasa::tokenizegibasa::prettifygibasa::pack
まず、doc_id列とtext列をもつデータフレームについて、gibasa::tokenizeでtidy textのかたちにできます(以下の例ではIPA辞書を使っています)。ちなみに、元のデータフレームのdoc_id列とtext列以外の列は戻り値にも保持されます。
gibasa::ginga[5]
dat <- data.frame(
doc_id = seq_along(gibasa::ginga[5:8]),
text = gibasa::ginga[5:8],
meta = c("aaa", "bbb", "ccc", "ddd")
)
res <- gibasa::tokenize(dat, text, doc_id)
head(res)
gibasa::prettifyでfeature列の素性情報をパースして分割できます。このとき、col_select引数でパースしたい列を指定すると、それらの列だけをパースすることができます。
head(gibasa::prettify(res))
head(gibasa::prettify(res, col_select = 1:3))
head(gibasa::prettify(res, col_select = c(1, 3, 5)))
head(gibasa::prettify(res, col_select = c("POS1", "Original")))
gibasa::packを使うと、pull引数で指定した列について、いわゆる「分かち書き」にすることができます。デフォルトではtoken列について分かち書きにします。
res <- gibasa::prettify(res)
gibasa::pack(res)
gibasa::pack(res, POS1)
詳しい使い方
より詳しい使い方については、次のサイトを参照してください。
ベンチマーク
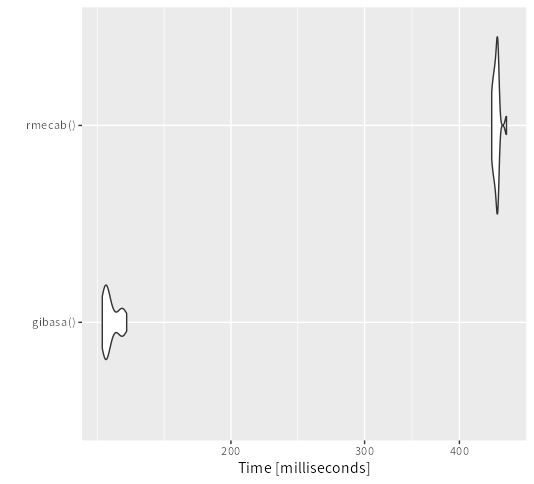
TokyoR #98でのLTのスライドでも紹介しましたが、ある程度の分量がある文字列ベクトルに対して、ごくふつうに形態素解析だけをやるかぎりにおいては、RMeCabよりもgibasaのほうが解析速度が速いと思います(というより、RMeCabでもMeCabを呼んでいる部分はおそらく十分速いのですが、欲しいかたちに加工するためのRの処理に時間がかかることが多いです)。
dat <- data.frame(
doc_id = seq_along(gibasa::ginga),
text = gibasa::ginga
)
gibasa <- function() {
gibasa::tokenize(dat) |>
gibasa::prettify(col_select = "POS1") |>
dplyr::mutate(doc_id = as.integer(doc_id)) |>
dplyr::select(doc_id, token, POS1) |>
as.data.frame()
}
rmecab <- function() {
purrr::imap_dfr(
RMeCab::RMeCabDF(dat, 2),
~ data.frame(doc_id = .y, token = unname(.x), POS1 = names(.x))
)
}
bench <- microbenchmark::microbenchmark(gibasa(),
rmecab(),
times = 20L,
check = "equal")
ggplot2::autoplot(bench)
paithiov909/gibasa documentation built on June 14, 2025, 4:31 p.m.
knitr::opts_chunk$set( tidy = "styler", collapse = TRUE, comment = "#>" )
gibasaパッケージについて
gibasaは、RからMeCabを利用して形態素解析をおこなうためのパッケージです。
モチベーションとしては、tidytext::unnest_tokensを意識した処理をMeCabを利用しつつできるようにしたいということで開発しています。また、gibasaはv0.5.0からMeCabのソースコードをソースパッケージ内に含んでいるため、ビルドするのにMeCabのバイナリファイルを必要としないという長所があります。
インストールの仕方
CRANのほか、r-universeからもインストールできます。
# Install gibasa from r-universe repository install.packages("gibasa", repos = c("https://paithiov909.r-universe.dev", "https://cloud.r-project.org"))
バイナリパッケージが用意されていない環境では、ソースパッケージをビルドしてインストールします。ビルド時にMECAB_DEFAULT_RCという環境変数を内部的に指定するため、正しく動作させるにはmecabrcファイルとMeCabの辞書があらかじめ適切な位置に配置されている必要があります。使っているOSのパッケージマネージャなどからインストールしておいてください。
# Sys.setenv(MECAB_DEFAULT_RC = "/fullpath/to/your/mecabrc") # if necessary remotes::install_github("paithiov909/gibasa")
Windowsの場合、ソースビルドにはRtoolsが必要です。また、インストールの方法によらず、gibasaを使用するにはMeCabの辞書が必要なため、これなどを使ってMeCabをインストールしておいてください。
なお、v0.9.4から、オリジナルのMeCabと同様にmecabrcファイルを参照できるようになったため、辞書とmecabrcファイルが適切に配置されていれば、MeCabのバイナリががなくても形態素解析が可能になっています。mecabrcファイルは、環境変数MECABRCで指定されたファイルか、~/.mecabrcというファイルが参照されます。
たとえば、IPA辞書(ipadic)をPyPIからダウンロードして利用するには、ターミナルから次のコマンドを実行します。
$ python3 -m pip install ipadic
$ python3 -c "import ipadic; print('dicdir=' + ipadic.DICDIR);" > ~/.mecabrc
また、gibasaはMeCabのシステム辞書をビルドする機能もラップしているため、辞書のソースファイルを用意できれば、パッケージのインストール後に辞書を配置することによっても使用可能になります。たとえば、kelpbedsというパッケージを利用してIPA辞書を配置して使えるようにするには、次のようにします。
install.packages("kelpbeds", repos = c("https://paithiov909.r-universe.dev", "https://cran.r-project.org")) dic_dir <- fs::dir_create(file.path(Sys.getenv("HOME"), "ipadic-utf8")) kelpbeds::prep_ipadic(dic_dir) gibasa::build_sys_dic( dic_dir = dic_dir, out_dir = dic_dir, encoding = "utf8" ) readr::write_lines( paste0("dicdir=", dic_dir), file.path(Sys.getenv("HOME"), ".mecabrc") )
基本的な使い方
gibasaは、次にあげる関数を使って、CJKテキストの分かち書きをすることができるというパッケージです。
gibasa::tokenizegibasa::prettifygibasa::pack
まず、doc_id列とtext列をもつデータフレームについて、gibasa::tokenizeでtidy textのかたちにできます(以下の例ではIPA辞書を使っています)。ちなみに、元のデータフレームのdoc_id列とtext列以外の列は戻り値にも保持されます。
gibasa::ginga[5] dat <- data.frame( doc_id = seq_along(gibasa::ginga[5:8]), text = gibasa::ginga[5:8], meta = c("aaa", "bbb", "ccc", "ddd") ) res <- gibasa::tokenize(dat, text, doc_id) head(res)
gibasa::prettifyでfeature列の素性情報をパースして分割できます。このとき、col_select引数でパースしたい列を指定すると、それらの列だけをパースすることができます。
head(gibasa::prettify(res)) head(gibasa::prettify(res, col_select = 1:3)) head(gibasa::prettify(res, col_select = c(1, 3, 5))) head(gibasa::prettify(res, col_select = c("POS1", "Original")))
gibasa::packを使うと、pull引数で指定した列について、いわゆる「分かち書き」にすることができます。デフォルトではtoken列について分かち書きにします。
res <- gibasa::prettify(res) gibasa::pack(res) gibasa::pack(res, POS1)
詳しい使い方
より詳しい使い方については、次のサイトを参照してください。
ベンチマーク
TokyoR #98でのLTのスライドでも紹介しましたが、ある程度の分量がある文字列ベクトルに対して、ごくふつうに形態素解析だけをやるかぎりにおいては、RMeCabよりもgibasaのほうが解析速度が速いと思います(というより、RMeCabでもMeCabを呼んでいる部分はおそらく十分速いのですが、欲しいかたちに加工するためのRの処理に時間がかかることが多いです)。
dat <- data.frame( doc_id = seq_along(gibasa::ginga), text = gibasa::ginga ) gibasa <- function() { gibasa::tokenize(dat) |> gibasa::prettify(col_select = "POS1") |> dplyr::mutate(doc_id = as.integer(doc_id)) |> dplyr::select(doc_id, token, POS1) |> as.data.frame() } rmecab <- function() { purrr::imap_dfr( RMeCab::RMeCabDF(dat, 2), ~ data.frame(doc_id = .y, token = unname(.x), POS1 = names(.x)) ) } bench <- microbenchmark::microbenchmark(gibasa(), rmecab(), times = 20L, check = "equal") ggplot2::autoplot(bench)

Add the following code to your website.
For more information on customizing the embed code, read Embedding Snippets.
