In martscht/PsyMSc1: Übungen für die Seminare im Sommersemester 2020
library(learnr) # package to generate interactive HTML
library(gradethis) # grade Code from input in HTML
library(shiny) # nice appearance in R
library(fontawesome) # nice fonts
library(psych) # EFA durchführen
library(lavaan)
library(semPlot)
knitr::opts_chunk$set(exercise.checker = gradethis::grade_learnr)
source('startup.R')
Einleitung zu Pfadanalysen und Strukturgleichungsmodellen (SEM)
Pfadanalysen sind im Grunde genommen mehrere Regressionsanalysen, welche simultan geschätzt werden können. So werden auch mehrere Abhängigkeiten zwischen Variablen berücksichtigt. Strukturgleichungsmodelle kombinieren Pfadanalysen mit Messmodellen. Wir könnten also sagen: "SEM = CFA + Pfadanalyse"!
In dieser Sitzung erweitern wir unsere Kenntnisse mit dem R-Paket lavaan um gerichtete Abhängigkeiten. Möchten Sie die Grundlagen im Umgang damit wiederholen, so empfiehlt es sich die erste Sitzung nochmals anzusehen (PsyMSc1::Sitzung_1()). Auch baut diese Sitzung auf der vergangenen Sitzung zur CFA auf (PsyMSc1::Sitzung_3()).
Bevor wir mit den Analysen beginnen können, laden wir zunächst alle Pakete, welche wir im Folgenden benötigen werden.
library(lavaan)
library(semPlot) # grafische Darstellung von Pfadanalyse- und Strukturgleichungsmodellen
Den gesamten R-Code, der in dieser Sitzung genutzt wird, können Sie r fontawesome::fa("download") hier herunterladen.
Datensatz
Der Datensatz StressAtWork, den wir im Folgenden untersuchen wollen, ist eine Zusammenstellung aus mehreren Studien der Arbeits- und Organisationspsychologie- Abteilung der Goethe-Universität, in welchen Call-Center-Mitarbeiter untersucht wurden. Wir können diesen wie gewohnt laden:
data("StressAtWork", package = "PsyMSc1")
Der Datensatz enthält das Geschlecht der Probanden (sex, 1=weiblich, 2=männlich) sowie ausgewählte Messungen der Variablen Zeitdruck (zd1, zd2 und zd6) und Arbeitsorganisationale Probleme (aop3, aop4 und aop8) aus dem Instrument zur stressbezogenen Tätigkeitsanalyse (ISTA) von Semmer, Zapf und Dunckel (1999), Psychosomatische Beschwerden (auch Befindlichkeit: bf1,...,bf20) aus der Psychosomatischen Beschwerdenliste von Mohr (1986) sowie Messungen zu Subskalen von Burnout: Emotionale Erschöpfung (bo1, bo6, b12 und b19) und Leistungserfüllung (bo7, bo8 und bo21) aus Maslachs Burnout-Inventar (Maslach & Jackson, 1986) in der deutschen Übersetzung von Büssing und Perrar (1992).
head(StressAtWork)
names(StressAtWork)
Skalenbeschreibungen und Beispielitems
Unter Zeitdruck verstehen wir hier zusätzliche Zeiteinschränkungen, die dazu führen, dass mehr Energie nötig ist, um eine Handlung durchzuführen. Gleichzeitig kann dies auch dazu führen, dass eine Handlung nicht mehr oder nicht mehr rechtzeitig durchführbar ist. Ein Beispielitem ist: "Wie häufig passiert es, dass Sie schneller arbeiten, als sie es normalerweise tun, um die Arbeit zu schaffen?" Die Items wurden auf einer 5-Punkt-Likert Skala beantwortet (Semmer, et al., 1999).
Arbeitsorganisationale Probleme hindern hier bei der Durchführung einer Handlung durch zum Beispiel veraltete Informationen, schlechte Arbeitsgeräte oder organisationale Probleme, die zu einem Mehraufwand führen. Ein Beispielitem ist: "A kann die Arbeitsaufträge gut erledigen, wenn er/sie sich an die vom Betrieb vorgesehenen Wege hält. B kann die Arbeitsaufträge nur bewältigen, wenn er/sie von den vom Betrieb vorgesehenen Wegen abweicht. Welcher der beiden Arbeitsplätze ist Ihrem am ähnlichsten?" Die Items wurden auf einer 5-Punkt-Likert-Skala beantwortet (Semmer, et al., 1999).
Psychosomatische Beschwerden beschreiben körperliche Befindlichenkeiten, die mögliche Langzeitfolgen von Stress sind, wie etwa Schlaflosigkeit, Kopfschmerzen oder Nervosität. Im Gegensatz zu allen anderen Skalen, die hier verwendet werden, handelt es sich bei den psychosomatischen Beschwerden um einen Index, welchem ein formatives Messmodell zu Grunde (im Gegensatz zu reflexiven Messmodellen) liegt (siehe dazu Abschnitt reflexive vs. formative Messmodelle). Ein Beispielitem ist: "Ermüden Sie schnell?" Die Items wurden auf einer 5-Punkt-Likert-Skala beantwortet (Mohr, 1986).
Emotionale Erschöpfung ist hier das Gefühl, erschöpft, niedergeschlagen und frustriert durch die Arbeit zu sein und die Zusammenarbeit mit anderen als besonders anstrengend anzusehen. Ein Beispielitem ist: "Am Ende eines Arbeitstages fühle ich mich verbraucht." Die Items wurden auf einer 7-Punkt-Likert-Skala beantwortet (Maslach & Jackson, 1986).
Wir verstehen unter Leistungserfüllung energetische Gefühle, Dinge anzupacken und das Gefühl, die Möglichkeit zu haben, die eigenen Ambitionen zu erfüllen. Ein Beispielitem ist: "Ich fühle mich sehr tatkräftig." Die Items wurden auf einer 7-Punkt-Likert-Skala beantwortet (Maslach & Jackson, 1986).
Theoretische Grundlage {#Hypothesen}
Pfadanalysen und Strukturgleichungsmodelle gehören, wie auch die CFA, zu den konfirmatorischen, also Theorie bestätigenden, Verfahren und sollen ganz im Popper’schen Sinn durch Vergleiche der Daten mit theoretischen Modellen wissenschaftliche Erkenntnis gewinnen. Wir haben in unseren Daten drei Arten von Variablen: Zeitdruck und Arbeitsorganisationale Probleme sind Stressoren und sollten also mit einer gewissen Wahrscheinlichkeit in einem Individuum zu einer Stressreaktion führen. Emotionale Erschöpfung ist eine Facette von Burnout und gehört somit zu den kurzfristigeren Stressfolgen. Psychosomatische Beschwerden treten unter anderem auf, wenn Stress über einen langen Zeitraum auf ein Individuum einwirkt. Somit können wir postulieren, dass Stress über emotionale Erschöpfung auf psychosomatische Beschwerden wirkt und somit nur einen indirekten Effekt auf die psychosomatischen Beschwerden hat:
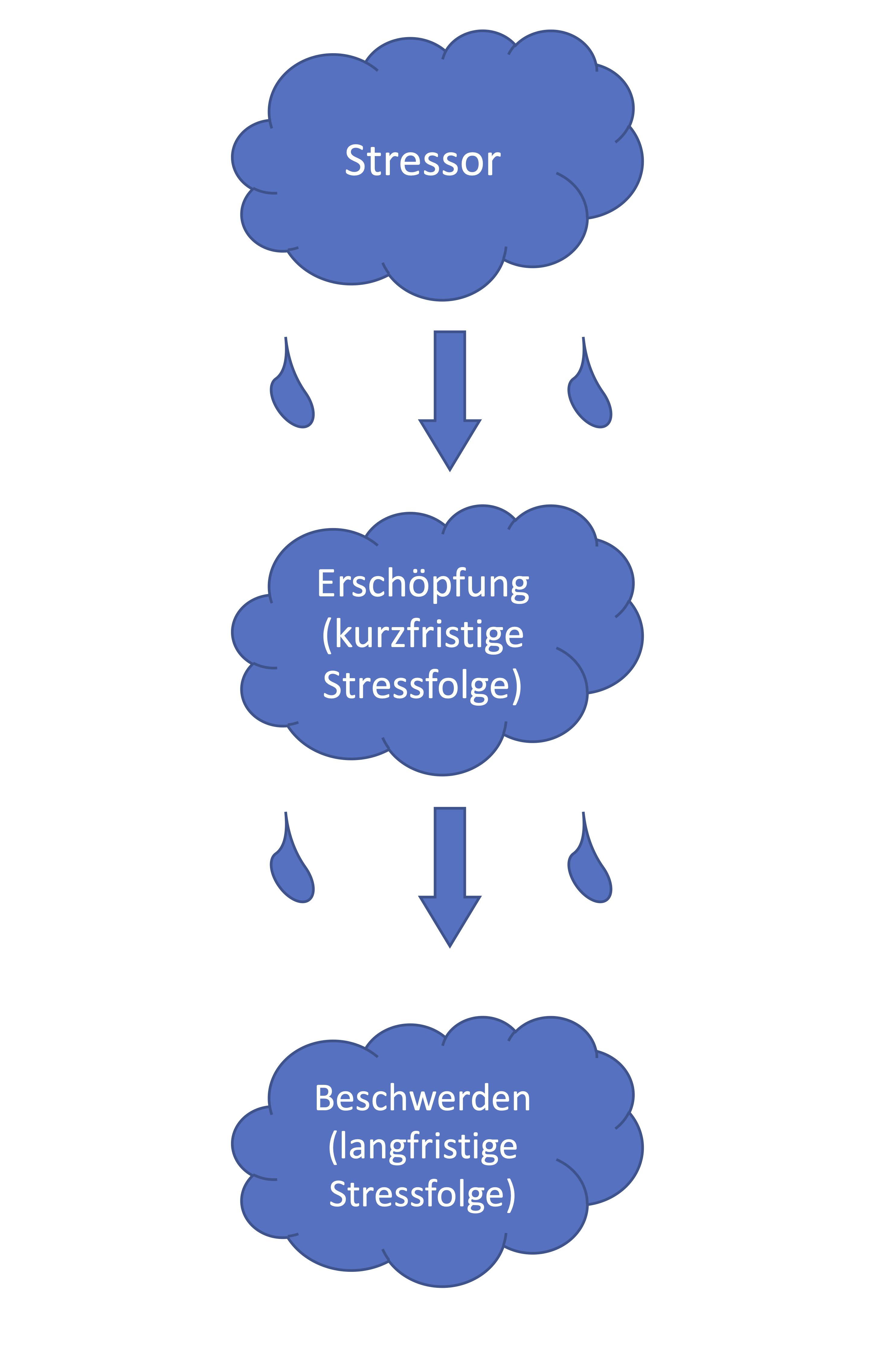
Das hier dargestellte schematische Modell postuliert also eine sogenannte vollständige Mediation (dazu mehr im nächsten Abschnitt zur Pfadanalyse) vom Stressor über die kurzfristige (Erschöpfung) auf die langfristigen (Beschwerden) Stressfolgen.
Der Einfachheit halber wollen wir diese Hypothesen zunächst nur mit drei Variablen untersuchen: Zeitdruck, emotionale Erschöpfung und psychosomatische Beschwerden.
Pfadanalyse {#Pfadanalyse}
Wir möchten die Hypothesen aus der letzten Sektion zunächst mit Skalenmittelwerten und einer Pfadanalyse untersuchen. Wir wollen außerdem den indirekten Effekt quantifizieren und inferenzstatistisch untersuchen. Dazu müssen wir pro Proband einen Skalenmittelwert pro Variable berechnen. Dazu verwenden wir die bereits in der ersten Sitzung kennengelernte Funktion rowMeans und berechnen so den Mittelwert der psychosomatischen Beschwerden als BFs, den Zeitdruck als ZDs und die emotionale Erschöpfung als BOEEs; das kleine "s" hängen wir an die Skalennamen dran, um zu signialisieren, dass es sich hier um manifeste Skalenmittelwerte handelt:
StressAtWork$ZDs <- rowMeans(StressAtWork[,paste0("zd",c(1, 2, 6))])
StressAtWork$BOEEs <- rowMeans(StressAtWork[,paste0("bo",c(1, 6, 12, 19))])
StressAtWork$BFs <- rowMeans(StressAtWork[,paste0("bf",1:20)])
Hierbei hilft uns paste0 die Schreibweise abzukürzen: wir müssen nicht alle Itemnamen einzeln tippen, sondern die "Strings" werden automatisch erzeugt. Um dies genauer zu verstehen, könnten wir bspw. paste0("zd",c(1, 2, 6)) einmal ausführen. Dies kommt zum gleichen Ergebnis, wie wenn wir c("zd1", "zd2", "zd6") getippt hätten. Das Modell wird nun, ähnlich dem Regressionsmodell aus der ersten Sitzung, aufgestellt. In lavaan werden gerichtete Beziehung zwischen Variablen mit ~ dargestellt, wobei links der Tilde die abhängige Variable (das Kriterium) und rechts der Tilde die unabhängige Variable (der Prädiktor) steht. Für unser angenommenes Modell gibt es folgende Beziehung: Zeitdruck wirkt auf emotionale Erschöpfung und auf psychosomatische Beschwerden. Emotionale Erschöpfung wirkt auf psychosomatische Beschwerden. In diesem Modell wird Zeitdruck die unabhängige Variable genannt, emotionale Erschöpfung ist der Mediator, der die Beziehung der unabhängigen auf die abhängige Variable psychosomatische Beschwerden mediiert. Hier hat Zeitdruck eine direkte Beziehung mit emotionaler Erschöpfung. Emotionale Erschöpfung hat eine direkte Beziehung mit psychosomatischen Beschwerden und Zeitdruck hat eine direkte und eine indirekte (über emotionale Erschöpfung) Beziehung mit psychosomatischen Beschwerden.
Es muss folglich zwei Gleichungen geben: in einer Gleichung ist BOEEs die abhängige Variable und wird durch ZDs vorhergesagt. In der zweiten Gleichung ist BFs die abhängige Variable und wird durch BOEEs und ZDs vorhergesagt. Nun hängt es außerdem von der Schätzfunktion ab, welche weiteren Beziehungen wir in unserem Modell spezifizieren müssen. Wir wollen die sem Funktion verwenden, um das Modell zu schätzen: sem ist, wie cfa (diese kennen Sie aus PsyMSc1::Sitzung_3(), um CFAs zu schätzen) auch eine "Convenience"-Funktion, die gewisse Voreinstellungen verwendet und diese an die lavaan-Funktion, die Sie in der ersten Sitzung kennengelernt hatten, übergibt. Sie hatten damals eine Regression mit lavaan geschätzt und mussten dabei bspw. mit Y ~ 1 + X die Regression anfodern, mit welcher durch X die abhängige Variable Y vorhergesagt wurde. Sie mussten hierbei explizit das Interzept (also den durch X bedingten Mittelwert von Y) sowie die Residualvarianz von Y anfordern via Y ~~ Y (für eine Wiederholung schauen Sie gerne in PsyMSc1::Sitzung_1() vorbei). Wie auch die cfa-Funktion übernimmt die sem Funktion einige dieser Einstellungen für uns. So müssen wir bspw. Residualvarianzen nicht explizit anfragen. Die Mittelwertsstruktur wird in sem per Default nicht mitmodelliert. Wenn wir Mittelwerte betrachten wollen, können wir allerdings der sem-Funktion die Zusatzeinstellung meanstructure = TRUE übergeben. Die Default-Einstellungen für Messmodelle sind identisch mit jenen der Funktion cfa. Wir wiederholen diese, wenn es soweit ist. Somit landen wir bei dieser sehr effizienten Schreibweise für unser Modell:
model_paths <- '
BOEEs ~ ZDs
BFs ~ BOEEs + ZDs
'
Hätten wir dieses Modell mit lavaan schätzen wollen, so hätten wir folgendes formulieren müssen (wir wollen die Mittelwertsstruktur auch hier ignorieren und lassen daher ~1 weg!):
model_paths_lavaan <- '
BOEEs ~ ZDs
BFs ~ BOEEs + ZDs
BOEEs ~~ BOEEs
BFs ~~ BFs
'
Wir können dieses Modell nun schätzen, indem wir die Funktion sem verwenden und ihr unser Modell sowie die Daten übergeben.
Mit summary erhalten wir detaillierte Informationen über Modellfit, Parameterschätzungen und deren Signifikanz.
fit_paths <- sem(model_paths, data = StressAtWork)
summary(fit_paths, rsq = T, fit.measures = T)
lavaan::summary(fit_paths, rsq = T, fit.measures = T)
## Skalen bilden
StressAtWork$ZDs <- rowMeans(StressAtWork[,paste0("zd",c(1, 2, 6))])
StressAtWork$BOEEs <- rowMeans(StressAtWork[,paste0("bo",c(1, 6, 12, 19))])
StressAtWork$BFs <- rowMeans(StressAtWork[,paste0("bf",1:20)])
## Modell spezifizieren
model_paths <- '
BOEEs ~ ZDs
BFs ~ BOEEs + ZDs
'
## Modell fitten
fit_paths <- sem(model_paths, data = StressAtWork)
An
abbrev(X = fit_paths, begin = "Model Test User Model", end = "Model Test Baseline Model", fit.measures = T)
erkennen wir, dass es sich hier um das saturierte Modell handelt. Die Korrelationen zwischen den Skalenmittelwerten sind also nur retransformiert, um unser Modell abzubilden. Ein Modellfit-Test ist nicht möglich.
abbrev(X = fit_paths, begin = "User Model versus Baseline Model", end = "Parameter Estimates", fit.measures = T)
Alle Fit-Indizes zeigen perfekten Fit an (CFI = 1, TLI = 1, RMSEA = 0, SRMR = 0).
abbrev(X = fit_paths, begin = "Regressions", end = "Variances", fit.measures = T)
Im Gegensatz zur CFA wird uns nun ein Block in der Summary gezeigt, welcher die Regressionskoeffizienten unseres Modells enthält (ohne Interzept). Hier ist zu erkennen, dass alle Koeffizienten auf dem 5% Niveau signifikant von 0 verschieden sind. Es kann sich also maximal um eine partielle Mediation handeln, da die direkte Beziehung zwischen Zeitdruck und psychsomatischen Beschwerden laut dieser Signifikanzentscheidung mit einer Irrtumswahrscheinlichkeit von 5% auch in der Population besteht und somit der Effekt von Zeitdruck auf die Psychosomatischen Beschwerden nicht vollständig über die emotionale Erschöpfung mediiert wird.
Von einer vollständigen Mediation würden wir sprechen, wenn die direkte Beziehung zwischen Zeitdruck und psychosomatischen Beschwerden (also zwischen unabhängiger und abhängiger Variable) nicht bedeutsam von 0 verschieden ist und der indirekte Effekt von Zeitdruck über emotionale Erschöpfung auf psychsomatische Beschwerden signifikant von Null verschieden ist (dies war unsere Hypothese, die wir im vorherigen Abschnitt postuliert hatten). Für die Population würden wir also bei einer vollständigen Mediation davon ausgehen, dass die unabhängige Variable nur über den Mediator mit der abhängigen Variable zusammenhängt, also jegliche Veränderung in der unabhängigen Variable mit Veränderungen im Mediator zusammenhängt und durch diese Beziehung auch mit der abhängigen Variable kovariiert.
abbrev(X = fit_paths, begin = "R-Square", end = NULL, shift = 1, rsq = T)
Insgsamt ist die Vorhersage der emotionalen Erschöpfung und der psychosomatischen Beschwerden zwar statistisch signifikant, allerdings werden "nur" ca. r round(inspect(fit_paths, "r2")[1], 4)*100% der Variation von BOEEs erklärt sowie ca. r round(inspect(fit_paths, "r2")[2], 4)*100% der Variation von
BFs.
Im nächsten Abschnitt wollen wir unser Modell grafisch veranschaulichen. In der darauf folgenden Sektion wollen wir zusätzlich prüfen, ob der indirekte Effekt von Zeitdruck auf die psychosomatischen Beschwerden signfikant ist.
Grafische Veranschaulichung des Modells und der Ergebnisse
Das Paket semPlot bietet die Möglichkeit, Regressionen, CFAs, Pfadanalysen und Strukturgleichungsmodelle, die bspw. mit lavaan geschätzt wurden, grafisch zu veranschaulichen. Diese Grafiken sind denen, die wir in den inhaltlichen Sitzungen kennen gelernt haben, sehr ähnlich. semPaths (aus dem semPlot-Paket) ist die Funktion, welche wir hierzu nutzen wollen. Sie nimmt als Argument das geschätzte Objekt, welches in unserem Fall die Pfadanalyse enthält, entgegen; hier: fit_paths. Ohne weitere Zusatzeinstellungen sieht dieses so aus:
semPaths(fit_paths)
Der Default stellt also einfach nur das Modell grafisch dar, was sehr praktisch ist, um bspw. zu prüfen, ob alle wichtigen Beziehung im Modell enthalten sind. Gestrichelte Pfeile stehen hierbei für Restriktionen, hier wird also nichts geschätzt: in unserem Bespiel wird die Varianz von ZDs nicht geschätzt, da die Varianz der unabhängigen Variable nur implizit in die Regressionskoeffizenten einfließt, aber kein Koeffizient des Modells darstellen. Würden wir wollen, dass die Varianz von ZDs geschätzt wird, so könnten wir prinzipiell ZDs ~~ ZDs in unser Modell mit aufnehmen.
Gerichtete Pfeile sind regressive Beziehung (also Regressionsparameter, Pfadkoeffizienten oder Faktorladungen). Ungerichtete Pfeile stellen Kovarianzen oder Residualvarianzen dar. Hierbei wird immer dann von einer Kovarianz im Gegensatz zu einer Residualkovarianz gesprochen, wenn es sich um eine exogene (unabhängige) Variable handelt. Salopp gesprochen: hier kommen keine gerichteten Pfeile an! Ein weiteres wichtiges Argument, welches semPaths entgegennimmt ist what. Hiermit wird festgelegt, was genau geplottet werden soll. Wählen wir what = "model", so wird das Modell ohne Parameterschätzungen und Einfärbungen grafisch dargestellt - dies ist der Default, welchen wir bereits oben gesehen haben. Wählen wir hingegen what = "est", so werden alle geschätzten Parameter in das Modell eingezeichnet; diese werden auch farblich hinsichtlich ihrere Ausprägung kodiert.
semPaths(fit_paths, what = "est")
Probieren Sie doch selbst einmal aus, was die folgenden Zusatzeinstellungen bewirken:
semPaths(object = fit_paths, what = "model", layout = "tree2", rotation = 2,
col = list(man = "skyblue"), edge.label.cex=1, sizeMan = 5)
Sie können die Argumente der Funktion semPaths nachlesen, indem sie ??semPaths in einem neuen R-Studio Fenster ausführen und dann semPlot::semPaths in der Übersicht auswählen oder sie schauen sich die Dokumentation online hier an.
semPaths(object = fit_paths, what = "est", layout = "tree2", rotation = 2,
col = list(man = "skyblue"), edge.label.cex=1, sizeMan = 5)
Wenn Sie eine solche Grafik in R-Studio erzeugen (nicht hier im HTML), gibt es viele Möglichkeiten, diese abzuspeichern. Bspw. können Sie mit dev.print(device = pdf, "MeinPlot.pdf") die Grafik als PDF abspeichern und ihr den Namen "MeinPlot.pdf" geben. Die Endung ".pdf" ist hierbei obligatorisch. Eine andere Möglichkeit ist in R-Studio im Grafikfenster auf "Export" zu klicken und dann den Anweisungen zu folgen.
Berechnen und Testen des indirekten Effektes von Zeitdruck über emotionale Erschöpfung auf psychosomatische Beschwerden
Der indirekte Effekt ist der Effekt, der von der unabhängigen Variable über den Mediator auf die abhängige Variable wirkt. Der Effekt der unabhängigen Variable auf den Mediator wird häufig mit a bezeichnet. Der Effekt des Mediators wird häufig b genannt. Der verbleibende direkte Effekte der unabhängigen auf die abhängige Variable wird, sie haben es sich womöglich schon gedacht, mit c bezeichnet. Nach diesem Schema wollen wir auch die Koeffizienten in unserem Modell benennen. Schauen wir uns dazu einmal die Gleichungen an (der Vollständigkeit halber führen wir auch die Interzepts $a_0$ und $c_0$ mit):
$$BOEE_i = a_0 + aZD_i + \varepsilon_{BOEE,i},$$
$$BF_i = c_0 + bBOEE_i + cZD_i + \varepsilon_{BF,i}.$$
Nun haben wir allerdings eine Gleichung für $BOEE_i$, also können wir diese in die Gleichung von $BF_i$ einstetzen und erhalten den indirekten Effekt (IE) als Produkt der Parameter $IE:=ab$; der direkte Effekt (DE) verbleibt $DE:=c$:
$$BF_i = c_0 + b(a_0 + aZD_i + \varepsilon_{BOEE,i}) + cZD_i + \varepsilon_{BF,i} = \underbrace{(c_0 + ba_0)}{\text{Interzept}} + \underbrace{ab}{{\text{IE}}}ZD_i + \underbrace{c}{{\text{DE}}}ZD_i + \underbrace{(b \varepsilon{BOEE,i}+ \varepsilon_{BF,i})}_\text{Residuum}.$$
Der totale Effekt von Zeitdruck auf die psychosomatischen Beschwerden ergibt sich als $TE:=IE + DE = ab+c$. Da wir bisher in lavaan die Mittelwertstruktur nicht mitmodelliert hatten, haben wir $a_0$ und $c_0$ auch noch nicht untersucht. Für weitere Informationen zu Pfadanalysen, direkten, indirekten und totalen Effekten lesen Sie gerne in Eid, Gollwitzer und Schmitt (2017, pp. 952-).
In lavaan Koeffizienten zu bennen, ist ganz einfach. Sie haben es vielleicht schon im Appendix der letzten Sitzung (PsyMSc1::Sitzung_3()) gesehen: der Variable wird der Koeffizientenname gefolgt von dem Multiplikationszeichen * vorangestellt. Also wird die Beziehung zwischen BOEEs und ZDs um das Präfix a* ergänzt zu: BOEEs ~ a*ZDs, usw.:
model_paths_abc <- '
BOEEs ~ a*ZDs
BFs ~ b*BOEEs + c*ZDs
'
Der Output bleibt nach Schätzen des Modells identisch, allerdings werden die Namen der Koeffizienten im Output mitgeführt. So lässt sich leicht prüfen, ob die Bennungen an den richtigen Stellen gelandet sind:
fit_paths_abc <- sem(model_paths_abc, data = StressAtWork)
summary(fit_paths_abc)
abbrev(fit_paths_abc, begin = "Regressions", end = "Variances")
Um nun den indirekten Effekt $ab$ und den totalen Effeket $ab + c$ mit aufzunehmen, können wir diese in das lavaan Modell inkludieren. Diese neu definierten Parameter stellen allerdings keine weiteren Modellparameter dar, es gehen also keine Freiheitsgrade verloren. Die lavaan-Syntax, die hinzukommt, ist :=; das mathematische "definiert als"-Zeichen. Links davon steht der Namen, den wir dem neuen definierten Parameter geben wollen und rechts steht die Funktion der anderen Parameter aus unserem Modell. Beispielsweise können wir den indirekten Effekt wie folgt definieren: IE := a*b; das Produkt der beiden Pfadkoeffizienten, die wir bereits benannt haben.
model_paths_IE_TE <- '
BOEEs ~ a*ZDs
BFs ~ b*BOEEs + c*ZDs
# Neue Parameter
IE := a*b
TE := IE + c
'
Wenn wir nun das Modell erneut schätzen, erhalten wir einen neuen Teil im Output der Summary, welcher unsere definierten Parameter und deren Standardfehler, sowie die zugehörigen p-Werte enthält.
fit_paths_IE_TE <- sem(model_paths_IE_TE, data = StressAtWork)
summary(fit_paths_IE_TE)
abbrev(fit_paths_IE_TE, begin = "Defined Parameters", shift = 1)
Achtung! Leider können wir den angezeigten Standardfehlern und dem zugehörigen p-Wert nicht unbedingt vertrauen, da einige Studien gezeigt haben, dass der indirekte Effekt asymptotisch nicht immer normalverteilt bzw. symmetrisch verteilt ist, weswegen ein einfaches Teilen des Estimates durch SE und der Vergleich mit der $z$-Verteilung (so wie wir dies eigentlich immer tun; also die einfache Parametersignifikanzentscheidung in komplexen Modellen) in diesem Fall nicht sinnvoll erscheint. Aus diesem Grund möchten wir dieser Problematik entgehen, indem wir die Bootstrap-Methode verwenden.
Bootstrapping
Bootstrapping ist das wiederholte Ziehen mit Zurücklegen aus unserer Stichprobe, solange, bis wir eine neue Stichprobe erhalten, die genauso groß ist, wie die ursprünglich Beobachtete. In dieser neuen, gezogenen Stichproben schätzen wir erneut unser Modell und notieren uns den indirekten Effekt. Dieses Vorgehen wiederholen wir sehr häufig (es hat sich irgendwie so ergeben, dass in der Statistik sehr häufig bei ungefähr und ziemlich genau 1000 liegt), sodass wir die Verteilung des indirekten Effektes und den wahren Standardfehler (also die Streuung des indirekten Effekts) annähern können. Dieses Vorgehen sollte, wenn unsere Stichprobe unabhängig gezogene Personen enthält und repräsentativ für die Gesamtpopulation ist, eine gute Schätzung der Verteilung des indirekten Effektes liefern, welcher Schlüsse auf die Gesamtpopulation zulässt. Wir können dann einfach den 2.5-ten und den 97.5-ten Prozentrang dieser Verteilung verwenden und erhalten ein 95%-Konfidenzintervall für den indirekten Effekt (dieses muss nicht immer symmetrisch sein!). Zum Glück müssen wir dies nicht mit Hand programmieren, sondern können einfach unserer Parameterschätzung mit sem die Zusatzargumente se = "boot" und bootstrap = 1000 hinzufügen. Es empfiehlt sich, diese Objekt anschließend anders zu nennen. Wir lassen unsere Phantasie freien Lauf und nennen die neue "gebootstrapte" Schätzung fit_paths_IE_TE_boot. Wenn wir dann auf das geschätze (neue) Objekt die Funktion parameterEstimates mit der Zusatzeinstellung ci = TRUE anwenden, so werden uns alle Parameter inklusive Konfidenzintervall zurückgegeben. Da es sich hierbei um einen Zufallsprozess handelt, werden die Werte bei mehrmaligem Wiederholen (leicht) unterschiedlich sein. Möchten wir das Ergebnis replizierbar machen, so können wir set.seed() verwenden, wir müssen dieser Funktion lediglich eine beliebige natürliche Zahl übergeben (wie wäre es bspw. mit 1234? Wenn Sie die gleiche R-Version haben, sollte der folgende Code zum selben Ergebnis kommen!).
set.seed(1234)
fit_paths_IE_TE_boot <- sem(model_paths_IE_TE, data = StressAtWork, se = "boot", bootstrap = 1000)
parameterEstimates(fit_paths_IE_TE_boot, ci = TRUE)
load('./data/fit_paths_IE_TE_boot.RData')
cat("[...]")
print(parameterEstimates(fit_paths_IE_TE_boot, ci = TRUE)[7:8,])
cat("[...]")
Der Output zeigt die Konfidenzintervalle (sowie weitere Informationen) für alle Parameter in unserem Modell sowie für den indirekten und den totalen Effekt. Hierbei steht ci.lower für die untere Grenze und ci.upper für die obere Grenze des Konfidenzintervalls. Die Nummern 7 und 8 am Anfang der Zeilen zeigen an, dass hier insgesamt 8 Zeilen ausgegeben werden, wir uns aber nur die 7. und die 8. ansehen. Zeile 1 bis 6 (hier nicht dargestellt) zeigen den gleichen Output für alle weiteren Parameter im Modell (wir können help("parameterEstimates") für weitere Informationen in R-Studio ausführen, nachdem wir lavaan via library(lavaan) geladen haben). Das "gebootstrapte" Konfidenzintervall des indirekten Effekts erstreckt sich von r round(parameterEstimates(fit_paths_IE_TE_boot, ci = TRUE)[7,9], 3) bis r round(parameterEstimates(fit_paths_IE_TE_boot, ci = TRUE)[7,10], 3) und schließt die 0 somit nicht ein. Dies bedeutet, dass auf dem 5%-Signifikanzniveau der indirekte Effekt in der Population nicht 0 ist.
Dem Output zufolge scheint es also in der Population einen indirekten Effekt zu geben. Der totale Effekt ist auch signifikant. Die zugehörigen Grafiken unterscheiden sich kaum von denen, die wir uns zuvor angesehen hatten. Schauen wir uns die Modellstruktur an, so werden uns allerdings die Parameternamen mit eingezeichnet, was durchaus von Nutzen sein kann. Der indireke oder der totale Effekt sind keine Modellparameter, weswegen sie auch nicht in der Grafik dargestellt werden können.
semPaths(object = fit_paths_IE_TE, what = "model", layout = "tree2", rotation = 2,
col = list(man = "skyblue"), edge.label.cex=1)
Die hier durchgeführten Analysen unterliegen leider einigen Einschränkungen. Bspw. wird beim Mitteln zu Skalenwerten davon ausgegangen, dass jede Messung (jede Variable pro Skala) gleichermaßen aus der dahinterliegende latenten Variable besteht. Dies hatten wir in der letzten Sitzung im Anhang unter essentieller $\tau$-Äquivalenz kennengelernt (gleiche $\lambda$s in einem [CFA-] Messmodell). Essentielle $\tau$-Äquivalenz ist eine strenge Annahme, welche wir prüfen müssten, um den Analysen mit Skalenwerten komplett vertrauen zu können. Außerdem werden durch das Mitteln die Messfehler nicht vollständig modeliert, auch wenn die Analysen somit reliabler als Einzelitemanalysen (z.B. hätten wir auch jeweils ein Item pro Skala verwenden können) sind.
Hierbei ist es nun so, dass Effekte stochastischer Regressoren konsistent unterschätzt werden, wenn diese messfehlerbehaftet sind. Wären die Variablen messfehlerfrei, würden die Effekte (die Regressionsparameter) größer ausfallen. Leider können wir nicht davon ausgehen, dass unsere beobachteten Variablen messfehlerfrei sind, wenn wir sie mit Fragebögen erheben! Aus diesem Grund ist in der multiplen Regression bspw. auch eine der Voraussetzungen die Messfehlerfreiheit der (stochastischen) Regressoren (siehe Eid, et al., 2017, Kapitel 19.13).
Wir können die Analysen auf die latente Ebene heben und Messmodelle für die latenten Variablen aufstellen, welche dann die unterschiedlichen Messgenauigkeiten pro Messung berücksichtigen und die Beziehung zwischen den latenten Variablen um die Ungenauigkeit der Einzelitems bereinigen.
Strukturgleichungsmodelle
Strukturgleichungsmodelle (SEM) kombinieren Pfadanalysen mit Messmodellen, also CFAs. Wir können mit SEM in unseren Analysen Messfehler berücksichtigen, aber dennoch gerichtete Beziehungen zwischen den latenten Variablen untersuchen. Bevor wir Messmodelle für unsere latenten Variablen aufstellen, müssen wir uns überlegen, ob dies denn für alle Variablen sinnvoll ist.
Reflexive vs. formative Messmodelle {#formvsreflMessmodell}
Wenn es um Messmodelle geht, kann zwischen reflexiven und formativen Messmodellen unterschieden werden. Reflexive Messmodelle sind die Messmodelle, die wir in der letzten Sitzung detailliert besprochen haben. Darin sind Eigenschaften von Personen latente Variable, die sich nicht direkt beobachten lassen, sich aber in Verhalten und Aussagen niederschlagen. "Konservativismus" ist eine Einstellung von Personen (latente Variable), die sich z.B. darin äußert, dass Personen einer Aussage wie "Ein noch so geschulter und kritischer Verstand kann letzten Endes doch keine echte innere Befriedigung geben." eher zustimmen (Item 36 der Machiavellismus-Konservatismus Skala; Cloetta, 2014). Die Annahme ist also, dass die latente Variable ursächlich für die Ausprägung bestimmter beobachtbarer (manifester) Variablen ist. Wie wir in Sitzung 3 gesehen haben, sieht ein entsprechendes Pfaddiagramm so aus:
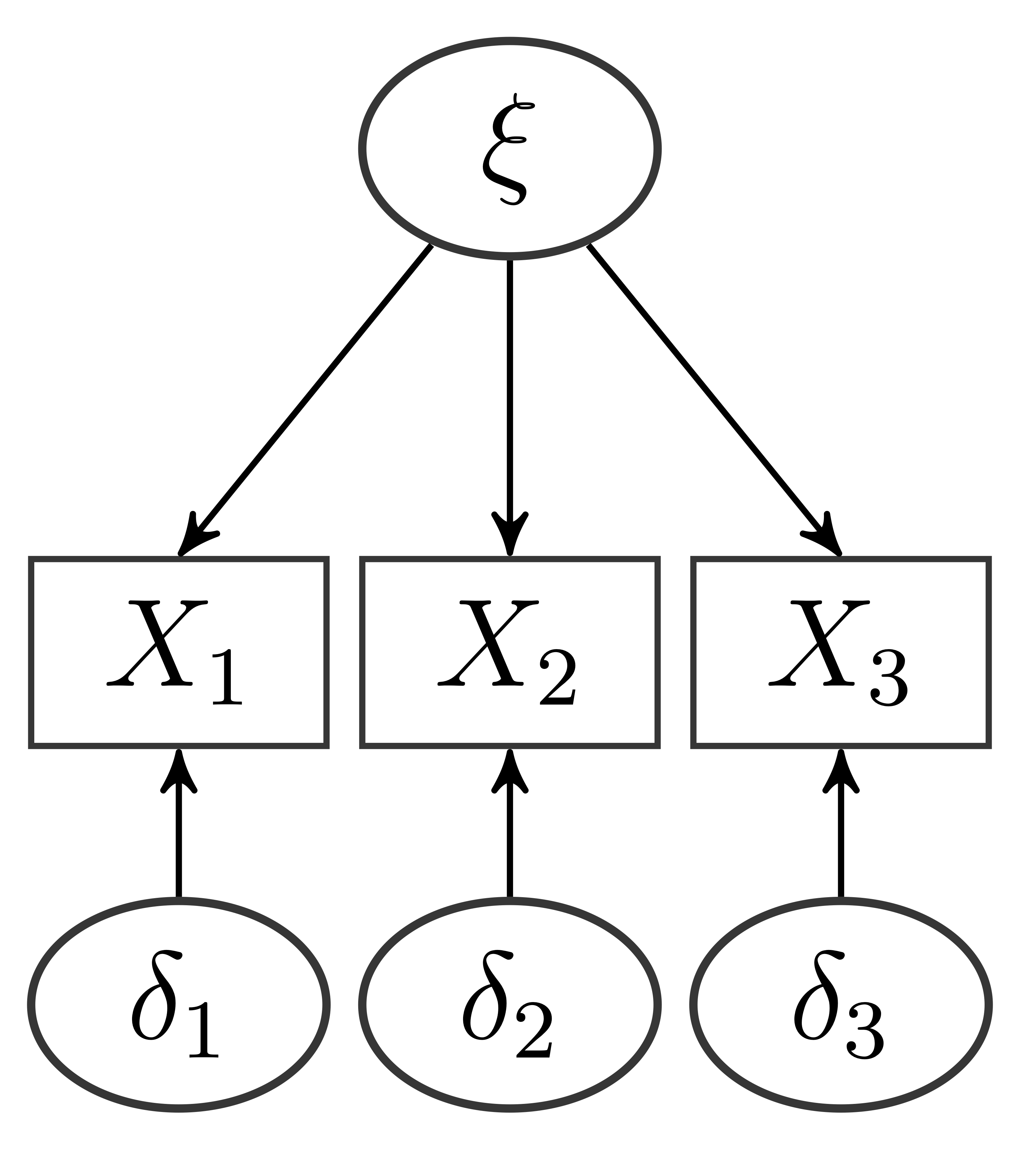
Die Variable $X_1$ ist also eine abhängige Variable, deren Ausprägung von der Ausprägung der latenten Variable $\xi$ und vom Messfehler $\delta_1$ abhängt: $X_1=\lambda\xi + \delta_1$. Deswegen gehen die Pfeile im Pfaddiagramm von den latenten Variablen (UV) zu den Items (AVs).
Bei formativen Messmodellen ist es so, dass die latente Variable erst "geformt" wird. Sie ist eine Zusammensetzung aus den manifesten (beobachteten) Items, die als die (gewichtete) Summe (oder [gewichteter] Mittelwert) ihrer Items aufgefasst wird. Das zugehörige Pfaddiagramm sieht folgendermaßen aus:
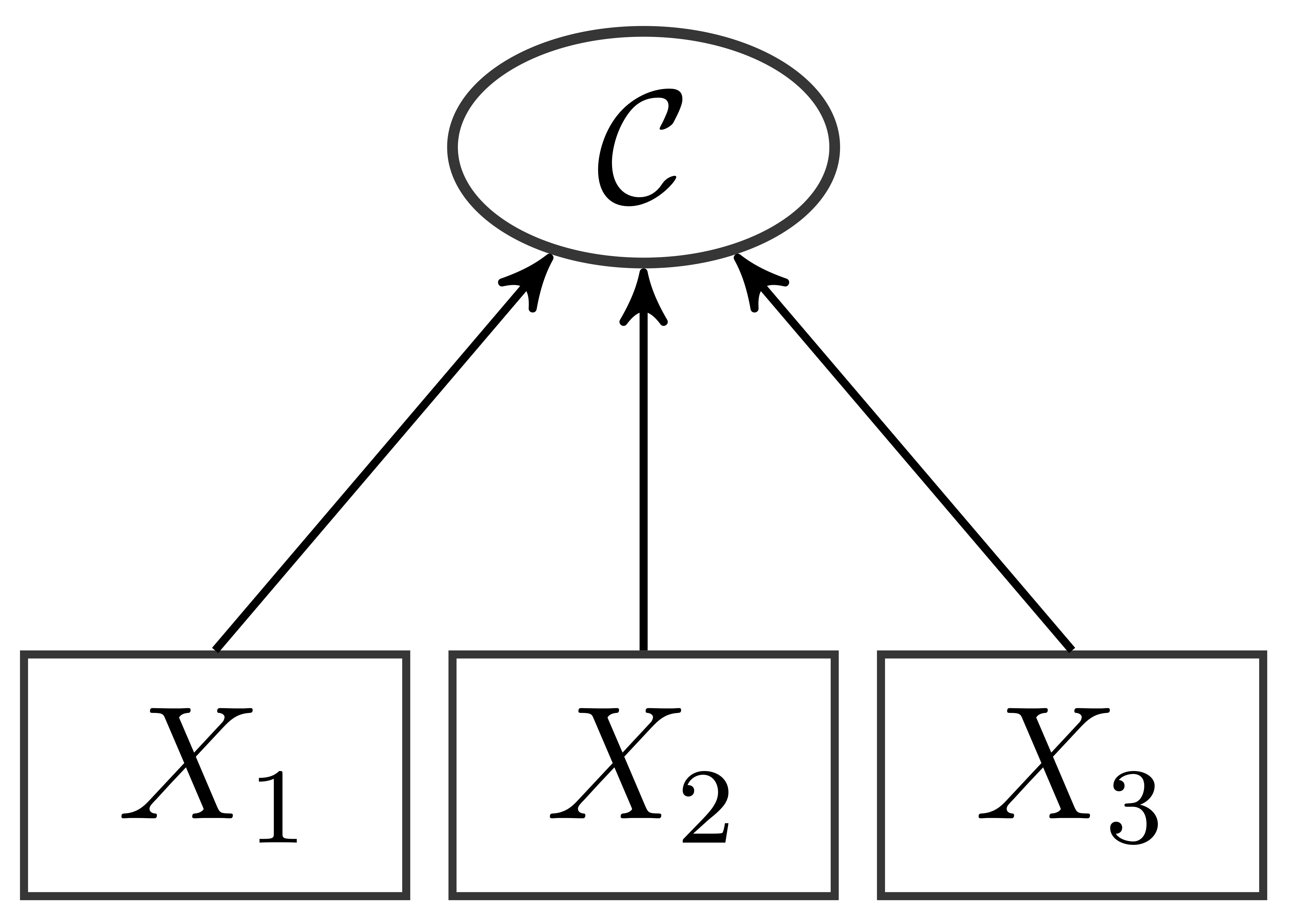
Es können keine Messfehler berücksichtigt werden und die "Kompositvariable" (deshalb auch die Benennung $\mathcal{C}$, engl. composite) ist eine Linearkombination der Items: $\mathcal{C}=X_1+X_2+X_3$ oder $\mathcal{C}=\lambda_1^\mathcal{C}X_1+\lambda_2^\mathcal{C}X_2+\lambda_3^\mathcal{C}X_3$ (also auch: $\mathcal{C}=\frac{X_1+X_2+X_3}{3}$)). Somit wirken sich Unterschiede zwischen Personen in bspw. $X_1$ (und gleichen $X_2$ und $X_3$) auf Unterschiede in $\mathcal{C}$ aus. Es lassen sich also Unterschiede in der latenten Variable auf unterschiedliche Kombinationen von Ausprägungen der Beobachtungen zurückführen. Gerade in den Wirtschaftswissenschaften und der Soziologie sind solche formativen Variablen sehr verbreitet. Ein bekanntes Beispiel ist der Human Development Index, der aus verschiedenen Komponenten wie Lebenserwartung, Schulbildung und Bruttoinlandsprodukt eine Kennzahl für den Entwicklungsstand eines Landes ermittelt. Ein psychologisches Beispiel für ein formatives Messmodell ist die Variable Umgebungsbelastung, welche auch mit dem ISTA erhoben werden kann (Semmer, et al., 1999). Mit dieser Variable sollen Umwelteinflüsse erfasst werden, die die Arbeit erschweren. Dies könnten z.B. schlechte Lichtverhältnisse oder Lärmbelästigung sein, welche durch längeres Auftreten Stress zur Folge haben können. Allerdings ist es in der Regel nicht so, dass an einem Arbeitsplatz immer dann Lärm auftritt, wenn auch die Lichtverhältnisse schlecht sind. Wenn jedoch beides auftritt, so ist die Umgebungsbelastung an diesem Arbeitsplatz besonders ausgeprägt. Demnach müssen beide Aspekte berücksichtigt werden, um eine gute Schätzung für die Umgebungsbelastung eines Arbeitsplatzes zu erhalten. Würden wir von einem reflexiven Messmodell ausgehen, würde eine hohe Ausprägung auf der latenten Variable Umgebungsbelastung auch zu einer erhöhten Antworttendenz auf beiden Items (Licht- und Lärmverhältnisse) führen.
Wem dieses Konzept bekannt vorkommt, hat sich wahrscheinlich an die Unterscheidung zwischen EFA und PCA zurückerinnert: Hier war es so, dass bei der PCA die Hauptkomponenten Linearkombinationen, also gewichtete Summen, der beobachteten Items waren, während bei EFA ein Messmodell mit dahinterliegenden latenten Variablen formuliert wurde und somit Messfehler modeliert werden konnten.
Bei Zeitdurck und emotionaler Erschöpfung handelt es sich um reflexive Messmodelle. Bei psychosomatischen Beschwerden werden unterschiedliche Beschwerden wie bspw. Kopfschmerzen und Rückenschmerzen abgefragt. Demnach handelt es sich bei dieser Variable eher um ein ein formatives Messmodell. Entsprechend könnten wir die Kompositformulierung in lavaan (<~) nutzen, um eine latente Kompositvariable zu erzeugen oder wir behalten der Einfachheit halber unseren Skalenmittelwert BFs bei.
Das Modell
Das Modell in SEM unterscheidet sich im Vergleich zu CFAs: Es wird zwischen Messmodell und Strukturmodell unterschieden. Im Strukturmodell gibt es gerichtete Beziehungen, in welchen zwischen unabhängigen und abhängigen latenten Variablen unterschieden werden kann. Diese werden häufig auch exogene (unabhängige) und endogene (abhängige) latente Variablen genannt. Außerdem gibt es zwei Arten von Messmodellen: ein exogenes und ein endogenes Messmodell. Diese Messmodelle sind quasi CFAs für die exogenen und die endogenen latenten Variablen; spezifizieren also die Beziehungen zwischen den latenten und den manifesten unabhängen bzw. abhängen Variablen. Wir wollen uns die Messmodelle sowie das Strukturmodell unserer Analyse ansehen:
- Exogenes Messmodell
$$\begin{pmatrix}ZD_1\ZD_2\ZD_6\end{pmatrix}=\begin{pmatrix}\tau_1^{x}\\tau_2^{x}\\tau_6^{x}\end{pmatrix}+\begin{pmatrix}\lambda_{11}^{x}=1\\lambda_{21}^{x}\\lambda_{31}^{x}\end{pmatrix}\xi_{\text{ZD}} + \begin{pmatrix}\delta_1\\delta_2\\delta_6\end{pmatrix}$$
- Endogenes Messmodell
$$\begin{pmatrix}BO_1\BO_6\BO_{12}\BO_{19}\end{pmatrix}=\begin{pmatrix}\tau_1^{y}\\tau_6^{y}\\tau_{12}^{y}\\tau_{19}^{y}\end{pmatrix}+\begin{pmatrix}\lambda_{11}^{y}=1\\lambda_{21}^{y}\\lambda_{31}^{y}\\lambda_{41}^{y}\end{pmatrix}\eta_{\text{BOEE}} + \begin{pmatrix}\varepsilon_1\\varepsilon_6\\varepsilon_{12}\\varepsilon_{19}\end{pmatrix}$$
- Strukturmodell in Matrixnotation (wobei wir $BFs$ als latente Variable mitführen)
$$\begin{pmatrix}\eta_\text{BOEE}\BF_s\end{pmatrix}=\begin{pmatrix}\gamma_{11}\\gamma_{21}\end{pmatrix}\xi_\text{ZD} + \begin{pmatrix}0&0\\beta_{21}&0\end{pmatrix}\begin{pmatrix}\eta_\text{BOEE}\BF_s\end{pmatrix} + \begin{pmatrix}\zeta_\text{BOEE}\\zeta_{BF_s}\end{pmatrix}$$
Für die einzelnen abhängigen latenten Variablen erhalten wir also:
$$\eta_\text{BOEE}=\gamma_{11}\xi_\text{ZD} + \zeta_\text{BOEE}$$
und
$$BFs = \gamma_{21}\xi_\text{ZD} + \beta_{21}\eta_\text{BOEE} + \zeta_{BFs}.$$
Durch Einsetzten folgt:
$$BFs = \gamma_{21}\xi_\text{ZD} + \beta_{21}\gamma_{11}\xi_\text{ZD}+ \beta_{21}\zeta_\text{BOEE} + \zeta_{BFs}.$$
Wenn wir nun genauer hinschauen, erkennen wir, dass $\gamma_{11}$ dem Pfad $a$, $\beta_{21}$ dem Pfad $b$ und $\gamma_{21}$ dem Pfad $c$ aus dem Mediationspfadmodell entspricht! Entsprechend quantifiziert $\beta_{21}\gamma_{11}$ den indirekten Effekt von $\xi_\text{ZD}$ über $\eta_\text{BOEE}$ auf $BFs$. Wir wollen das Modell model_paths_IE_TE, welches so aussah:
model_paths_IE_TE <- '
BOEEs ~ a*ZDs
BFs ~ b*BOEEs + c*ZDs
# Neue Parameter
IE := a*b
TE := IE + c
'
so umstellen, dass es nicht mehr die Beziehung von manifesten Skalenwerten, sondern die Beziehungen zwischen latenten Variablen untersucht. Probieren Sie unten aus, ein solches Modell aufzustellen, indem Sie Ihr Wissen um die CFA aus der letzten Sitzung mit dem kombinieren, was gerade in der Pfadaanalyse besprochen wurde. Nennen Sie die latente Variable für Zeitdruck am besten ZD (Items: zd1, zd2 und zd6) und die latente Variable emotionale Erschöpfung BOEE (Items: bo1, bo6, bo12 und bo19; Achtung: ohne "s" - diese Variablen gibt es nämlich im Datensatz - das waren die Skalenmittelwerte für Zeitdruck und emotionale Erschöpfung). Nehmen Sie als Platzhalter der psychosomatischen Beschwerden die Variable BFs in das Modell auf. Inkludieren Sie auch die Berechnung des indirekten und des totalen Effekts mit den gleichen Bezeichnungen. Sie brauchen folglich 2 Messmodelle, eine Regression für die Beziehung zwischen ZD und BOEE, sowie eine Regression für die Beziehung zwischen ZD, BOEE und BFs. Zum Schluss brauchen Sie noch die neu definierten Koeffizienten für den indirekten und den totalen Effekt (vgl. oben). Schätzen Sie anschließend das Modell mit sem und schauen Sie sich die Summary an! (In den Tips finden Sie Schrittweise mehr und mehr Vorgaben dazu, wie dieses Modell aussehen soll)
model_sem_IE_TE <- '
# Messmodelle
...
# Strukturmodell
...
# Neue Parameter
...
'
fit_sem_IE_TE <- ...
...
model_sem_IE_TE <- '
# Messmodelle
ZD =~ ... + ...
...
# Strukturmodell
...
# Neue Parameter
...
'
fit_sem_IE_TE <-
...
model_sem_IE_TE <- '
# Messmodelle
ZD =~ zd1 + ...
...
# Strukturmodell
...
# Neue Parameter
...
'
fit_sem_IE_TE <-
...
model_sem_IE_TE <- '
# Messmodelle
ZD =~ zd1 + ...
...
# Strukturmodell
BOEE ~ ...
BFs ~ ... + ...
# Neue Parameter
...
'
fit_sem_IE_TE <-
...
model_sem_IE_TE <- '
# Messmodelle
ZD =~ zd1 + ...
BOEE =~ ...
# Strukturmodell
BOEE ~ ...
BFs ~ ... + ...
# Neue Parameter
IE := a*b
TE := ...
'
fit_sem_IE_TE <-
...
model_sem_IE_TE <- '
# Messmodelle
ZD =~ zd1 + ...
BOEE =~ ...
# Strukturmodell
BOEE ~ a*ZD
BFs ~ ... + ...
# Neue Parameter
IE := a*b
TE := ...
'
fit_sem_IE_TE <- sem(...)
summary(...)
model_sem_IE_TE <- '
# Messmodelle
ZD =~ zd1 + ...
BOEE =~ ...
# Strukturmodell
BOEE ~ a*ZD
BFs ~ ... + ...
# Neue Parameter
IE := a*b
TE := ...
'
fit_sem_IE_TE <- sem(model_sem_IE_TE, data = StressAtWork)
summary(fit_sem_IE_TE)
model_sem_IE_TE <- '
# Messmodelle
ZD =~ zd1 + zd2 + zd6
BOEE =~ bo1 + bo6 + bo12 + bo19
# Strukturmodell
BOEE ~ a*ZD
BFs ~ b*BOEE + c*ZD
# Neue Parameter
IE := a*b
TE := IE + c
'
fit_sem_IE_TE <- sem(model_sem_IE_TE, StressAtWork)
## Skalen bilden
StressAtWork$ZDs <- rowMeans(StressAtWork[,paste0("zd",c(1, 2, 6))])
StressAtWork$BOEEs <- rowMeans(StressAtWork[,paste0("bo",c(1, 6, 12, 19))])
StressAtWork$BFs <- rowMeans(StressAtWork[,paste0("bf",1:20)])
## Modell spezifizieren
model_paths <- '
BOEEs ~ ZDs
BFs ~ BOEEs + ZDs
'
## Modell fitten
fit_paths <- sem(model_paths, data = StressAtWork)
#### SEM:
# Modell
model_sem_IE_TE <- '
# Messmodelle
ZD =~ zd1 + zd2 + zd6
BOEE =~ bo1 + bo6 + bo12 + bo19
# Strukturmodell
BOEE ~ a*ZD
BFs ~ b*BOEE + c*ZD
# Neue Parameter
IE := a*b
TE := IE + c
'
fit_sem_IE_TE <- sem(model_sem_IE_TE, StressAtWork)
Sollten Sie alles richtig formuliert haben (und die Zusatzeinstellungen in der summary als fit.measures = T, rsq = T gewählt haben), so sollten Sie folgenden Modellfit erhalten:
abbrev(fit_sem_IE_TE, begin = "Model Test User Model", end = "Parameter Estimates", fit.measures = T, rsq = T)
Falls dem so ist - herzlichen Glückwunsch, Sie haben gerade ein vollständiges Strukturgleichungsmodell formuliert und geschätzt! Jetzt bleibt nur noch die leichteste Übung: Ergebnisse korrekte interpretieren.
Dem Modelfit ist zu entnehmen, dass wir, anders als im Beispiel der Pfadanalyse, nun die Passung zwischen Modell und Daten untersuchen können: der $\chi^2$-Wert liegt bei r round(fitmeasures(fit_sem_IE_TE)[3], 3) bei $df=$ r round(fitmeasures(fit_sem_IE_TE)[4], 3) mit zugehörigem $p$-Wert von r round(fitmeasures(fit_sem_IE_TE)[5], 3). Demnach verwerfen unsere Daten das Modell nicht.
Die Fit-Indizes zeigen hier allesamt einen guten Fit des Modells an. Dies war Aufgrund des $\chi^2$-Wertes zu erwarten, da die Stichprobengröße ausreichend Power liefert (sodass das Modell sinnvoll geschätzt werden kann) und der $\chi^2$-Wert trotzdem nicht signifikant auf dem 5%-Niveau war. Es gibt also keinen Grund an der $H_0$ (auf Modellpassung zwischen Daten und Modell) zu zweifeln. Denn nur in solchen Modellen, in denen tatsächlich die $H_1$-Hypothese gilt, wächst der $\chi^2$-Wert mit der Stichprobengröße (dies steht im Widerspruch zu einigen Textbüchern, welche propagieren, dass der $\chi^2$-Wert immer mit der Stichprobengröße wächst!). Es empfiehlt sich sehr über die Beziehung zwischen Stichprobengröße, $\chi^2$-Wert und den Fit-Indizes noch einmal im Appendix A nachzulesen!
Für die Messmodelle ergibt sich Folgendes:
abbrev(fit_sem_IE_TE, begin = "Latent Variables", end = "Regressions")
Diesen entnehmen wir, dass die Variablen auf der latenten Dimension ähnlich stark diskriminieren (ähnlich große Faktorladungen). Wir sehen außerdem, dass die ersten Faktorladungen jeweils auf 1 gesetzt sind und hier entsprechend keine Signifikanzentscheidung vonnöten ist (wir haben den Wert hier "ohne Unsicherheit" vorgegeben). Um die Reliabilität beurteilen zu können, schauen wir uns die erklärten Varianzanateile an:
abbrev(fit_sem_IE_TE, begin = "R-Square", end = "Defined Parameters", rsq = T)
Die beobachteten Variablen zeigen Reliabilitäten zwischen r round(min(inspect(fit_sem_IE_TE, "r2")[1:7]), 3) und r round(max(inspect(fit_sem_IE_TE, "r2")[1:7]), 3).
Nun wollen wir die Ergebnisse mit den Ergebnissen der Pfadanalyse vergleichen. Dazu schauen wir uns die gerichteten Beziehungen zwischen den latenten Variablen an:
abbrev(fit_sem_IE_TE, begin = "Regressions", end = "Variances")
Hier werden uns erneut die Label "(a)", "(b)" und "(c)" angezeigt. Der direkte Effekt (c) von Zeitdruck auf psychosomatische Beschwerden scheint, im Gegensatz zu den Ergebnissen der Pfadanalyse im Abschnitt zuvor, nicht signifikant von 0 verschieden zu sein. Die beiden anderen Pfade sind statistisch signifikant:
Auch für diese Analysen ist der $R^2$-Output interessant:
abbrev(fit_sem_IE_TE, begin = "R-Square", end = "Defined Parameters", rsq = T)
Im Bezug auf die latenten Regressionen enthält der R-Square Output die Anteile erklärter Varianzen von BOEE und BFs. Demnach können r round(inspect(fit_sem_IE_TE, "r2")[8]*100, 2)% der Variation der psychosomatischen Beschwerden durch die emotionale Erschöpfung und Zeitdruck (obwohl die Beziehung nicht signifikant ist) erklärt werden. r round(inspect(fit_sem_IE_TE, "r2")[9]*100, 2)% der Variation der emotionale Erschöpfung werden durch Zeitdruck erklärt. Somit kann etwas mehr Variation erklärt werden als im Pfadanalysefall (dort: r round(inspect(fit_paths_IE_TE, "r2")[2]*100, 2)% der Variation der psychosomatischen Beschwerden durch die emotionale Erschöpfung und Zeitdruck sowie r round(inspect(fit_paths_IE_TE, "r2")[1]*100, 2)% der Variation der emotionale Erschöpfung durch Zeitdruck).
Berechnen und Testen des indirekten Effektes von Zeitdruck über emotionale Erschöpfung auf psychosomatische Beschwerden im SEM
Der indirekte und der totale Effekt liegen bei:
abbrev(fit_sem_IE_TE, begin = "Defined Parameters", end = NULL, shift = 1)
Der indirekte Effekt aus dem Pfadanalysemodell lag bei r round(parameterEstimates(fit_paths_IE_TE)[7,5], 3) und der totale bei r round(parameterEstimates(fit_paths_IE_TE)[8,5], 3). Somit liegen auch diese beiden Werte (deskriptiv gesehen) etwas höher, wenn wir latente Modellierung nutzen! Um den indirekten Effekt auf Signifikanz zu prüfen, benutzten wir wieder die Bootstrap-Methode (set.seed soll die Analyse replizierbar machen; schauen Sie dazu noch einmal im Abschnitt zur Pfadanalyse nach):
set.seed(12345)
fit_sem_IE_TE_boot <- sem(model_sem_IE_TE, data = StressAtWork, se = "boot", bootstrap = 1000)
parameterEstimates(fit_sem_IE_TE_boot, ci = TRUE)
load('./data/fit_sem_IE_TE_boot.RData')
print(parameterEstimates(fit_sem_IE_TE_boot, ci = TRUE)[21:22,])
Die Nummern 21 und 22 am Anfang der Zeilen zeigen an, dass hier insgesamt 22 Zeilen ausgegeben werden, wir uns aber nur die 21. und die 22. ansehen. Auch hier ist zu sehen, dass der indirekte Effekt signifikant von 0 abweicht. Da der direkte Effekt von Zeitdruck auf die psychosomatischen Beschwerden nicht signifikant ist, handelt es sich in diesem Zusammenhang um eine vollständige Mediation. Die vollständige lineare Beziehung zwischen Zeitdruck und psychosomatischen Beschwerden "fließt" über die emotionale Erschöpfung. Die Frage ist nun, warum es hier zu Unterschieden zwischen der Pfadanalyse und SEM kommt. Naiverweise würde wir zunächst erwarten, dass alle Effekte eher signifikant werden, wenn für Messfehler kontrolliert wird. So hatten wir zuvor mit messfehlerbehaften Regressoren in einer Regressionsanalyse argumentiert. Demnach würden wir eher erwarten, dass der direkte Effekt mit SEM signfikant ist. Allerdings haben wir in der Pfadanalyse die Daten unter der Annahme der essentiellen $\tau$-Äquivalenz zusammengefasst: wir haben einfach Mittelwerte ohne bestimmte Gewichtung verwendet. Im SEM haben wir die Faktorladungen frei Schätzen lassen - wir haben also statt eines gleichgewichteten formativen Konstrukts ein reflexives Konstrukt aus den Beziehungen in den Daten schätzen lassen. Zusammenfassend gehen wir also von einer vollständigen Mediation aus.
Grafische Repräsentation
Wir können diese Modelle auch wieder grafisch darstellen. Probieren Sie doch einmal aus, was in diesem Zusammenhang durch die folgendenen weiteren Einstellungen passiert:
semPaths(object = fit_sem_IE_TE, what = "model", layout = "tree2",
rotation = 2, curve = T, col = list(man = "skyblue", lat = "yellow"),
curvePivot = T, edge.label.cex=1.2, sizeMan = 5, sizeLat = 8)
semPaths(object = fit_sem_IE_TE, what = "est", layout = "tree2",
rotation = 2, curve = T, col = list(man = "skyblue", lat = "yellow"),
curvePivot = T, edge.label.cex=1.2, sizeMan = 5, sizeLat = 8)
curve = T und curvePivot = T haben in diesem Modell keinen Effekt, da es hierbei um die Kovarianzen geht und wird nur der Vollständigkeit halber aufgeführt. Der Effekt dieser beiden kann im Appendix A der ersten Grafik des $H_0$-Modells entnommen werden: diese Einstellungen bewirken die nicht vollständig runde Kurve der Fehlerkovarianz (durch curvePivot = T), welche vor allem dann die Übersichtlichkeit erhöht, wenn mehrere Kovarianzen vorhanden sind. Mit curve = F würden alle Fehlerkovarianzen und Kovarianzen (wenn vorhanden) als gerade Linien eingezeichnet werden.
BFs wird hier als Viereck dargestellt, da wir hier Werte gemittelt haben und diesen Skalenwert als direkt beobachtbare Variable in das Modell aufgenommen haben.
Multi Sample Analysis {#MSA}
In einer Multi-Sample-Analysis wird in mehreren Gruppen gleichzeitig ein Strukturgleichungsmodell geschätzt. Wir könnten uns bspw. fragen, ob die gleichen Beziehungen, so wie wir sie gerade beobachtet haben, gleichermaßen für Männer und Frauen gelten. Im Datensatz StressAtWork ist die Variable sex enthalten. Hier sind Frauen mit 1 und Männer mit 2 kodiert. Wenn wir diese Variable als Gruppierung verwenden, können wir die Invarianz der Parameter über das Geschlecht untersuchen. Um die Gruppierung in das Modell mit aufzunehmen, können wir in sem einfach dem Argument group den Namen der Gruppierungsvariable übergeben (hierbei sind die "Gänsefüßchen" wichtig!).
fit_sem_IE_TE_MSA <- sem(model_sem_IE_TE, data = StressAtWork, group = "sex")
summary(fit_sem_IE_TE_MSA)
Wir sehen, dass im Output nun für jede Gruppe das Modell einzeln geschätzt wurde. Alle Parameter werden sowohl für Frauen als auch für Männer geschätzt. Wir entnehmen,
abbrev(X = fit_sem_IE_TE_MSA, begin = "Number of observations per group", end = "Model Test User Model")
dass insgesamt r sum(StressAtWork$sex == 1) der Probanden Frauen und r sum(StressAtWork$sex == 2) Männer waren. Auch erhalten wir einen globalen sowie einen substichprobenspezifischen Modellfitwert:
abbrev(X = fit_sem_IE_TE_MSA, begin = "Model Test User Model", end = "Parameter Estimates")
Der $\chi^2$-Wert für das gesamte Modell liegt bei r round(fitmeasures(fit_sem_IE_TE_MSA)[3], 3) bei $df=$ r round(fitmeasures(fit_sem_IE_TE_MSA)[4], 3) mit zugehörigem $p$-Wert von r round(fitmeasures(fit_sem_IE_TE_MSA)[5], 3). Demnach verwerfen unsere Daten das Modell nicht. Die Freiheitsgrade sind doppelt so hoch, wie im Ein-Stichprobenfall, da wir alle Parameter für beide Stichproben schätzen müssen. Die $\chi^2$-Werte der beiden Stichproben waren 21.400 für die Frauen und 14.403 für die Männer. Der $\chi^2$-Wert für das gesamte Modell ist also einfach die Summe der subpopulationsspezifischen $\chi^2$-Werte ($\chi^2_{g=1}$ und $\chi^2_{g=2}$, wobei $g=1$ und $g=2$ für die erste und zweite Gruppe steht):
$$\chi^2=\chi^2_{g=1}+\chi^2_{g=2}.$$
In der gesamten Summary fällt uns auf, dass nur in Gruppe 1 die Benennungen der Pfadkoeffizienten mit $a, b$ und $c$ vorkommen und in Gruppe 2 keine Benennung mehr auftaucht.
abbrev(X = fit_sem_IE_TE_MSA, begin = "Group 1", end = "Intercepts")
abbrev(X = fit_sem_IE_TE_MSA, begin = "Group 2", end = "Intercepts")
Dies liegt daran, dass Benennungen für Parameter über Gruppen hinweg als Vektor übergeben werden müssen. Somit gehört auch der indirekte Effekt, den wir der Summary entnehmen können, nur zur Gruppe 1, also den Frauen:
abbrev(X = fit_sem_IE_TE_MSA, begin = "Defined Parameters", end = NULL, shift = 1)
Wir müssten also BOEE ~ c(a1, a2)*ZD schreiben, um den Effekt der unabhängigen Variable auf den Mediator in den Gruppen jeweils a1 und a2 zu nennen.
Außerdem fällt uns auf, dass sowohl die Faktorladungen als auch die Pfadkoeffizienten sich kaum über die Gruppen hinweg unterscheiden. Wir wollen den indirekten Effekt auch für die Männer berechnen und erweitern unser Modell entsprechend:
model_sem_IE_TE_MSA_abc <- '
# Messmodelle
ZD =~ zd1 + zd2 + zd6
BOEE =~ bo1 + bo6 + bo12 + bo19
# Strukturmodell
BOEE ~ c(a1, a2)*ZD
BFs ~ c(b1, b2)*BOEE + c(c1,c2)*ZD
# Neue Parameter
IE1 := a1*b1
TE1 := IE1 + c1
IE2 := a2*b2
TE2 := IE2 + c2
'
fit_sem_IE_TE_MSA_abc <- sem(model_sem_IE_TE_MSA_abc, StressAtWork, group = "sex")
summary(fit_sem_IE_TE_MSA_abc)
Nun sind alle Pfadkoeffizienten benannt:
abbrev(X = fit_sem_IE_TE_MSA_abc, begin = "Group 1", end = "Intercepts")
abbrev(X = fit_sem_IE_TE_MSA_abc, begin = "Group 2", end = "Intercepts")
Bis auf die hinzukommenden indirekten und totalen Effekte:
abbrev(X = fit_sem_IE_TE_MSA_abc, begin = "Defined Parameters", end = NULL, shift = 1)
hat sich nichts am Output geändert. Wir haben ja auch nur Labels vergeben und neu definierte Parameter hinzugefügt, die allerdings, wie zuvor schon erwähnt, die Modellstruktur (und somit auch die $df$) nicht tangieren.
Die totalen und die indirekten Effekte müssten in einer wissenschaftlichen Untersuchung erneut mithilfe von Bootstrapping inferenzstatistisch geprüft werden. Diesen Schritt lassen wir an dieser Stelle weg.
Invarianzstufen
Mit Invarianz meinen wir die Gleichheit von Parametern über Gruppen hinweg, also bspw., dass es keine Unterschiede über das Geschlecht hinweg gibt. Welche Stufen der Invarianz es gibt, was diese bedeuten und wie wir diese spezifizieren, gucken wir uns im Folgenden an.
So wie wir die indirekten Effekte bestimmt und die Koeffizienten für beide Gruppen benannt haben, lassen sich auch Invarianzen händisch prüfen. Wenn zwei Koeffizienten das selbe Label tragen, werden diese Parameter in den Gruppen auf den gleichen Wert gesetzt. Wir könnten nun für die jeweiligen Invarianzstufen die Parameter händisch gleichsetzen. Dieses ganze Prozedere erscheint recht aufwendig. Allerdings kann so in jedem Schritt überprüft werden, dass die Parameter richtig restringiert wurden. Auch lassen sich so auch leicht partielle Invarianz einbauen, in welchen bspw. nicht alle Faktorladungen über die Gruppen hinweg gleich sind. Außerdem könnten Invarianzen nur für bestimmte Variablen angenommen werden. Diesen kompletten, händischen Prozess sehen Sie in Appendix B. Glücklicherweise enthält das lavaan-Paket aber Möglichkeiten, Invarianzen global zu definieren. Dazu müssen wir lediglich in der Schätzung unserer Modelle in sem das Zusatzargument group.equal spezifizieren. Für partielle Invarianzen gibt es zusätzlich group.partial. Bevor wir mit den Analysen beginnen, sehen Sie in der folgenden Tabelle noch einmal eine Übersicht über die Invarianzstufen. Eine detaillierte Wiederholung dessen, was auch in den inhaltlichen Sitzungen zu den Invarianzstufen behandelt wurde, finden Sie im Appendix D. Die beiden Spalten "Annahme" und "Implikation" sind kumulativ: Invarianzstufen, die weiter unten stehen, enthalten immer auch alle vorherigen Annahmen und erlauben auch immer alle vorherigen Aussagen. Die jeweiligen Einträge einer Zeile sind lediglich für diese Stufe zusätzlich.
Invarianzstufe | Annahme | Implikation
---- | ------- | --------
konfigural | gleiche Modellstruktur | gleiche Konstruktdefinition
metrisch (schwach) | gleiche Faktorladungen | latente Variablen haben gleiche Bedeutung; Beziehungen zwischen latenten Variablen vergleichbar
skalar (stark) | gleiche Interzepte | mittlere Gruppenunterschiede in manifesten Variablen auf Unterschiede in latenten Mittelwerten zurückführbar; latente Mittelwerte vergleichbar
strikt | gleiche Residualvarianzen | Varianzunterschiede in manifesten Variablen auf Varianzunterschiede in latenten Varianzen zurückführbar
Probieren wir dies doch gleich einmal aus (das Modell sollte hierzu keine Parameterbenennungen haben, da diese die gloablen Invarianzeinstellungen in sem überschreiben könnten):
model_sem <- '
# Messmodelle
ZD =~ zd1 + zd2 + zd6
BOEE =~ bo1 + bo6 + bo12 + bo19
# Strukturmodell
BOEE ~ ZD
BFs ~ BOEE + ZD
'
Um BFs hier wie eine latente Variable zu behandeln, müssen wir bestimmen, dass das Interzept und die Residualvarianz nicht mit den manifesten Variablen zusammen gleichgesetzt werden, sondern erst mit den latenten Variablen über die Gruppen restringiert werden (bei der Testung der vollständigen Invarianz). Dazu müssen wir zusätzlich die partielle Invarianzeinstellung verwenden: group.partial = c("BFs ~ 1", "BFs ~~BFs"). Hiermit wird bestimmt, welche Koeffizienten nicht von den Invarianzeinstellungen betroffen sein sollen. Auch wenn diese Einstellungen erst bei der skalaren/starken Invarianz ("BFs~1") und bei der strikten Invarianz ("BFs~~BFs") zum tragen kommen, stellen wir diese auch beim konfigural-invarianten und beim metrisch-invarianten (schwach-invarianten) Modell mit ein. Fangen wir mit dem Fitten des konfigural-invarianten Modells an.
Konfigurale Invarianz
Bei der konfiguralen Invarianz geht es darum, dass in beiden Gruppen die gleichen Modelle aufgestellt werden. Gilt diese Annahme bereits nicht, so macht es keinen Sinn, das Modell weiter einzuschränken und Parameter über die Gruppen zu restringieren. Glücklicherweise passt das Modell zu den Daten, in welchem das Modell für das Geschlecht jeweils geschätzt wurde. Hier schauen wir uns dies noch einmal zur Wiederholung und zum Umbenennen des geschätzten Modells an und spezifizieren mit group.equal = c(""), dass keine Parameter über die Gruppen als identisch angenommen werden sollen:
fit_sem_sex_konfigural <- sem(model_sem, data = StressAtWork, group = "sex",
group.equal = c(""), group.partial = c("BFs ~ 1", "BFs ~~*BFs"))
summary(fit_sem_sex_konfigural, fit.measures = T)
Dem Modell-Fit Teil der Summary entnehmen wir, dass das Modell gut zu den Daten passt:
abbrev(X = fit_sem_sex_konfigural, begin = "Model Test User Model", end = "Parameter Estimates", fit.measures = T)
Der $\chi^2$-Wert ist nicht signifikant und auch die Fit-Indizes CFI, TLI, RMSEA und SRMR sind unauffällig und deuten auf guten Modell-Fit hin. Dies bedeutet, dass wir frohen Mutes das Modell einschränken können, um zu prüfen, welche Invarianz über das Geschlecht hinweg gilt.
Metrische Invarianz
Unter metrischer oder schwacher Invarianz verstehen wir, dass die Faktorladungen ($\lambda$, bzw. $\Lambda$) über die Gruppen hinweg gleich sind. Somit ist der Anteil jedes Items, der auf die latenten Variablen zurückzuführen ist, über die Gruppen hinweg gleichen. Dies ist wichtig, um zu prüfen, ob die Konstrukte über die beiden Gruppen hinweg die gleiche Bedeutung haben. Wir erreichen dies, indem wir group.equal = c("loadings") spezifizieren.
fit_sem_sex_metrisch <- sem(model_sem, data = StressAtWork, group = "sex",
group.equal = c("loadings"), group.partial = c("BFs ~ 1", "BFs ~~BFs"))
summary(fit_sem_sex_metrisch, fit.measures = T)
Wir entnehmen,
abbrev(X = fit_sem_sex_metrisch, begin = "Model Test User Model", end = "Parameter Estimates", fit.measures = T)
dass das Modell immer noch gut zu den Daten passt. Die Frage ist nur, ob das metrisch-invariante Modell nicht doch vielleicht signifikant schlechter zu den Daten passt als das konfigural-invariante Modell. Bevor wir dieser Frage nachgehen, schauen wir uns noch schnell an, wie Parameter hier per Default benannt werden:
abbrev(X = fit_sem_sex_metrisch, begin = "Group 1", end = "Intercepts")
abbrev(X = fit_sem_sex_metrisch, begin = "Group 2", end = "Intercepts")
Wir erkennen, dass "einfach" nur die Parameter durchnummeriert werden, wobei Parameter, die auf 1 restringiert sind, mitgezählt werden, aber nicht ihr eigenes Label erhalten. So heißt $\lambda_{21}^x$, der Ladungskoeffizient von zd2 hier .p2., wobei das p für Parameter steht und die Punkte andeuten, dass es sich hierbei um ein intern vergebenes (also ein durch die Funktion selbst vergebenes) Label handelt. Wollen wir nun wissen, ob sich die Modelle statistisch signifikant von einander underscheiden, können wir wieder den Likelihood-Ratio-Test ($\chi^2$-Differenzentest) heranziehen.
lavTestLRT(fit_sem_sex_metrisch, fit_sem_sex_konfigural)
res_metrisch <- lavTestLRT(fit_sem_sex_metrisch, fit_sem_sex_konfigural)
print(res_metrisch)
Die $\chi^2$-Differenz liegt bei r round(res_metrisch[[5]][2], 4) bei $\Delta df=$ r res_metrisch[[6]][2] mit dem zugehörigen $p$-Wert von r round(res_metrisch[[7]][2], 4). Somit können wir weiter von metrischer Invarianz ausgehen. Dies bedeutet, dass sich Unterschiede zwischen Frauen auf der latenten Variable in gleicher Weise in den beobachtbaren Variablen niederschlagen, wie sie es bei Männern tun.
Skalare Invarianz
Als nächsten wollen wir prüfen, ob zusätzlich zu den Faktorladungen auch die Interzepte ($\tau$) über die Gruppen hinweg gleich sind (insgesamt also $\lambda$s und $\tau$s gleich über die Gruppen hinweg). Dazu passen wir erneut unser Modell an. Hierbei ist zu beachten, dass wir nicht das Interzept von BFs über die Gruppen hinweg gleichsetzten, da sich die Interzepte auf die manifesten Variablen beziehen, wir BFs hier allerdings wie eine latente Variable behandeln wollen. Dazu haben wir die group.partial = c("BFs ~ 1", "BFs ~~BFs") Einstellungen verwendet. Eine Besonderheit der skalaren Invarianz ist, dass wir sobald wir die Interzepte über die Gruppen hinweg gleichsetzten, die Freiheitsgrade haben, mit welchen wir die latenten Interzepte von ZD und BOEE schätzen können. Dies ist dann eine Art Effektkodierung, wobei der Mittelwert der einen Gruppe auf 0 gesetzt und in der anderen Gruppe dann die Abweichung zu dieser Gruppe mitgeführt wird. Andernfalls würden wir fälschlicherweise Invarianz der latenten Mittelwerte annehmen, was wir hier noch gar nicht prüfen wollen! Wir schauen uns dies im Output an.
fit_sem_sex_skalar <- sem(model_sem, data = StressAtWork, group = "sex",
group.equal = c("loadings", "intercepts"), group.partial = c("BFs ~ 1", "BFs ~~BFs"))
summary(fit_sem_sex_skalar, fit.measures = T)
abbrev(X = fit_sem_sex_skalar, begin = "Group 1", end = "Variances")
abbrev(X = fit_sem_sex_skalar, begin = "Group 2", end = "Variances")
Wir erkennen, dass nun lavaan sogar nur Zahlen zwischen zwei Punkten als Labels vergibt. Diese Nummer enspricht weiterhin der Parameternummer. Hier greift nun tatsächlich die Einstellung "BFs~1" in group.partial. BFs hat zwei unterschiedlichte Interzepte. Bei ZD und BOEE fällt auf, dass diese in Gruppe 1 auf 0 gesetzt sind (ohne Unsicherheit) und in Gruppe 2 hier eine Effektkodierung durchgeführt wurde: hier wurden die Interzepte geschätzt. Unterscheidet sich dieser Interzept nun von 0, so unterscheiden sich die Gruppe in ihren Interzepten ([möglicherweise bedingten] Mittelwerten). Dazu später mehr!
Nun wollen wir untersuchen, ob das metrisch- und das skalar-invariante Modell sich signifikant in der Modellbeschreibung unterscheiden.
lavTestLRT(fit_sem_sex_skalar, fit_sem_sex_metrisch)
res_skalar <- lavTestLRT(fit_sem_sex_skalar, fit_sem_sex_metrisch)
print(res_skalar)
Die $\chi^2$-Differenz ist erneut klein (r round(res_skalar[[5]][2], 3)) und der p-Wert zeigt auch hier an, dass es sich um keine signifikante Verschlechterung des Modells handelt p = r round(res_skalar[[7]][2], 3). Die Null-Hypothese, dass die Interzepte über die Faktorladungen hinaus über das Geschlecht hinweg gleich sind, wird somit nicht verworfen. Dies bedeutet nun, dass Unterschiede im Mittelwert der Items zwischen den beiden Gruppen tatsächlich auch auf Unterschiede der Mittelwerte der latenten Variablen zurückzuführen sind. Das heißt, dass es erst ab dieser Invarianzstufe zulässig ist, Mittelwerte zwischen den Gruppen zu vergleichen.
Strikte Invarianz
Unter strikter Invarianz verstehen wir, dass zusätzlich zu den Faktorladungen und den Interzepten auch die Residualvarianzen ($\theta$) gleich sind (insgesamt also $\lambda$s, $\tau$s und $\theta$s gleich über die Gruppen hinweg). Wir passen entsprechend das Modell an:
fit_sem_sex_strikt <- sem(model_sem, data = StressAtWork, group = "sex",
group.equal = c("loadings", "intercepts", "residuals"), group.partial = c("BFs ~ 1", "BFs ~~BFs"))
Hier greift nun tatsächlich die Einstellung "BFs~~BFs" in group.partial. Wir vergleichen das skalar- und das strikt-invariante Modell hinsichtlich der Modellbeschreibung.
lavTestLRT(fit_sem_sex_strikt, fit_sem_sex_skalar)
res_strikt <- lavTestLRT(fit_sem_sex_strikt, fit_sem_sex_skalar)
print(res_strikt)
Die $\chi^2$-Differenz ist erneut klein (r round(res_strikt[[5]][2], 3)) und der p-Wert zeigt auch hier an, dass es sich um keine signifikante Verschlechterung des Modells handelt p = r round(res_strikt[[7]][2], 3). Die Null-Hypothese, dass die Fehlervarianzen über die Faktorladungen und Interzepte hinaus über das Geschlecht hinweg gleich sind, wird somit nicht verworfen.
Dies bedeutet nun, dass Unterschiede in der Varianz der manifesten Variablen tatsächlich auf Unterschiede in den Varianzen der latenten Variablen zurückzuführen sind. In anderen Worten, wenn wir z.B. beobachten, dass Männer in den beobachtbaren Verhaltensweisen homogener sind als Frauen, können wir bei dieser Varianzstufe davon ausgehen, dass dies daher kommt, dass Männer im Konstrukt ähnlicher sind und nicht nur daher, dass sie z.B. aufgrund der Formulierung der Fragen genauer gemessen werden konnten.
Vollständige Invarianz
Unter vollständiger Invarianz verstehen wir das Gleichsetzten aller Strukturparameter. Hier werden nun alle Varianzen, Residualvarianzen, ungerichtete und gerichtete Effekte des Strukturmodells über die Gruppen hinweg gleichgesetzt (insgesamt also $\lambda$s, $\tau$s, $\theta$s, $\gamma$s, $\beta$s, $\kappa$s, $\phi$s und $\psi$s gleich über die Gruppen hinweg). Wir passen entsprechend das Modell an. Außerdem müssen wir nun das Interzept und die Residualvarianz von BFs invariant zwischen den Gruppen setzen, was wir erreichen, indem wir die group.partial Option rausnehmen.
fit_sem_sex_voll <- sem(model_sem, data = StressAtWork, group = "sex",
group.equal = c("loadings", "intercepts", "residuals",
"means", "lv.variances", "lv.covariances", "regressions"))
Wenn wir nun den Modellvergleich zwischen dem strikt invarianten und dem vollständig invarianten Modell durchführen,
lavTestLRT(fit_sem_sex_voll, fit_sem_sex_strikt)
res_voll <- lavTestLRT(fit_sem_sex_voll, fit_sem_sex_strikt)
print(res_voll)
erhalten wir diesmal eine große $\chi^2$-Differenz (r round(res_voll[[5]][2], 3)) und der p-Wert zeigt an, dass es sich um eine signifikante Verschlechterung des Modells handelt p = r round(res_voll[[7]][2], 3) $< 0.05$, wenn wir die Restriktion der vollständigen Invarianz in das Modell aufnehmen. Die Null-Hypothese, dass alle Parameter im Modell über das Geschlecht hinweg gleich sind, wird somit verworfen. Dies bedeutet also, dass es Geschlechtsunterschiede in den Beziehungen zwischen den latenten Variablen gibt. Wir schauen uns dies an, indem wir uns den Output der Summary des strikt-invarianten Modells ansehen, da sich hier Pfadkoeffizienten, sowie die Mittelwerte und Varianzen der latenten Variablen (bzw. von BFs, was wir als latente Variable mitführen) noch unterscheiden:
summary(fit_sem_sex_strikt)
abbrev(X = fit_sem_sex_strikt, begin = "Group 1", end = "Variances")
abbrev(X = fit_sem_sex_strikt, begin = "Group 2", end = "Variances")
Dem jeweiligen Unterpunkt Regressions in beiden Gruppen entnehmen wir, dass die Pfadkoeffizienten recht ähnlich groß zu sein scheinen. Die Standardfehler der Koeffizienten in beiden Gruppen überlappen sich stark, wenn wir jeweils Estimate $\pm$ Std.Err für einen Koeffizienten rechnen (zur Erinnerung: ein 95% Konfidenzintervall würden wir mit $Est \pm 1.96SE$ erhalten, was noch viel größer wäre und sich diese also "noch stärker" überlappen würde). Schauen wir uns jeweils die Interzepte in den Unterpunkten Intercepts in den beiden Gruppen an, erkennen wir an der Effektkodierung für ZD und BOEE, dass die latenten Interzepte ($\kappa$) in der einen Gruppe auf 0 gesetzt und in der anderen Gruppe frei geschätzt wurden. Das Interzept in Gruppe 2 (den Männern) von Zeitdruck ist signifikant von 0 verschieden, das von emotionaler Erschöpfung nicht. Dies bedeutet, dass sich Männer und Frauen in ihrem latenten Mittelwert von Zeitdruck unterscheiden, da der Mittelwert von Zeitdruck in der Gruppe der Frauen auf 0 gesetzt war und der Mittelwert von Zeitdruck der Männer signifikant von 0 abweicht (Est = r round(parameterEstimates(fit_sem_sex_strikt)[59,7], 3), $p < .05$). Das negative Vorzeichen verrät uns, dass Männer im Durchschnitt weniger Zeitdruck erleben. Auch wenn wir Konfidenzintervalle um die Interzeptschätzung von BFs legen, erhalten wir einen signifikanten Unterschied: die untere Grenze des Konfidenzintervalls in Gruppe 1 liegt bei $2.486-1.960.05 \approx 2.38$ und und die obere Grenze in Gruppe 2 liegt bei $2.206+1.960.069 \approx 2.35$; hier haben wir konservativer "gerundet", um den $\beta$-Fehler zu minimieren; - die Konfidenzintervalle überlappen sich nicht! Diese signifikanten Unterschiede könnte der Grund gewesen sein, dass die vollständige Invarianz verworfen wurde. Um dies genauer zu prüfen, müssten wir sukzessive alle Parameter über die Gruppen gleichsetzen und schauen, für welchen Parameter diese Gleichsetzung zu einer signifikanten Verschlechterung des Modells führt.
Zusammenfassend können wir also nur von strikter Invarianz des Modells über das Geschlecht ausgehen.
Wer davon noch nich genug hat, ist herzlich eingeladen im Appendix C sich die mit der Invarianzidee verwandten Gleichsetzung der Faktorladungen innerhalb einer latenten Variable anzusehen: den Test auf essentielle $\tau$-Äquivalenz. Für weiterführende Literatur, die zum Teil für diesen Abschnitt aufgearbeitet wurde, siehe bspw. Gregorich (2006) - dies ist keine Prüfungsliteratur.
Appendix A {#AppendixA}
Modell-Fit, Stichprobengröße und Fehlspezifikation
Der Likelihood-Ratio-Test ($\chi^2$-Differenzentest) vergleicht die Likelihoods zweier Modelle und somit implizit eigentlich die Kovarianzmatrizen (und Mittelwerte). In Lehrbüchern steht häufig der $\chi^2$-Wert ist stichprobenabhängig und wächst mit der Stichprobengröße, was ebenfalls als Grund für die Fit-Indizes genannt wird. Das ist allerdings nur teilweise richtig, denn der $\chi^2$-Wert ist nur für Modelle stichprobenabhängig, in welchen die $H_0$-Hypothese nicht gilt. In einigen Lehrbüchern steht zudem die Formel für den $\chi^2$-Wert wie folgt:
$$F(\hat{\Sigma}M,S) = \log(\hat{\Sigma}_M)-\log(S)+\text{Spur}\left[S\hat{\Sigma}_M^{-1}\right] - p,$$
wobei $\hat{\Sigma}_M$ die modellimplizierte Kovarianzmatrix und $S$ die Kovarianzmatrix der Daten ist und $p$ die Anzahl an beobachteten Variablen. $\text{Spur}$ bezeichnet hierbei die Summe der Diagonalelemente des jeweiligen Objekts (der resultierenden quadratischen Matrix). Die Null-Hypothese besagt:
$$H_0:S=\Sigma_M$$
Der $\chi^2$-Wert ergibt sie wie folgt:
$$\chi^2:=(n-1)F(\hat{\Sigma}_M,S)$$
Somit wirkt es so, dass der $\chi^2$-Wert zwangsläufig mit der Stichprobengröße wachsen muss. Allerdings ist für wachsendes ($n\to\infty$) $F(\hat{\Sigma}_M,S)\to0$, solange die Null-Hypothese gilt. Dies liegt daran, dass $F(\hat{\Sigma}_M,S)$ gerade die Differenz zwischen den beiden Matrizen quantifiziert und diese Differenz unter der Null-Hypothese im Mittel gegen 0 konvergiert. Diese Differenz geht für steigende Stichprobengröße gegen den Wert 0, wird also kleiner mit steigender Stichprobengröße. Um eine Verteilung als Referenz verwenden zu können (hier: die kritischen Werte der $\chi^2$-Verteilung), ist eine Reskalierung vonnöten. Aus diesem Grund wird $F(\hat{\Sigma}_M,S)$ mit $n-1$ multipliziert und die bekannte $\chi^2$-Verteilung entsteht. Gilt nun eine Alternativ-Hypothese:
$$H_1:S\neq\Sigma_M$$
dann konvergiert $F(\hat{\Sigma}_M,S)$ im Mittel nicht mehr gegen Null; es gilt also ($n\to\infty$) $F(\hat{\Sigma}_M,S)\nrightarrow0$, sondern $F(\hat{\Sigma}_M,S)\to d$, wobei $d>0$ gerade die wahre Differenz zwischen den beiden Modellen quantifiziert. Das bedeutet gleichzeitig, dass für den zugehörige mittlere $\chi^2$-Wert unter $H_1$ gilt: $\chi^2{H_1}\to dn \to \infty$, der $\chi^2$-Wert also mit der Stichprobengröße wächst (da $dn$ gerade proportional zu $n$ wächst)! Wir wollen uns dies an folgendem Modell klar machen:
## Example of a Full LISREL model path diagram with the same number of
## exgenous and endogenous variables:
# Lambda matrices:
LoadingsY <- matrix(c(1,0,
1,0,
0,1,
0,1),4,2, byrow = T)
LoadingsX <- as.matrix(c(1,1,1))
# Phi and Psi matrices:
Phi <- diag(1, 1, 1)
Psi <- diag(2)
# Beta matrix:
Beta <- matrix(0, 2, 2)
Beta[2, 1] <- 1
# Theta matrices:
ManVarX <- diag(1, nrow(LoadingsX), nrow(LoadingsX))
ManVarX[2,1] <- 1; ManVarX[1,2] <- 1
ManVarY <- diag(1, nrow(LoadingsY), nrow(LoadingsY))
# Gamma matrix:
Gamma <- as.matrix(c(1,1))
# Tau matrices:
tauX <- rep(0, nrow(LoadingsX))
tauY <- rep(0, nrow(LoadingsY))
# Alpha and Kappa matrices:
LatInts <- rep(0, 2)
# Combine model:
mod <- lisrelModel(LY = LoadingsY, PS = Psi, BE = Beta, TE = ManVarY, LX = LoadingsX,
PH = Phi, GA = Gamma, TD = ManVarX, TY = tauY, TX = tauX, AL = c(0),
KA = LatInts)
# Plot path diagram:
semPaths(mod, as.expression = c("nodes", "edges"), sizeMan = 5, sizeInt = 3,
sizeLat = 7, label.prop = 1, layout = "tree2", edge.label.cex = 1)
Als Populationsmodell wählen wir das Folgende:
pop_model_H0 <- '
# Messmodelle
Xi1 =~ x1 + 0.7*x2 + 0.6*x3
Eta1 =~ y1 + 0.8*y2
Eta2 =~ y3 + 0.9*y4
# Strukturmodell
Eta1 ~ 0.5*Xi1
Eta2 ~ 0.54*Xi1 + 0.4*Eta1
# Fehlerkovarianzen
x1 ~~ 0.4*x2
'
set.seed(123456)
data <- simulateData(model = pop_model_H0, meanstructure = F, sample.nobs = 200)
Die Werte, die in diesem Modell stehen, symbolisieren die wahren Populationsparameter. Bspw. bedeutet Xi1 =~ x1 + 0.7*x2 + 0.6*x3, dass in der Population gilt: $\lambda_{11}=1,\lambda_{21}=.7$ und $\lambda_{31}=.6$ (wobei $\lambda_{11}=1$ auch der Skalierer ist!). Oder Eta2 ~ 0.54*Xi1 + 0.4*Eta1 steht für: $\eta_2=0.54\xi_1+0.4\eta_1+\zeta_2$ in der Population. x1 ~~ 0.4*x2 symbolisert eine Fehlerkovarianz von 0.4, also $\theta_{21}=.4$.
Wenn ein Modell in dieser Form vorliegt, so kann die simulateData Funktion in lavaan verwendet werden, um diese Modell zu simulieren. Wir übergeben der Funktion dazu das Modell model = pop_model_H0, spezifizieren, dass alle Mittelwerte im Mittel 0 sind meanstructure = F und legen die Stichprobengröße fest sample.nobs = 200. In data liegen nun die simulierten manifesten Variablen (die latenten Variablen werden nicht abgespeichert). Hierbei entscheiden die Kürzel, die wir vergeben (z.B. x1 oder y2) über die Namen in data:
head(data)
print(head(data))
In data liegen nun simulierte Daten mit $n=200$. Wir verwenden das $H_0$ Modell auch, um die Daten zu analysieren (dies ist das Modell von oben ohne jegliche Zahlen, also in dem Format, welches wir bereits kennen!):
model_H0 <- '
# Messmodelle
Xi1 =~ x1 + x2 + x3
Eta1 =~ y1 + y2
Eta2 =~ y3 + y4
# Strukturmodell
Eta1 ~ Xi1
Eta2 ~ Xi1 + Eta1
# Fehlerkovarianzen
x1 ~~ x2
'
Das Pfaddiagramm sieht so aus (hier wurden die Zusatzeinstellungen curve = T, curvePivot = T im semPlot verwenden)
fit_H0 <- sem(model = model_H0, data = data)
semPaths(fit_H0, curve = T, curvePivot = T)
Schätzen wir nun das Modell und gucken uns den den $\chi^2$-Wert an.
fit_H0 <- sem(model = model_H0, data = data)
summary(fit_H0)
lavaan::summary(fit_H0)
Besonders von Interesse sind der $\chi^2$-Wert, die $df$ und der zugehörige $p$-Wert, die wir auch so erhalten:
fitmeasures(fit_H0, c("chisq", 'df', "pvalue"))
Außerdem wollen wir zwei fehlspezifizierte Modell betrachten. Unter model_H1_kov speichern wir ein Modell, welches, bis auf die fehlende Fehlerkovarianz, äquivalent zu model_H0 ist.
model_H1_kov <- '
# Messmodelle
Xi1 =~ x1 + x2 + x3
Eta1 =~ y1 + y2
Eta2 =~ y3 + y4
# Strukturmodell
Eta1 ~ Xi1
Eta2 ~ Xi1 + Eta1
'
semPaths(sem(model_H1_kov, data))
Unter model_H1_Strukspeichern wir ein Modell, welches erneut äquivalent zu model_H0ist, bis auf die fehlende gerichtete Beziehung zwischen $\xi_1$ und $\eta_2$.
model_H1_Struk <- '
# Messmodelle
Xi1 =~ x1 + x2 + x3
Eta1 =~ y1 + y2
Eta2 =~ y3 + y4
# Strukturmodell
Eta1 ~ Xi1
Eta2 ~ Eta1
# Fehlerkovarianzen
x1 ~~ x2
'
semPaths(sem(model_H1_Struk, data))
Hierbei ist die Fehlerkovarianz wegzulassen ein "kleiner" Fehler, während eine vollständige Mediation anzunehmen hier zu einem deutlichen Fehler führen sollte, da in die Fehlervarianz nur zwei Variablen involviert sind, während die gerichtete Beziehung zwischen den beiden latenten Variablen $\xi_1$ und $\eta_2$ mindestens alle manifesten Variabel, die Messungen von $\xi_1$ und $\eta_2$ sind, betrifft. Wir gucken uns den Modellfit für alle drei Modelle an:
fit_H1_kov <- sem(model_H1_kov, data)
fit_H1_Struk <- sem(model_H1_Struk, data)
fitmeasures(fit_H0, c("chisq", 'df', "pvalue"))
fitmeasures(fit_H1_kov, c("chisq", 'df', "pvalue"))
fitmeasures(fit_H1_Struk, c("chisq", 'df', "pvalue"))
fit_H1_kov <- sem(model_H1_kov, data)
fit_H1_Struk <- sem(model_H1_Struk, data)
cat("H0:")
print(round(fitmeasures(fit_H0, c("chisq", 'df', "pvalue")), 3))
cat("H1: Fehlerkovarianz")
print(round(fitmeasures(fit_H1_kov, c("chisq", 'df', "pvalue")), 3))
cat("H1: Vollständige Mediation")
print(round(fitmeasures(fit_H1_Struk, c("chisq", 'df', "pvalue")), 3))
Nun wiederholen wir das ganze für eine größere Stichprobengröße von $n=1000$.
set.seed(123456)
data <- simulateData(model = pop_model_H0, meanstructure = F, sample.nobs = 1000)
fit_H0 <- sem(model = model_H0, data = data)
fit_H1_kov <- sem(model_H1_kov, data)
fit_H1_Struk <- sem(model_H1_Struk, data)
fitmeasures(fit_H0, c("chisq", 'df', "pvalue"))
fitmeasures(fit_H1_kov, c("chisq", 'df', "pvalue"))
fitmeasures(fit_H1_Struk, c("chisq", 'df', "pvalue"))
cat("H0:")
print(round(fitmeasures(fit_H0, c("chisq", 'df', "pvalue")), 3))
cat("H1: Fehlerkovarianz")
print(round(fitmeasures(fit_H1_kov, c("chisq", 'df', "pvalue")), 3))
cat("H1: Vollständige Mediation")
print(round(fitmeasures(fit_H1_Struk, c("chisq", 'df', "pvalue")), 3))
Wir sehen, dass das Weglassen der gerichteten Beziehung zu einem größeren mittleren Fehler führt, also zu einem größeren mittleren $\chi^2$-Wert. Gilt die Null-Hypothese, so sollte der mittlere $\chi^2$-Wert bei der Anzahl der $df$ liegen. Nun wollen wir uns die mittleren $\chi^2$-Werte ansehen für verschiedene $n$. Da diese Simulation länger dauern würde, schauen wir uns nur die Ergebnisse an:
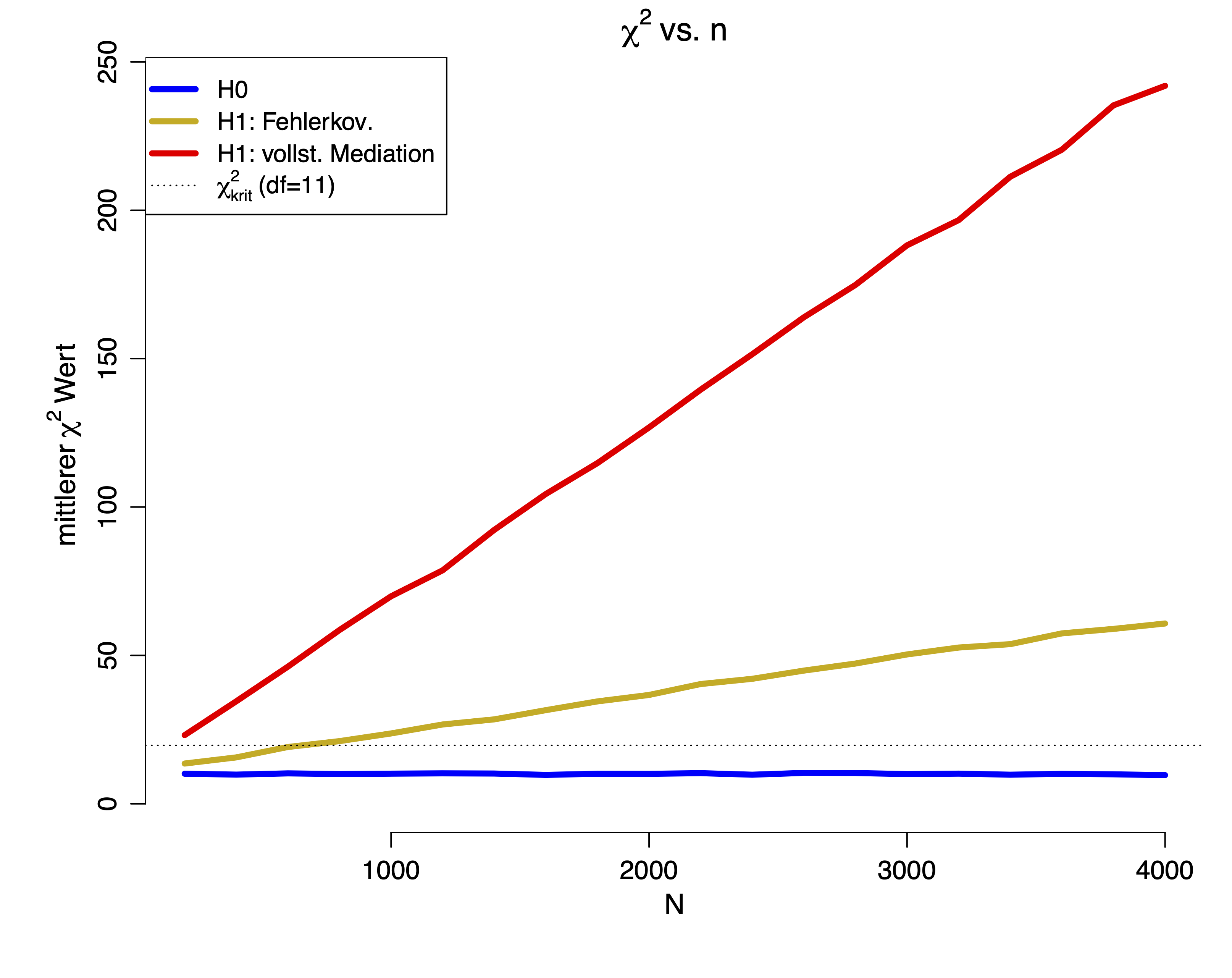
Wir sehen deutlich, dass in beiden $H_1$-Bedingungen der mittlere $\chi^2$-Wert mit der Stichprobengröße wächst. Nur in der $H_0$-Bedingung pendelt sich der mittlere $\chi^2$-Wert gerade bei den $df$ ein. Die gestrichelte Linie repräsentiert den $\chi^2_\text{krit}(df=11)$, somit ist ersichtlich, dass beide $H_1$-Modelle ab einer gewissen Stichprobengröße verworfen werden. Nun ist es aber so, dass in der Wissenschaft Daten häufig nicht perfekt vorliegen, sondern kleine Fehlspezifikationen (also Abweichungen von der Theorie, die aber an sich nicht bedeutsam sind) vorhanden sind. Aus diesem Grund wurden Fit-Indizes entwickelt, welche kleine Fehlspezifikationen relativieren sollen. Ansonsten würde das Verhalten dieses Tests die Wissenschaft dazu bringen kleinere Stichproben zu untersuchen, was allerdings das Aufdecken von Effekten erschwert. Um diesem Dilemma aus dem Weg zu gehen, wird auf die Fit-Indizes zurückgegriffen.
Beispielhaft gucken wir uns nun das Verhalten des $CFI$ und des $RMSEA$ an. Die Definition der Fit-Indizes ist:
$$CFI:= 1- \frac{\max(\chi^2_t-df_t,0)}{\max(\chi^2_t-df_t,\chi^2_i-df_i,0)},$$
wobei die Subskripts $t$ und $i$ für das $target$-Modell, also unser Modell und das $independence$-Modell stehen, welches keine Beziehung zwischen den manifesten Variablen annimmt (das am schlechtesten passende Modell). Einen Ausdruck wie $\max(\chi^2_t-df_t,0)$ bzw. $\max(\chi^2_t-df_t,\chi^2_i-df_i,0)$ oder einfacher $\max(a,0)$ bzw. $\max(a,b,0)$ lesen wir so: hier wird das Maximum zwischen 2 bzw. 3 Ausdrücken bestimmt und damit weitergerechnet; dadurch, dass einer der 2 bzw. 3 Ausdrücke gerade die 0 ist, bedeutet dies, dass dieses Maximum immer größer oder gleich 0 sein wird ($\ge0$).
Der $CFI$ ist ein Vergleich zwischen dem schlechtesten und dem betrachteten Modell. Der mittlere $CFI$ unter der $H_0$-Hypothese sollte bei 1 liegen für große $n$, da für große $n$ der $\chi^2$-Wert im Mittel bei den $df$ liegt und somit $\chi^2_t-df_t=0$, also der Bruch im Mittel bei 0 liegt (somit wird von der 1 im Mittel nichts abgezogen unter der $H_0$). Dies erkennen wir in der Grafik daran, dass im $H_0$-Modell der mittlere $CFI$-Wert gegen 1 geht (dies bedeutet gleichzeitig, dass kleine $CFI$s gerade für einen schlechten Fit sprechen!):

Der $RMSEA$
$$RMSEA:= \sqrt{\max\left(\frac{F(S,\hat{\Sigma}_M)}{df}-\frac{1}{n-1}, 0\right)}.$$
ist die mittlere Abweichung pro Freiheitsgrad kontrolliert für die Stichprobengröße. Der mittlere $RMSEA$ unter der $H_0$-Hypothese sollte bei 0 liegen für große $n$, da für große $n$, $F(S,\hat{\Sigma}_M)$ nahe 0 liegt und damit $\frac{F(S,\hat{\Sigma}_M)}{df}-\frac{1}{n-1} < 0$ also negativ ist. Das Maximum wiederum zwischen einer negativen Zahl und 0 liegt gerade bei 0. In der Grafik erkennen wir dies daran, dass der mittlere $RMSEA$-Wert des $H_0$-Modells gegen 0 geht (dies bedeutet gleichzeitig, dass große $RMSEA$s gerade für einen schlechten Fit sprechen!):

Der $CFI$ sowie der $RMSEA$ pendeln sich für die $H_1$-Modelle gerade bei den "wahren" Abweichungen des Modells unabhängig von der Stichprobengröße ein. Somit ist ersichtlich, dass dies nicht die tatsächlichen Modelle sind, welche den Daten zugrunde liegen, aber zumindest wird quantifiziert, wie stark diese Modelle vom wahren Modell abweichen.
Die gestrichelten Linien geben jeweils die Grenze an, ab welchem Wert nicht mehr von einem "guten" Fit gesprochen werden sollte: $CFI < .97$ und $RMSEA > .05$ (Schermelleh-Engel, Moosbrugger, & Müller, 2003).
Die Abweichungen von 0 bzw. 1 beim $RMSEA$ sowie bei $CFI$ sind allerdings nur für eines der beiden $H_1$ Modelle "extrem". Nur in diesem würde von keinem guten Fit mehr gesprochen werden: nämlich beim Modell, in welchem fälschlicherweise eine vollständige Mediation angenommen wird (keine gerichtete Beziehung zwischen $\xi_1$ und $\eta_2$).
Wann genau die Fit-Indizes für eine deutliche Abweichung zwischen Daten und Modell sprechen, hängt von vielen Dingen ab. Im Artikel von Schermelleh et al. (2003) wurden Cut-Kriterien für spezifische Modelle entwickelt ($CFI < .97$ und $RMSEA > .05$), welche nicht notwendigerweise auf alle anderen Modelle mit unterschiedlicher Komplexität, Stichprobengröße und Anzahl manifester Variablen, etc. übertragbar sind. Diesem Umstand geschuldet wurde das R-Paket ezCutoffs entwickelt, welches simulationsbasierte Cut-Kriterien speziell angepasst an das vorliegende Modell berechnet. Der ezCutoffs-Funktion müssten wir dazu einfach unser angenommenes Modell sowie die Daten übergeben. Schauen wir uns dem Output zu unserem model_sem Modell, in welchem die Mediation von Zeitdruck über emotionale Erschöpfung auf Burnout modelliert wurde, an:
library(ezCutoffs)
ezCutoffs(model = model_sem, data = StressAtWork)
cat("Data Generation
|==================================================| 100% elapsed = 11s ~ 0s
Model Fitting
|==================================================| 100% elapsed = 8s ~ 0s
Empirical fit Cutoff (alpha = 0.05)
chisq 18.444008456 29.49324699
cfi 0.999650351 0.97982435
tli 0.999456102 0.96861566
rmsea 0.008993101 0.04575465
srmr 0.027500985 0.04021870")
Emprircal fit ist hier gerade der Modelfit, den wir auch in unseren Daten beobachtet haben. Der $\chi^2$-Wert von 18.444 kommt uns auch sehr bekannt vor! In der Spalte Cutoff (alpha = 0.05) steht der zugehörige Cut-Off-Wert, ab welchem von keinem guten Fit mehr zu sprechen wäre. Hierbei ist zu beachten, dass für $TLI$ und $CFI$ kleine Werte (also kleiner als der Cut-Off-Wert) für einen schlechten Fit sprechen, während für $RMSEA$ und $SRMR$ große Werte (also größer als der Cut-Off-Wert) für einen schlechten Fit sprechen.
Appendix B {#AppendixB}
MSA zu Fuß
Wir wollen uns die Invarianztestung auch noch einmal zu Fuß ansehen. Um das ganze abzukürzen, schauen wir uns immer mit fitMeasures nur den $\chi^2$-Wert, die $df$ sowie den $p$-Wert an und vergleichen diese mit den bereits geschätzten Modell aus dem Abschnitt zur MSA.
Konfigurale Invarianz
Wir beginnen mit der Spezifikation des konfigural invarianten Modells. Hier muss nichts gleichgesetzt werden. Wir kopieren also einfach das Modell model_sem:
model_sem <- '
# Messmodelle
ZD =~ zd1 + zd2 + zd6
BOEE =~ bo1 + bo6 + bo12 + bo19
# Strukturmodell
BOEE ~ ZD
BFs ~ BOEE + ZD
'
und schätzen dies anschließend. Wir verwenden das Anhängsel 2, um zu zeigen, welches die händisch gleichgesetzte Variante ist:
fit_sem_sex_konfigural <- sem(model_sem, data = StressAtWork, group = "sex",
group.equal = c(""), group.partial = c("BFs ~ 1", "BFs ~~*BFs"))
fit_sem_sex_konfigural2 <- sem(model_sem, data = StressAtWork, group = "sex")
# chi^2, df, p-Wert
fitmeasures(fit_sem_sex_konfigural, c("chisq", 'df', "pvalue"))
fitmeasures(fit_sem_sex_konfigural2, c("chisq", 'df', "pvalue"))
cat("group.equal:")
print(round(fitmeasures(fit_sem_sex_konfigural, c("chisq", 'df', "pvalue")), 3))
cat("zu Fuß/händisch:")
print(round(fitmeasures(fit_sem_sex_konfigural2, c("chisq", 'df', "pvalue")), 3))
Wir erkennen keine Unterschiede, was nicht verwunderlich ist. Hier wurde noch nichts gleichgesetzt.
Metrische Invarianz
Wir müssen nun ein neues Modell spezifizieren, in welchem wir allen Faktorladungen ($\lambda$s) jeweils über die Gruppen hinweg das gleiche Label geben. Hierbei verwenden wir l als Label für die Faktorladungen und nummerieren alle Faktorladungen nacheinander durch (wie genau hier vorgegangen wird, ist immer dem Anwender überlassen. Es muss lediglich darauf geachtet werden, nicht mehrfach das gleiche Label für unterschiedliche Parameter zu verwenden, wenn dies nicht explizit gewünscht ist).
model_sem_metrisch <- '
# Messmodelle
ZD =~ zd1 + c(l1, l1)*zd2 + c(l2, l2)*zd6
BOEE =~ bo1 + c(l3,l3)*bo6 + c(l4, l4)*bo12 + c(l5, l5)*bo19
# Strukturmodell
BOEE ~ ZD
BFs ~ BOEE + ZD'
fit_sem_sex_metrisch <- sem(model_sem, data = StressAtWork, group = "sex",
group.equal = c("loadings"), group.partial = c("BFs ~ 1", "BFs ~~BFs"))
fit_sem_sex_metrisch2 <- sem(model_sem_metrisch, data = StressAtWork, group = "sex")
# chi^2, df, p-Wert
fitmeasures(fit_sem_sex_metrisch, c("chisq", 'df', "pvalue"))
fitmeasures(fit_sem_sex_metrisch2, c("chisq", 'df', "pvalue"))
cat("group.equal:")
print(round(fitmeasures(fit_sem_sex_metrisch, c("chisq", 'df', "pvalue")), 3))
cat("zu Fuß/händisch:")
print(round(fitmeasures(fit_sem_sex_metrisch2, c("chisq", 'df', "pvalue")), 3))
Erneut erkennen wir keine Unterschiede zwischen den Modellen. Beide Vorgehensweisen kommen zum selben Ergenis!
Skalare Invarianz
Erneut müssen wir ein neues Modell spezifizieren, in welchem wir jeweils allen Faktorladungen und Interzepten ($\lambda$s und $\tau$s) über die Gruppen hinweg das gleiche Label geben. Hierbei verwenden wir tau als Label für die Interzepte und nummerieren alle Interzepte der manifesten Variablen nacheinander durch. Wir fügen außerdem die Effektkodierung ein via ZD ~ c(0, NA)*1 und BOEE ~c(0, NA)*1, womit der latente Mittelwert in einer Gruppe auf 0 gesetzt und in der zweiten frei geschätzt wird und erweitern das Modell model_sem_metrisch zu:
model_sem_skalar <- '
# Messmodelle
ZD =~ zd1 + c(l1, l1)*zd2 + c(l2, l2)*zd6
BOEE =~ bo1 + c(l3,l3)*bo6 + c(l4, l4)*bo12 + c(l5, l5)*bo19
zd1 ~ c(tau1, tau1)*1
zd2 ~ c(tau2, tau2)*1
zd6 ~ c(tau3, tau3)*1
bo1 ~ c(tau4, tau4)*1
bo6 ~ c(tau5, tau5)*1
bo12 ~ c(tau6, tau6)*1
bo19 ~ c(tau7, tau7)*1
# Strukturmodell
BOEE ~ ZD
BFs ~ BOEE + ZD
BOEE ~ c(0, NA)*1
ZD ~ c(0, NA)*1
'
fit_sem_sex_skalar <- sem(model_sem, data = StressAtWork, group = "sex",
group.equal = c("loadings", "intercepts"), group.partial = c("BFs ~ 1", "BFs ~~BFs"))
fit_sem_sex_skalar2 <- sem(model_sem_skalar, data = StressAtWork, group = "sex")
# chi^2, df, p-Wert
fitmeasures(fit_sem_sex_skalar, c("chisq", 'df', "pvalue"))
fitmeasures(fit_sem_sex_skalar2, c("chisq", 'df', "pvalue"))
cat("group.equal:")
print(round(fitmeasures(fit_sem_sex_skalar, c("chisq", 'df', "pvalue")), 3))
cat("zu Fuß/händisch:")
print(round(fitmeasures(fit_sem_sex_skalar2, c("chisq", 'df', "pvalue")), 3))
Erneut erkennen wir keine Unterschiede zwischen den Modellen. Beide Vorgehensweisen kommen zum selben Ergenis!
Strikte Invarianz
Wieder müssen wir ein neues Modell spezifizieren, in welchem wir jeweils allen Faktorladungen, Interzepten und Fehlervarianzen ($\lambda$s, $\tau$s und $\theta$s) über die Gruppen hinweg das gleiche Label geben. Hierbei verwenden wir t als Label für die Fehlervarianzen und nummerieren alle Fehlervarianzen der manifesten Variablen nacheinander durch. Wir erweitern das Modell model_sem_skalar.
model_sem_strikt <- '
# Messmodelle
ZD =~ zd1 + c(l1, l1)*zd2 + c(l2, l2)*zd6
BOEE =~ bo1 + c(l3,l3)*bo6 + c(l4, l4)*bo12 + c(l5, l5)*bo19
zd1 ~ c(tau1, tau1)*1
zd2 ~ c(tau2, tau2)*1
zd6 ~ c(tau3, tau3)*1
bo1 ~ c(tau4, tau4)*1
bo6 ~ c(tau5, tau5)*1
bo12 ~ c(tau6, tau6)*1
bo19 ~ c(tau7, tau7)*1
zd1 ~~ c(t1, t1)*zd1
zd2 ~~ c(t2, t2)*zd2
zd6 ~~ c(t3, t3)*zd6
bo1 ~~ c(t4, t4)*bo1
bo6 ~~ c(t5, t5)*bo6
bo12 ~~ c(t6, t6)*bo12
bo19 ~~ c(t7, t7)*bo19
# Strukturmodell
BOEE ~ ZD
BFs ~ BOEE + ZD
BOEE ~ c(0, NA)*1
ZD ~ c(0, NA)*1
'
fit_sem_sex_strikt <- sem(model_sem, data = StressAtWork, group = "sex",
group.equal = c("loadings", "intercepts", "residuals"), group.partial = c("BFs ~ 1", "BFs ~~BFs"))
fit_sem_sex_strikt2 <- sem(model_sem_strikt, data = StressAtWork, group = "sex")
# chi^2, df, p-Wert
fitmeasures(fit_sem_sex_strikt, c("chisq", 'df', "pvalue"))
fitmeasures(fit_sem_sex_strikt2, c("chisq", 'df', "pvalue"))
cat("group.equal:")
print(round(fitmeasures(fit_sem_sex_strikt, c("chisq", 'df', "pvalue")), 3))
cat("zu Fuß/händisch:")
print(round(fitmeasures(fit_sem_sex_strikt2, c("chisq", 'df', "pvalue")), 3))
Erneut erkennen wir keine Unterschiede zwischen den Modellen. Beide Vorgehensweisen kommen zum selben Ergenis!
Vollständige Invarianz
Zum letzten Mal müssen wir ein neues Modell spezifizieren, in welchem wir allen Parametern des Modells (insgesamt also $\lambda$s, $\tau$s, $\theta$s, $\gamma$s, $\beta$s, $\kappa$s, $\phi$s und $\psi$s) über die Gruppen hinweg das gleiche Label geben. Wir müssen die Effektkodierung der latenten Mittelwert wieder herausnehmen und setzten die Mittelwerte in beiden Gruppen auf 0. Als Label verwenden wir wieder die Notation, die wir schon aus den Mediationanalysen kennen (a, b und c). Für die latenten Residualvarianzen führen wir psi als Label ein (hierbei zählen wir weiterhin BFs zu den latenten Variablen). Für die latente Varianz von ZD verwenden wir phi als Label. Für den Mittelwert von BFs verwenden wir ausnahmsweise kappa, da wir BFs, wie zuvor erwähnt, weiter als latente Variable zählen wollen.
model_sem_voll <- '
# Messmodelle
ZD =~ zd1 + c(l1, l1)*zd2 + c(l2, l2)*zd6
BOEE =~ bo1 + c(l3,l3)*bo6 + c(l4, l4)*bo12 + c(l5, l5)*bo19
zd1 ~ c(tau1, tau1)*1
zd2 ~ c(tau2, tau2)*1
zd6 ~ c(tau3, tau3)*1
bo1 ~ c(tau4, tau4)*1
bo6 ~ c(tau5, tau5)*1
bo12 ~ c(tau6, tau6)*1
bo19 ~ c(tau7, tau7)*1
zd1 ~~ c(t1, t1)*zd1
zd2 ~~ c(t2, t2)*zd2
zd6 ~~ c(t3, t3)*zd6
bo1 ~~ c(t4, t4)*bo1
bo6 ~~ c(t5, t5)*bo6
bo12 ~~ c(t6, t6)*bo12
bo19 ~~ c(t7, t7)*bo19
# Strukturmodell
BOEE ~ c(a, a)*ZD
BFs ~ c(b, b)*BOEE + c(c, c)*ZD
BOEE ~ c(0, 0)*1
ZD ~ c(0, 0)*1
BFs ~ c(kappa, kappa)*1
ZD ~~ c(phi, phi)*ZD
BOEE ~~ c(psi1, psi1)*BOEE
BFs ~~ c(psi2, psi2)*BFs
'
fit_sem_sex_voll <- sem(model_sem, data = StressAtWork, group = "sex",
group.equal = c("loadings", "intercepts", "residuals",
"means", "lv.variances", "lv.covariances", "regressions"))
fit_sem_sex_voll2 <- sem(model_sem_voll, data = StressAtWork, group = "sex")
# chi^2, df, p-Wert
fitmeasures(fit_sem_sex_voll, c("chisq", 'df', "pvalue"))
fitmeasures(fit_sem_sex_voll2, c("chisq", 'df', "pvalue"))
cat("group.equal:")
print(round(fitmeasures(fit_sem_sex_voll, c("chisq", 'df', "pvalue")), 3))
cat("zu Fuß/händisch:")
print(round(fitmeasures(fit_sem_sex_voll2, c("chisq", 'df', "pvalue")), 3))
Erneut erkennen wir keine Unterschiede zwischen den Modellen. Beide Vorgehensweisen kommen zum selben Ergenis! Zuvor hatten wir gesehen, dass ein Modellvergleich zwischen der strikten und der vollständigen Invarianz zu Gunsten der strikten Invarianz ausfällt. Somit wird die vollständige Invarianz verworfen und wir entscheiden uns final für die strikte Invarianz über das Geschlecht.
Appendix C {#AppendixC}
Essentielle $\tau$-Äquivalenz
Das Pfadanalysemodell auf Basis von Skalenmittelwerten hatte als Voraussetzung, dass alle Messunger der selben latenten Variable essentiell $\tau$-äquivalent sind, also alle $\lambda$s gleich sind pro latenter Variable. Dies können wir in unserem Modell prüfen, indem wir den Faktorladungen Namen geben, analog zur Bennenung der gerichteten Beziehungen zwischen den latenten Variablen, die wir zur Berechnung des indirekten etc. Effekts gebraucht haben. Das letzte Modell sah so aus:
model_sem_IE_TE <- '
# Messmodelle
ZD =~ zd1 + zd2 + zd6
BOEE =~ bo1 + bo6 + bo12 + bo19
# Strukturmodell
BOEE ~ a*ZD
BFs ~ b*BOEE + c*ZD
# Neue Parameter
IE := a*b
TE := IE + c
'
Wenn wir weiterhin die Defaulteinstellungen der Funktion sem nutzen wollen (1. Faktorladung = 1, etc.), dann müssen wir bei der Bennenung der Faktorladungen etwas aufpassen. Wenn nun alle Faktorladungen gleich sein sollen, so können wir einfach den Präfix 1* vor alle Faktorladungen schreiben. Dies kommt zum äquivalenten Ergebnis wie das Schreiben von bspw. l1* vor alle Messungen der selben latenten Variable (z.B. Zeitdruck): alle Faktorladungen würden auch so per Default auf 1 gesetzt und würden das Label l1 tragen. Das Vergeben von dem Label l1 ist der allgemeinere Ansatz, weswegen wir diesen hier verwenden wollen. Achtung, wenn wir hier die gleichen Labels über die latenten Variablen (auch l1) verwendet hätten, so wären die Faktorladungen gleich mit denen des Zeitdruck, was wir allerdings nicht wollen. Die essentielle $\tau$-Äquivalenzannahme gilt nur pro latenter Variable.
model_sem_IE_TE_tau <- '
# Messmodelle
ZD =~ zd1 + zd2 + zd6
BOEE =~ bo1 + bo6 + bo12 + bo19
# Strukturmodell
BOEE ~ a*ZD
BFs ~ b*BOEE + c*ZD
# Neue Parameter
IE := a*b
TE := IE + c
'
fit_sem_IE_TE_tau <- sem(model_sem_IE_TE_tau, StressAtWork)
summary(fit_sem_IE_TE_tau, fit.measures = T)
model_sem_IE_TE_tau <- '
# Messmodelle
ZD =~ l1*zd1 + l1*zd2 + ...*zd6
BOEE =~ l2*bo1 + ...*bo6 + bo12 + bo19
# Strukturmodell
BOEE ~ a*ZD
BFs ~ b*BOEE + c*ZD
# Neue Parameter
IE := a*b
TE := IE + c
'
fit_sem_IE_TE_tau <- sem(model_sem_IE_TE_tau, StressAtWork)
summary(fit_sem_IE_TE_tau, fit.measures = T)
model_sem_IE_TE_tau <- '
# Messmodelle
ZD =~ l1*zd1 + l1*zd2 + l1*zd6
BOEE =~ l2*bo1 + l2*bo6 + l2*bo12 + l2*bo19
# Strukturmodell
BOEE ~ a*ZD
BFs ~ b*BOEE + c*ZD
# Neue Parameter
IE := a*b
TE := IE + c
'
fit_sem_IE_TE_tau <- sem(model_sem_IE_TE_tau, StressAtWork)
## Skalen bilden
StressAtWork$ZDs <- rowMeans(StressAtWork[,paste0("zd",c(1, 2, 6))])
StressAtWork$BOEEs <- rowMeans(StressAtWork[,paste0("bo",c(1, 6, 12, 19))])
StressAtWork$BFs <- rowMeans(StressAtWork[,paste0("bf",1:20)])
## Modell spezifizieren
model_paths <- '
BOEEs ~ ZDs
BFs ~ BOEEs + ZDs
'
## Modell fitten
fit_paths <- sem(model_paths, data = StressAtWork)
#### SEM:
# Modell
model_sem_IE_TE <- '
# Messmodelle
ZD =~ zd1 + zd2 + zd6
BOEE =~ bo1 + bo6 + bo12 + bo19
# Strukturmodell
BOEE ~ a*ZD
BFs ~ b*BOEE + c*ZD
# Neue Parameter
IE := a*b
TE := IE + c
'
fit_sem_IE_TE <- sem(model_sem_IE_TE, StressAtWork)
## Modell für essentielle tau-Äquvivalenz
model_sem_IE_TE_tau <- '
# Messmodelle
ZD =~ l1*zd1 + l1*zd2 + l1*zd6
BOEE =~ l2*bo1 + l2*bo6 + l2*bo12 + l2*bo19
# Strukturmodell
BOEE ~ a*ZD
BFs ~ b*BOEE + c*ZD
# Neue Parameter
IE := a*b
TE := IE + c
'
fit_sem_IE_TE_tau <- sem(model_sem_IE_TE_tau, StressAtWork)
Sollten Sie alles richtig formuliert haben (und die Zusatzeinstellungen in der summary als fit.measures = T, rsq = T gewählt haben), so sollten Sie folgenden Modellfit erhalten:
abbrev(fit_sem_IE_TE_tau, begin = "Model Test User Model", end = "Parameter Estimates", fit.measures = T, rsq = T)
Sowie folgenden Ergebnisse für die Messmodelle:
abbrev(fit_sem_IE_TE_tau, begin = "Latent Variables", end = "Regressions")
Wir erkennen durch die gleiche Bennenung, dass es sich hier um restringierte Koeffizienten handelt. Auch erkennen wir, dass die Faktorladungen von ZD und BOEE alle gleich heißen und auf 1 gesetzt wurden.
Dem Modellfit ist zu entnehmen, dass das Modell immer noch zu den Daten passt: der $\chi^2$-Wert liegt bei r round(fitmeasures(fit_sem_IE_TE_tau)[3], 3) bei $df=$ r round(fitmeasures(fit_sem_IE_TE_tau)[4], 3) mit zugehörigem $p$-Wert von r round(fitmeasures(fit_sem_IE_TE_tau)[5], 3). Demnach verwerfen unsere Daten das Modell nicht. Außerdem sind die $df$ genau um r round(fitmeasures(fit_sem_IE_TE_tau)[4] - fitmeasures(fit_sem_IE_TE)[4], 3) größer als im unrestringierten Modell (ohne die Annahme der essentiellen $\tau$-Äquivalenz pro latenter Variable). Wir können diese Differrenz leicht prüfen: es mussten 2 Faktorladungen weniger für ZD geschätzt werden und 3 weniger für BOEE - aus diesem Grund unterscheiden sich die $df$ gerade um 5! Da das Modell nicht durch die Daten verworfen wird, müssen wir uns auch nicht unbedingt die Fit-Indizes ansehen. Diese sind hier nämlich auch unauffällig. Ob sie überhaupt im Bezug zu diesem Modell interpretiert werden sollten, wurde im Appendix A näher beleuchtet.
Auch wenn die Daten unser Modell nicht verwerfen, können wir die beiden Modelle mit und ohne essentieller $\tau$-Äquivalenzannahme inferenzstatistisch miteinander vergleichen. Dazu verwenden wir wieder die Funktion lavTestLRT und führen also einen Likelihood-Ratio-Test ($\chi^2$-Differenzentest) durch. Probieren Sie den Modellvergleichstest selbst aus:
## Skalen bilden
StressAtWork$ZDs <- rowMeans(StressAtWork[,paste0("zd",c(1, 2, 6))])
StressAtWork$BOEEs <- rowMeans(StressAtWork[,paste0("bo",c(1, 6, 12, 19))])
StressAtWork$BFs <- rowMeans(StressAtWork[,paste0("bf",1:20)])
## Modell spezifizieren
model_paths <- '
BOEEs ~ ZDs
BFs ~ BOEEs + ZDs
'
## Modell fitten
fit_paths <- sem(model_paths, data = StressAtWork)
#### SEM:
# Modell
model_sem_IE_TE <- '
# Messmodelle
ZD =~ zd1 + zd2 + zd6
BOEE =~ bo1 + bo6 + bo12 + bo19
# Strukturmodell
BOEE ~ a*ZD
BFs ~ b*BOEE + c*ZD
# Neue Parameter
IE := a*b
TE := IE + c
'
fit_sem_IE_TE <- sem(model_sem_IE_TE, StressAtWork)
## Modell für essentielle tau-Äquvivalenz
model_sem_IE_TE_tau <- '
# Messmodelle
ZD =~ (l1)*zd1 + (l1)*zd2 + (l1)*zd6
BOEE =~ (l2)*bo1 + (l2)*bo6 + (l2)*bo12 + (l2)*bo19
# Strukturmodell
BOEE ~ a*ZD
BFs ~ b*BOEE + c*ZD
# Neue Parameter
IE := a*b
TE := IE + c
'
fit_sem_IE_TE_tau <- sem(model_sem_IE_TE_tau, StressAtWork)
# Begin grading:
res <- lavTestLRT(fit_sem_IE_TE_tau, fit_sem_IE_TE) # 2 Faktoren passen besser zu den Daten!
sol2 <- lavTestLRT(fit_sem_IE_TE, fit_sem_IE_TE_tau) # 2 Faktoren passen besser zu den Daten!
grade_result(
fail_if(~ (!identical(.result, res) & !identical(.result, sol2)), 'Der Befehl lavTestLRT muss in diesem Zusammenhang zwei Argumente entgegennehmen: die zwei Objekte der ML-Schätzung unserer SEM-Modelle, welche wir zuvor erstellt haben! Das eine Modell ist jenes mit der Annahme der essentiellen tau-Äquivalenz, das andere ist das unrestringierte Modell. Die Modelle hießen `fit_sem_IE_TE` und `fit_sem_IE_TE_tau`.'),
pass_if(~ identical(.result, res) | identical(.result, sol2)),
correct = 'Sehr gut! Dies ist der Modellvergleich des Modells mit der Annahme der essentiellen tau-Äquivalenz, das andere ist das unrestringierte Modell!',
incorrect = 'Leider falsch.',
glue_correct = '{.correct}',
glue_incorrect = '{.incorrect} {.message}')
resLRT_tau <- lavTestLRT(fit_sem_IE_TE_tau, fit_sem_IE_TE) # 2 Faktoren passen besser zu den Daten!
Dem Modellvergleich ist zu entnehmen, dass das $\tau$-Äquivalenz-Modell signifikant schlechter zu den Daten passt. Der $p$-Wert liegt bei r round(resLRT_tau[[7]][2], 3) und ist somit $<.05$. Auch die Differenz der $df$ lässt sich diesem Output entnehmen. Dies zeigt erneut, dass die Annahme der essentiellen $\tau$-Äquivalenz, auf welcher das Pfadanalysemodell untersucht wurde, auf dem 5% Signifikanzniveau nicht haltbar ist.
Auf die gleiche Art und Weise können wir auch Invarianz über die Zeit oder über Gruppen hinweg untersuchen. Über die Zeit würden wir zwei latente Variablen formulieren, welche einmal $t_1$ und einmal $t_2$ symbolisiert. Die Annahme, dass zu beiden Zeitpunkte die Messmodelle gleich sind, ist tatsächlich in vielen Situation sinnvoll.
Appendix D {#AppendixD}
Invarianzstufen
Die Invarianzstufen sind nach Einschränkungen der Modellparameter sortiert und werden auch (fast) immer in dieser Reihenfolge sukzessive getestet: konfigurale, metrische (schwache), skalare (starke), strikte und vollständige Invarianz. Wir gehen so vor, wie dies per Default im R-Paket lavaan durchgeführt wird. Wir gehen hierzu davon aus, dass die Skalierung für die Varianzen auf den ersten Faktorladungen ($\lambda=1$) liegt und dass die Skalierung für die Mittelwerte (Interzepte) auf dem latenten Mittelwert liegt ($\kappa=0$).
Die erste und am wenigsten restriktive Invarianzstufe ist die konfigurale Invarianz. Unter konfiguraler Invarianz verstehen wir die Gleichheit der Modelle in den Gruppen - oder salopp gesprochen: gilt konfigurale Invarianz, dann gilt in beiden Gruppen die selbe Faktorstruktur (bzw. die gleiche Konstruktdefinition). Die folgende Grafik soll dies für ein einfaches CFA Modell veranschaulichen, welches auch die oben erwähnten Skalierer enthält. In Schwarz dargestellt sind Invarianzen über die Gruppen hinweg (natürlich ist das Modell, das wir hier behandeln, wesentlich komplexer, aber das angegeben Modell stellt die wichtigsten Aspekte der Gleichsetzung dar!):
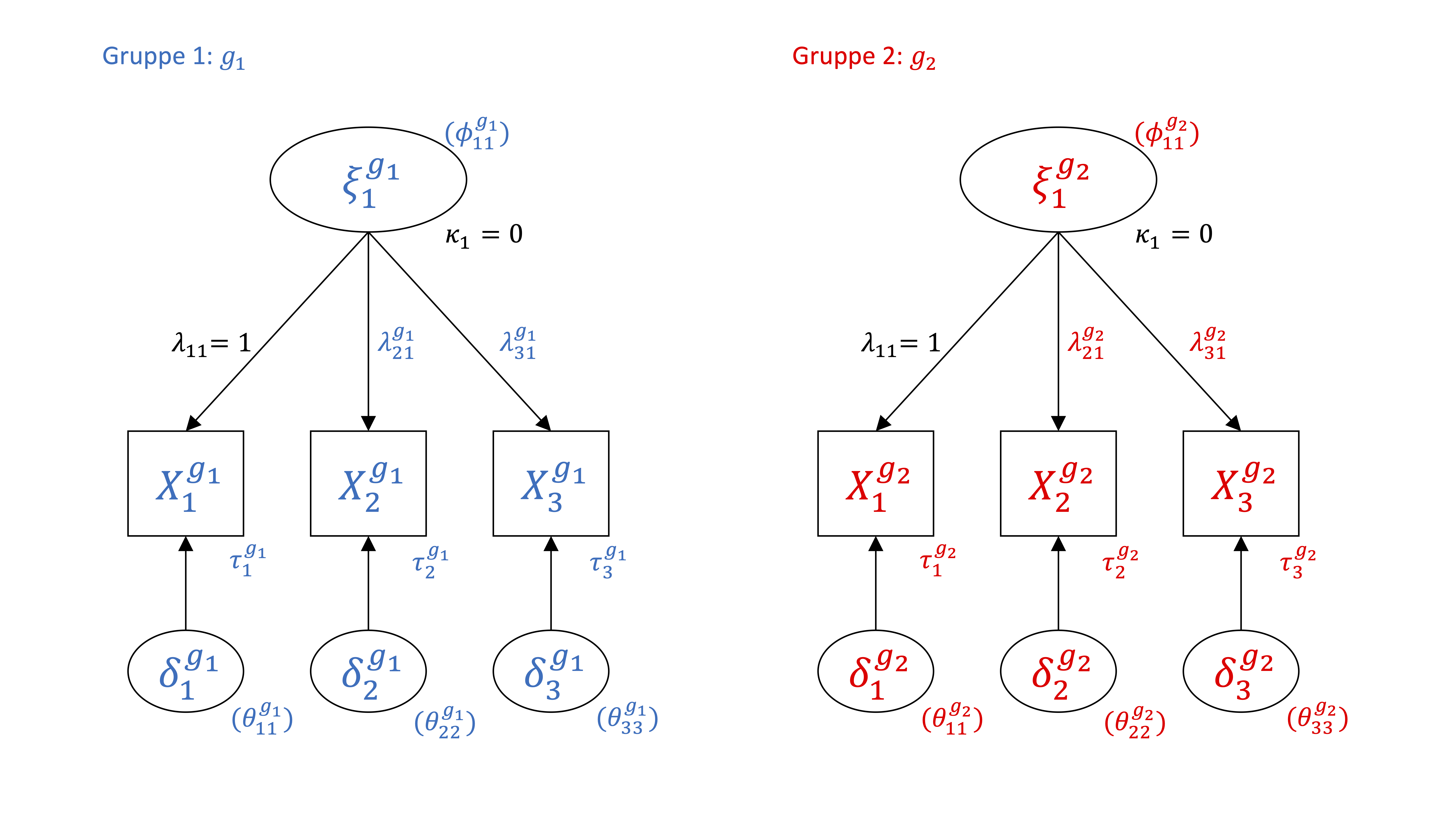
Ob diese Invarianzstufe hält, können wir durch die Modellpassung des Multigruppenmodells aus dem letzten Abschnitt prüfen. Weil wir das Modell aufgrund der sehr guten Passung nicht verwerfen müssen, gibt uns das den Hinweis, dass zumindest die Faktorstruktur in beiden Gruppen gleich ist (als konfigurale Invarianz hält). Wenn wir z.B. durch Fehlpassung feststellen würden, dass in einer der beiden Gruppen eine zusätzliche Residualkorrelation oder eine Querladung aufgenommen werden muss, müssten wir die Annahme der konfiguralen Messinvarianz verwerfen.
Um die weiteren Invarianzstufen und deren Implikationen besser zu verstehen, schauen wir uns noch einmal die modellimplizierte Varianz und die modellimplizierte Erwartung (den modell implizierten Mittelwert) einer manifesten Variable $X$ an. Wir verwenden $^{(g)}$ um Unterschiede zwischen Gruppen darzustellen, wobei dies als Platzhalter für bspw. die Gruppennummer fungiert. Lassen wir dies weg, so gehen wir davon aus, dass der Parameter gleich ist über die Gruppen hinweg. Alle anderen Indizes lassen wir weg, um das ganze übersichtlicher zu gestalten.
Eine einfache modellimplizierte Gleichung einer Messung $X$ ist:
$$X^{(g)}=\tau^{(g)}+\lambda^{(g)}\xi^{(g)}+\delta^{(g)}.$$
$\tau$ ist das Interzept und $\lambda$ ist die Faktorladung. Die Varianz von $X$ ist folglich:
$$\mathbb{V}ar\left[X^{(g)}\right]=\left(\lambda^{(g)}\right)^2\phi^{(g)}+\theta^{(g)}.$$
$\phi$ ist die Varianz der latenten Variable, $\xi$, und $\theta$ ist die Residualvarianz, also die Varianz von $\delta$. Wenn Sie sich nun fragen, wieso wir $\lambda$ hier quadrieren müssen, dann überlegen Sie sich folgendes Beispiel anhand der empirischen Stichprobenvarianz von einer Variable $Y$. Diese berechnen wir so:
$$\hat{\mathbb{V}ar}[Y] = \frac{1}{n}\sum_{i=1}^n(Y_i-\bar{Y})^2,$$
wobei $\bar{Y}$ der Mittelwert von $Y$ ist. Wenn wir nun alle Einträge von $Y$ mit einer Konstanten multiplizieren, also für jede Person $i$ das Produkt $aY_i$ berechnen (z.B. $a=10$) und die Varianz bestimmen (der Mittelwert von $aY$ ist einfach $a\bar{Y}$), dann ergibt sich:
$$\hat{\mathbb{V}ar}[aY] = \frac{1}{n}\sum_{i=1}^n(aY_i-a\bar{Y})^2=\frac{1}{n}\sum_{i=1}^n\big(a(Y_i-a\bar{Y})\big)^2=\frac{1}{n}\sum_{i=1}^na^2(Y_i-\bar{Y})^2=a^2\frac{1}{n}\sum_{i=1}^n(Y_i-\bar{Y})^2=a^2\hat{\mathbb{V}ar}[Y],$$
Da $a$ in der Klammer steht, die quadriert wird, muss natürlich $a$ quadriert werden. Da auch $a^2$ eine Konstante ist, kann sie vor die Summe gezogen werden. Daraus wird dann ersichtlich, dass die Varianz des Produktes einer Variablen mit einer Konstanten gleich der Konstanten zum Quadrat multipliziert mit der Varianz der Variablen ist. Der Mittelwert von $X$ ergibt sich als:
$$\mathbb{E}\left[X^{(g)}\right]=\tau^{(g)}+\lambda^{(g)}\kappa^{(g)},$$
wobei $\kappa$ das Interzept (den Mittelwert) von $\xi$ darstellt. Da $X$ von so vielen Parametern abhängt, ist ohne weitere Annahmen (von Invarianzen der Parameter über die Gruppen hinweg) nicht zu sagen, ob Unterschiede zwischen Gruppen bspw. im Mittelwert von $X$ auf Unterschiede in der latenten Variable oder auf Unterschiede in der Messung zurückzuführen sind. Ähnlich sieht es für die Varianz aus.
Die nächste Invarianzstufe ist die metrische oder schwache Invarianz. Nehmen wir metrische Invarianz an, so gehen wir davon aus, dass die Faktorladungen ($\lambda$) über die Gruppen hinweg gleich sind. Dies impliziert, dass die latenten Variablen über die Gruppen hinweg die gleiche Bedeutung haben. Die latenten Variablen lassen sich also inhaltlich gleich interpretieren und die Beziehungen zwischen ihnen sind vergleichbar. Dies ist sehr wichtig, wenn wir bspw. Fragebögen oder Tests über Gruppen (z.B. Länder oder das Geschlecht) hinweg vergleichen wollen. In der Grafik sind nun alle $\lambda$s schwarz (anstatt blau oder rot):
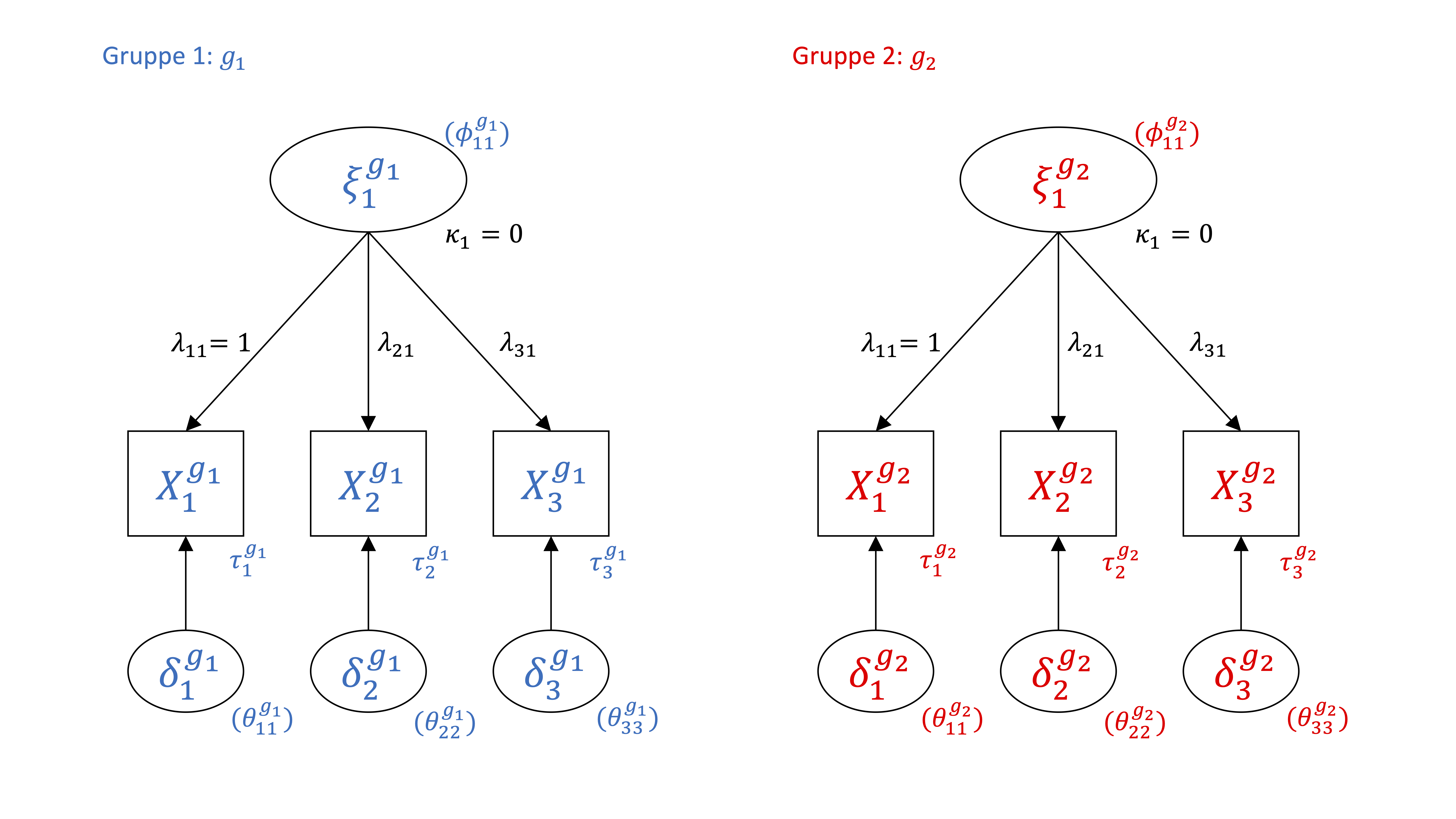
Die Varianz von $X$ vereinfacht sich zu (Weglassen von $^{(g)}$ symbolisiert Gleichheit über die Gruppen hinweg):
$$\mathbb{V}ar\left[X^{(g)}\right]=\lambda^2\phi^{(g)}+\theta^{(g)}.$$
Hierbei ist es so, dass $\lambda^2\phi^{(g)}$ gerade die Systematik oder den wahren Anteil in der Varianz von $X$ beschreibt. Falls metrische Invarianz gilt, so lassen sich also Unterschiede in den wahren Anteilen der Varianzen der Beobachtungen auf Unterschiede in den latenten Varianzen zurückführen (Achtung: dies ist nicht das Gleiche wie Gleichheit der Reliabilitäten, was anteilig die Systematik in $X$ an der gesamten Varianz von $X$ beschreibt - hierfür müssten ebenfalls $\theta$ gleich sein). Dies bedeutet, wenn wir metrische Invarianz nicht verwerfen (also weiter davon ausgehen, dass metrische Invarianz gilt), dass wir durch weitere Modellrestriktionen die latenten Varianzen auf Gleichheit prüfen können. Dies ist nicht zulässig, wenn metrische Invarianz nicht gilt, also die Faktorladungen sich über die Gruppen hinweg unterscheiden. Spezifizieren wir group.equal = c("loadings"), so wird das metrisch-invariante Modell geschätzt.
Unter skalarer oder starker Invarianz verstehen wir die nächste Invarianzstufe, welche zusätzlich zu den Faktorladungen auch noch die Interzepte ($\tau$) als invariant über die Gruppen hinweg annimmt. Der Mittelwert von $X$ vereinfacht sich zu:
$$\mathbb{E}\left[X^{(g)}\right]=\tau+\lambda\kappa^{(g)},$$
wobei nun ersichtlich ist, dass nur noch $\kappa$ über die Gruppen hinweg variiert (wegen $^{(g)}$). Dies bedeutet also, dass unter Annahme der skalaren Invarianz Unterschiede in den Mittelwerten der Beobachtungen auf Unterschiede in den Mittelwerten der latenten Variablen zurückzuführen sind. Um dies zu prüfen, würden überlicherweise über die skalare Invarianz hinaus auch noch $\kappa$ über die Gruppen hinweg gleichgesetzt werden. Der Likelihood-Ratio-Test ($\chi^2$-Differenzentest) zwischen dem skalar-invarianten Modell und dem zusätzlich $\kappa$-invarianten Modell entscheidet dann darüber, ob die latenten Mittelwerte sich über die Gruppen hinweg unterscheiden. Wir schätzen das skalar-invariante Modell durch group.equal = c("loadings", "intercepts"). Da auch für die Mittelwertsstruktur Skalierer gesetzt werden müssen und diese üblicherweise auf den latenten Mittelwerten liegen ($\kappa=0$), muss dies bei der Invarianztestung berücksichtigt werden. Wenn wir die Skalierer auf den latenten Mittelwerten lassen und die Interzept der manifesten Variablen gleichsetzen, so nehmen wir damit auch implizit an, dass die latenten Mittelwerte über die Gruppen hinweg gleich sind. Um diesem Problem aus dem Weg zu gehen, stellt die sem-Funktion klugerweise ein, dass nur in der ersten Gruppe die latenten Mittelwerte/Interzepte ($\kappa$) auf 0 gesetzt werden, alle übrigen werden frei geschätzt. Dies haben wir uns an entsprechender Stelle im Output unserer Modellschäzung angesehen! In der Grafik sind nun alle $\lambda$s und $\tau$s schwarz (anstatt blau oder rot). Außerdem ist die Effektkodierung der $\kappa$s zu sehen: diese bedeutet, dass der erste latente Mittelwert auf 0 restringiert wird, während der zweite frei geschätzt wird und somit die Abweichungen (den Effekt) quantifiziert (beide sind in der Gruppenfarbe dargestellt):
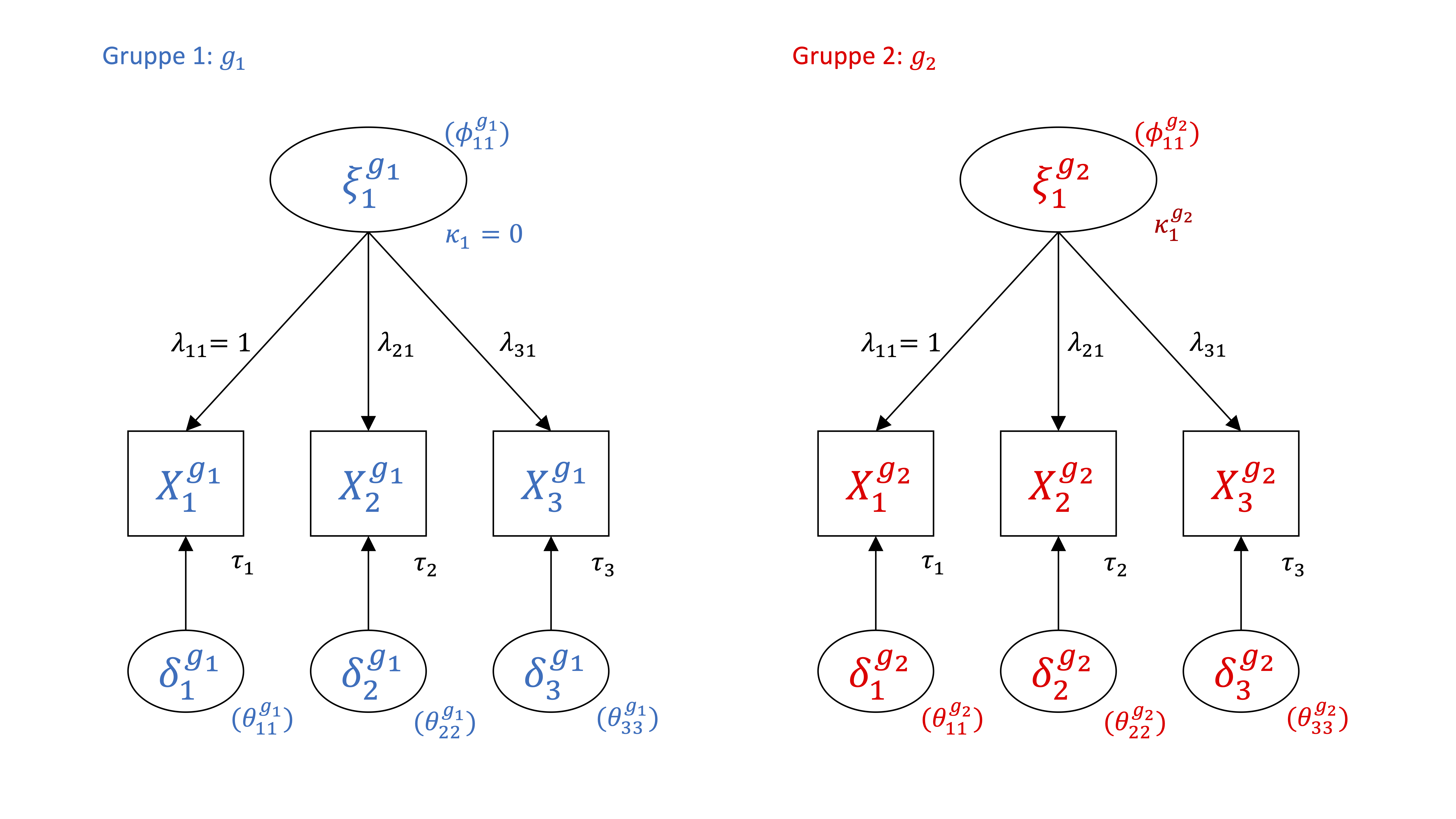
Setzen wir zusätzlich zu Faktorladungen und Interzepten auch noch die Residualvarianzen ($\theta$) gleich über die Gruppen, so sprechen wir von strikter Invarianz. Gilt diese Invarianzbedingung, so lassen sich Unterschiede in den beobachteten Varianzen ausschließlich auf Unterschiede in den latenten Varianzen (also die Varianzen der latenten Variablen) zurückführen. Dies lässt sich der Varianzformel entnehmen:
$$\mathbb{V}ar\left[X^{(g)}\right]=\lambda^2\phi^{(g)}+\theta.$$
Nur noch $\phi$ trägt auf der rechten Seite ein $^{(g)}$. Somit sind nur noch Unterschiede in $\phi$ verantwortlich für Unterschiede in $\mathbb{V}ar\left[X\right]$. In der Grafik sind nun alle $\lambda$s, $\tau$s und $\theta$s schwarz (anstatt blau oder rot):

Wir schätzen das strikt-invariante Modell durch group.equal = c("loadings", "intercepts", "residuals").
Gehen wir von vollständiger Invarianz aus, so sind alle Parameter über die Gruppen hinweg gleich. Es gibt also keinerlei Unterschiede mehr zwischen den Gruppen. Die $\phi$s und auch die $\kappa$s müssen wieder auf den gleichen Wert über die Gruppen hinweg gesetzt werden. Die Variablen selbst ($X$, $\xi$ und $\delta$) bleiben farbig, da wir nur Parameter über die Gruppen hinweg gleichsetzen. Die Grafik der vollständigen Invarianz sieht folgendermaßen aus:
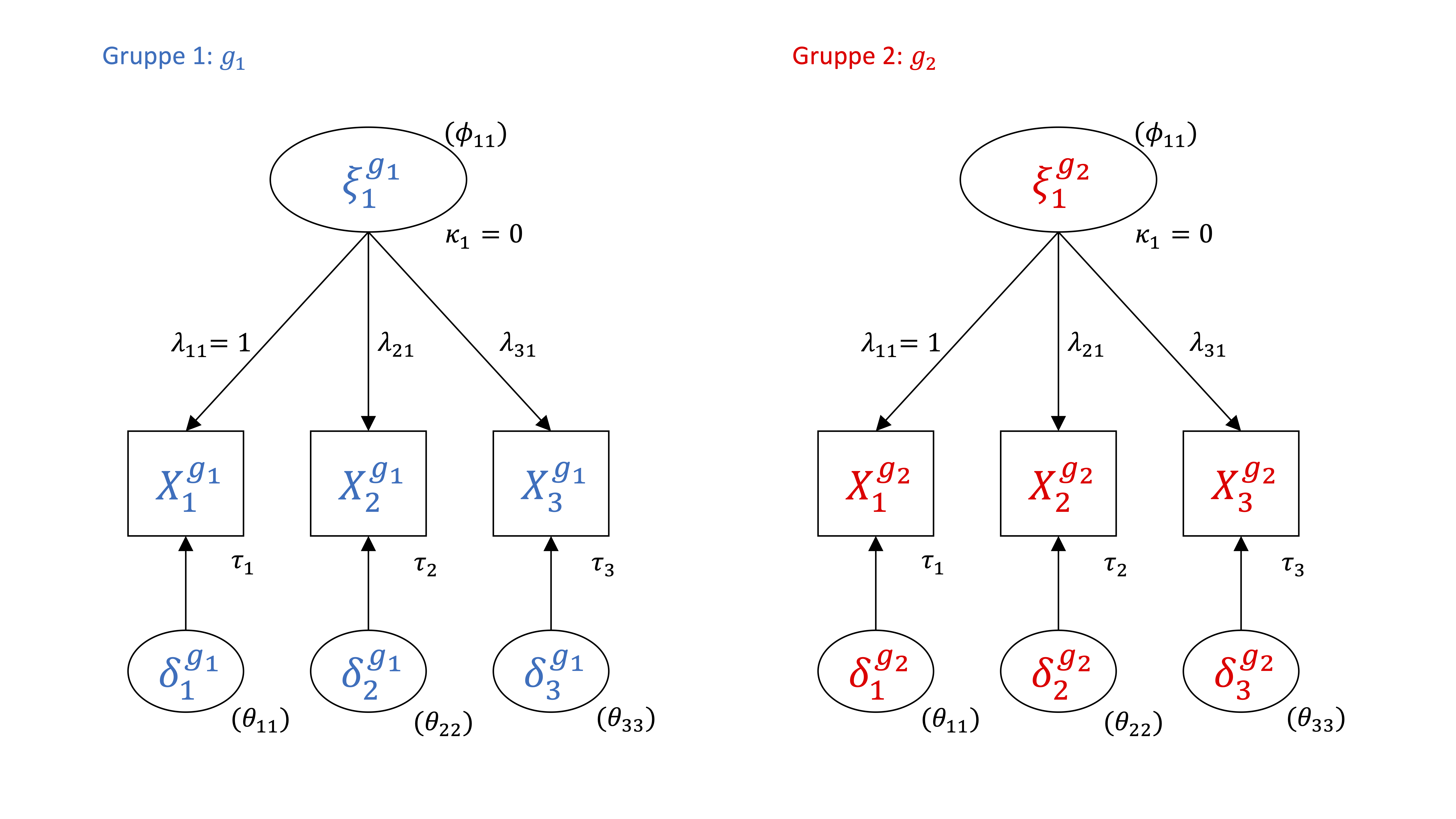
Das vollständig-invariante Modell erhalten wir mit group.equal = c("loadings", "intercepts", "residuals", "means", "lv.variances", "lv.covariances", "regressions"). Hierbei restringiert "means" die latenten Mittelwerte ($\kappa$), "lv.variances" die latenten (Residual-)Varianzen ($\phi$s und $\psi$s exogen und endogen) und "regressions" die Strukturpfadkoeffizienten ($\gamma$s und $\beta$s). Hätten wir Residualkovarianzen bei den manifesten oder latenten Variablen gehabt, so müssten wir diese auch mit "residual.covariances" sowie mit "lv.covariances" restringieren.
Die Invarianzstufen lassen sich nach der Anzahl der zu schätzenden Parameter oder den $df$ sortieren. Im konfigural-invarianten Modell müssen am meisten Parameter geschätzt werden, es besitzt also die wenigsten $df$, während im vollständig-invarianten Modell die wenigsten Parameter geschätzt werden müssen, weswegen dieses auch die meisten $df$ hat. Die anderen Modelle liegen wie folgt dazwischen (je weiter links, desto weniger restriktiv ist das Modell, desto mehr Parameter sind zu schätzen und desto weniger $df$ gibt es):
$$\textbf{konfigural}\quad<\quad\textbf{metrisch/schwach}\quad<\quad\textbf{skalar/stark}\quad<\quad\textbf{strikt}\quad<\quad\textbf{vollständig}.$$
Likelihood-Ratio-Tests ($\chi^2$-Differenzentests) werden herangezogen, um zu prüfen, ob die Invarianzeinschränkungen das Modell signifikant hinsichtlich der Passung zu den Daten verschlechtern.
Literatur
Eid, M., Gollwitzer, M., & Schmitt, M. (2017). Statistik und Forschungsmethoden (5. Auflage, 1. Auflage: 2010). Weinheim: Beltz.
Gregorich, S. E. (2006). Do self-report instruments allow meaningful comparisons across diverse population groups? Testing measurement invariance using the confirmatory factor analysis framework. Medical Care, 44(11), 78-94.
Schermelleh-Engel, K., Moosbrugger, H., & Müller, H. (2003). Evaluation the fit of structural equation models: tests of significance and descriptive goodness-of-fit measures. Methods of Psychological Research Online, 8(2), 23-74.
Inhaltliche Literatur
Büssing, A., & Perrar, K.-M. (1992). Die Messung von Burnout. Untersuchung einer deutschen Fassung des Maslach Burnout Inventory (MBI-D) [The measurement of Burnout. The study of a German version of the Maslach Burnout Inventory (MBI-D)]. Diagnostica, 38, 328 – 353.
Maslach, C., & Jackson, S.E. (1986). Maslach Burnout Inventory (Vol. 2). Palo Alto, CA: Consulting Psychologists Press.
Mohr, G. (1986). Die Erfassung psychischer Befindensbeeinträchti- gungen bei Arbeitern [Assessment of impaired psychological well-being in industrial workers]. Frankfurt am Main, Fermany: Lang.
Semmer, N. K., Zapf, D., & Dunckel, H. (1999). Instrument zur Stressbezogenen Tätigkeitsanalyse (ISTA) [Instrument for stress-oriented task analysis (ISTA)]. In H. Dunkel (Ed.), Handbuch psychologischer Arbeitsanalyseverfahren (pp. 179 – 204). Zürich, Switzerland: vdf Hochschulverlag an der ETH.
- Blau hinterlegte Autorenangaben führen Sie direkt zur universitätsinternen Ressource.
martscht/PsyMSc1 documentation built on July 11, 2020, 3:45 a.m.
library(learnr) # package to generate interactive HTML library(gradethis) # grade Code from input in HTML library(shiny) # nice appearance in R library(fontawesome) # nice fonts library(psych) # EFA durchführen library(lavaan) library(semPlot) knitr::opts_chunk$set(exercise.checker = gradethis::grade_learnr) source('startup.R')
Einleitung zu Pfadanalysen und Strukturgleichungsmodellen (SEM)
Pfadanalysen sind im Grunde genommen mehrere Regressionsanalysen, welche simultan geschätzt werden können. So werden auch mehrere Abhängigkeiten zwischen Variablen berücksichtigt. Strukturgleichungsmodelle kombinieren Pfadanalysen mit Messmodellen. Wir könnten also sagen: "SEM = CFA + Pfadanalyse"!
In dieser Sitzung erweitern wir unsere Kenntnisse mit dem R-Paket lavaan um gerichtete Abhängigkeiten. Möchten Sie die Grundlagen im Umgang damit wiederholen, so empfiehlt es sich die erste Sitzung nochmals anzusehen (PsyMSc1::Sitzung_1()). Auch baut diese Sitzung auf der vergangenen Sitzung zur CFA auf (PsyMSc1::Sitzung_3()).
Bevor wir mit den Analysen beginnen können, laden wir zunächst alle Pakete, welche wir im Folgenden benötigen werden.
library(lavaan) library(semPlot) # grafische Darstellung von Pfadanalyse- und Strukturgleichungsmodellen
Den gesamten R-Code, der in dieser Sitzung genutzt wird, können Sie r fontawesome::fa("download") hier herunterladen.
Datensatz
Der Datensatz StressAtWork, den wir im Folgenden untersuchen wollen, ist eine Zusammenstellung aus mehreren Studien der Arbeits- und Organisationspsychologie- Abteilung der Goethe-Universität, in welchen Call-Center-Mitarbeiter untersucht wurden. Wir können diesen wie gewohnt laden:
data("StressAtWork", package = "PsyMSc1")
Der Datensatz enthält das Geschlecht der Probanden (sex, 1=weiblich, 2=männlich) sowie ausgewählte Messungen der Variablen Zeitdruck (zd1, zd2 und zd6) und Arbeitsorganisationale Probleme (aop3, aop4 und aop8) aus dem Instrument zur stressbezogenen Tätigkeitsanalyse (ISTA) von Semmer, Zapf und Dunckel (1999), Psychosomatische Beschwerden (auch Befindlichkeit: bf1,...,bf20) aus der Psychosomatischen Beschwerdenliste von Mohr (1986) sowie Messungen zu Subskalen von Burnout: Emotionale Erschöpfung (bo1, bo6, b12 und b19) und Leistungserfüllung (bo7, bo8 und bo21) aus Maslachs Burnout-Inventar (Maslach & Jackson, 1986) in der deutschen Übersetzung von Büssing und Perrar (1992).
head(StressAtWork) names(StressAtWork)
Skalenbeschreibungen und Beispielitems
Unter Zeitdruck verstehen wir hier zusätzliche Zeiteinschränkungen, die dazu führen, dass mehr Energie nötig ist, um eine Handlung durchzuführen. Gleichzeitig kann dies auch dazu führen, dass eine Handlung nicht mehr oder nicht mehr rechtzeitig durchführbar ist. Ein Beispielitem ist: "Wie häufig passiert es, dass Sie schneller arbeiten, als sie es normalerweise tun, um die Arbeit zu schaffen?" Die Items wurden auf einer 5-Punkt-Likert Skala beantwortet (Semmer, et al., 1999).
Arbeitsorganisationale Probleme hindern hier bei der Durchführung einer Handlung durch zum Beispiel veraltete Informationen, schlechte Arbeitsgeräte oder organisationale Probleme, die zu einem Mehraufwand führen. Ein Beispielitem ist: "A kann die Arbeitsaufträge gut erledigen, wenn er/sie sich an die vom Betrieb vorgesehenen Wege hält. B kann die Arbeitsaufträge nur bewältigen, wenn er/sie von den vom Betrieb vorgesehenen Wegen abweicht. Welcher der beiden Arbeitsplätze ist Ihrem am ähnlichsten?" Die Items wurden auf einer 5-Punkt-Likert-Skala beantwortet (Semmer, et al., 1999).
Psychosomatische Beschwerden beschreiben körperliche Befindlichenkeiten, die mögliche Langzeitfolgen von Stress sind, wie etwa Schlaflosigkeit, Kopfschmerzen oder Nervosität. Im Gegensatz zu allen anderen Skalen, die hier verwendet werden, handelt es sich bei den psychosomatischen Beschwerden um einen Index, welchem ein formatives Messmodell zu Grunde (im Gegensatz zu reflexiven Messmodellen) liegt (siehe dazu Abschnitt reflexive vs. formative Messmodelle). Ein Beispielitem ist: "Ermüden Sie schnell?" Die Items wurden auf einer 5-Punkt-Likert-Skala beantwortet (Mohr, 1986).
Emotionale Erschöpfung ist hier das Gefühl, erschöpft, niedergeschlagen und frustriert durch die Arbeit zu sein und die Zusammenarbeit mit anderen als besonders anstrengend anzusehen. Ein Beispielitem ist: "Am Ende eines Arbeitstages fühle ich mich verbraucht." Die Items wurden auf einer 7-Punkt-Likert-Skala beantwortet (Maslach & Jackson, 1986).
Wir verstehen unter Leistungserfüllung energetische Gefühle, Dinge anzupacken und das Gefühl, die Möglichkeit zu haben, die eigenen Ambitionen zu erfüllen. Ein Beispielitem ist: "Ich fühle mich sehr tatkräftig." Die Items wurden auf einer 7-Punkt-Likert-Skala beantwortet (Maslach & Jackson, 1986).
Theoretische Grundlage {#Hypothesen}
Pfadanalysen und Strukturgleichungsmodelle gehören, wie auch die CFA, zu den konfirmatorischen, also Theorie bestätigenden, Verfahren und sollen ganz im Popper’schen Sinn durch Vergleiche der Daten mit theoretischen Modellen wissenschaftliche Erkenntnis gewinnen. Wir haben in unseren Daten drei Arten von Variablen: Zeitdruck und Arbeitsorganisationale Probleme sind Stressoren und sollten also mit einer gewissen Wahrscheinlichkeit in einem Individuum zu einer Stressreaktion führen. Emotionale Erschöpfung ist eine Facette von Burnout und gehört somit zu den kurzfristigeren Stressfolgen. Psychosomatische Beschwerden treten unter anderem auf, wenn Stress über einen langen Zeitraum auf ein Individuum einwirkt. Somit können wir postulieren, dass Stress über emotionale Erschöpfung auf psychosomatische Beschwerden wirkt und somit nur einen indirekten Effekt auf die psychosomatischen Beschwerden hat:

Das hier dargestellte schematische Modell postuliert also eine sogenannte vollständige Mediation (dazu mehr im nächsten Abschnitt zur Pfadanalyse) vom Stressor über die kurzfristige (Erschöpfung) auf die langfristigen (Beschwerden) Stressfolgen. Der Einfachheit halber wollen wir diese Hypothesen zunächst nur mit drei Variablen untersuchen: Zeitdruck, emotionale Erschöpfung und psychosomatische Beschwerden.
Pfadanalyse {#Pfadanalyse}
Wir möchten die Hypothesen aus der letzten Sektion zunächst mit Skalenmittelwerten und einer Pfadanalyse untersuchen. Wir wollen außerdem den indirekten Effekt quantifizieren und inferenzstatistisch untersuchen. Dazu müssen wir pro Proband einen Skalenmittelwert pro Variable berechnen. Dazu verwenden wir die bereits in der ersten Sitzung kennengelernte Funktion rowMeans und berechnen so den Mittelwert der psychosomatischen Beschwerden als BFs, den Zeitdruck als ZDs und die emotionale Erschöpfung als BOEEs; das kleine "s" hängen wir an die Skalennamen dran, um zu signialisieren, dass es sich hier um manifeste Skalenmittelwerte handelt:
StressAtWork$ZDs <- rowMeans(StressAtWork[,paste0("zd",c(1, 2, 6))]) StressAtWork$BOEEs <- rowMeans(StressAtWork[,paste0("bo",c(1, 6, 12, 19))]) StressAtWork$BFs <- rowMeans(StressAtWork[,paste0("bf",1:20)])
Hierbei hilft uns paste0 die Schreibweise abzukürzen: wir müssen nicht alle Itemnamen einzeln tippen, sondern die "Strings" werden automatisch erzeugt. Um dies genauer zu verstehen, könnten wir bspw. paste0("zd",c(1, 2, 6)) einmal ausführen. Dies kommt zum gleichen Ergebnis, wie wenn wir c("zd1", "zd2", "zd6") getippt hätten. Das Modell wird nun, ähnlich dem Regressionsmodell aus der ersten Sitzung, aufgestellt. In lavaan werden gerichtete Beziehung zwischen Variablen mit ~ dargestellt, wobei links der Tilde die abhängige Variable (das Kriterium) und rechts der Tilde die unabhängige Variable (der Prädiktor) steht. Für unser angenommenes Modell gibt es folgende Beziehung: Zeitdruck wirkt auf emotionale Erschöpfung und auf psychosomatische Beschwerden. Emotionale Erschöpfung wirkt auf psychosomatische Beschwerden. In diesem Modell wird Zeitdruck die unabhängige Variable genannt, emotionale Erschöpfung ist der Mediator, der die Beziehung der unabhängigen auf die abhängige Variable psychosomatische Beschwerden mediiert. Hier hat Zeitdruck eine direkte Beziehung mit emotionaler Erschöpfung. Emotionale Erschöpfung hat eine direkte Beziehung mit psychosomatischen Beschwerden und Zeitdruck hat eine direkte und eine indirekte (über emotionale Erschöpfung) Beziehung mit psychosomatischen Beschwerden.
Es muss folglich zwei Gleichungen geben: in einer Gleichung ist BOEEs die abhängige Variable und wird durch ZDs vorhergesagt. In der zweiten Gleichung ist BFs die abhängige Variable und wird durch BOEEs und ZDs vorhergesagt. Nun hängt es außerdem von der Schätzfunktion ab, welche weiteren Beziehungen wir in unserem Modell spezifizieren müssen. Wir wollen die sem Funktion verwenden, um das Modell zu schätzen: sem ist, wie cfa (diese kennen Sie aus PsyMSc1::Sitzung_3(), um CFAs zu schätzen) auch eine "Convenience"-Funktion, die gewisse Voreinstellungen verwendet und diese an die lavaan-Funktion, die Sie in der ersten Sitzung kennengelernt hatten, übergibt. Sie hatten damals eine Regression mit lavaan geschätzt und mussten dabei bspw. mit Y ~ 1 + X die Regression anfodern, mit welcher durch X die abhängige Variable Y vorhergesagt wurde. Sie mussten hierbei explizit das Interzept (also den durch X bedingten Mittelwert von Y) sowie die Residualvarianz von Y anfordern via Y ~~ Y (für eine Wiederholung schauen Sie gerne in PsyMSc1::Sitzung_1() vorbei). Wie auch die cfa-Funktion übernimmt die sem Funktion einige dieser Einstellungen für uns. So müssen wir bspw. Residualvarianzen nicht explizit anfragen. Die Mittelwertsstruktur wird in sem per Default nicht mitmodelliert. Wenn wir Mittelwerte betrachten wollen, können wir allerdings der sem-Funktion die Zusatzeinstellung meanstructure = TRUE übergeben. Die Default-Einstellungen für Messmodelle sind identisch mit jenen der Funktion cfa. Wir wiederholen diese, wenn es soweit ist. Somit landen wir bei dieser sehr effizienten Schreibweise für unser Modell:
model_paths <- ' BOEEs ~ ZDs BFs ~ BOEEs + ZDs '
Hätten wir dieses Modell mit lavaan schätzen wollen, so hätten wir folgendes formulieren müssen (wir wollen die Mittelwertsstruktur auch hier ignorieren und lassen daher ~1 weg!):
model_paths_lavaan <- ' BOEEs ~ ZDs BFs ~ BOEEs + ZDs BOEEs ~~ BOEEs BFs ~~ BFs '
Wir können dieses Modell nun schätzen, indem wir die Funktion sem verwenden und ihr unser Modell sowie die Daten übergeben.
Mit summary erhalten wir detaillierte Informationen über Modellfit, Parameterschätzungen und deren Signifikanz.
fit_paths <- sem(model_paths, data = StressAtWork) summary(fit_paths, rsq = T, fit.measures = T)
lavaan::summary(fit_paths, rsq = T, fit.measures = T)
## Skalen bilden StressAtWork$ZDs <- rowMeans(StressAtWork[,paste0("zd",c(1, 2, 6))]) StressAtWork$BOEEs <- rowMeans(StressAtWork[,paste0("bo",c(1, 6, 12, 19))]) StressAtWork$BFs <- rowMeans(StressAtWork[,paste0("bf",1:20)]) ## Modell spezifizieren model_paths <- ' BOEEs ~ ZDs BFs ~ BOEEs + ZDs ' ## Modell fitten fit_paths <- sem(model_paths, data = StressAtWork)
An
abbrev(X = fit_paths, begin = "Model Test User Model", end = "Model Test Baseline Model", fit.measures = T)
erkennen wir, dass es sich hier um das saturierte Modell handelt. Die Korrelationen zwischen den Skalenmittelwerten sind also nur retransformiert, um unser Modell abzubilden. Ein Modellfit-Test ist nicht möglich.
abbrev(X = fit_paths, begin = "User Model versus Baseline Model", end = "Parameter Estimates", fit.measures = T)
Alle Fit-Indizes zeigen perfekten Fit an (CFI = 1, TLI = 1, RMSEA = 0, SRMR = 0).
abbrev(X = fit_paths, begin = "Regressions", end = "Variances", fit.measures = T)
Im Gegensatz zur CFA wird uns nun ein Block in der Summary gezeigt, welcher die Regressionskoeffizienten unseres Modells enthält (ohne Interzept). Hier ist zu erkennen, dass alle Koeffizienten auf dem 5% Niveau signifikant von 0 verschieden sind. Es kann sich also maximal um eine partielle Mediation handeln, da die direkte Beziehung zwischen Zeitdruck und psychsomatischen Beschwerden laut dieser Signifikanzentscheidung mit einer Irrtumswahrscheinlichkeit von 5% auch in der Population besteht und somit der Effekt von Zeitdruck auf die Psychosomatischen Beschwerden nicht vollständig über die emotionale Erschöpfung mediiert wird.
Von einer vollständigen Mediation würden wir sprechen, wenn die direkte Beziehung zwischen Zeitdruck und psychosomatischen Beschwerden (also zwischen unabhängiger und abhängiger Variable) nicht bedeutsam von 0 verschieden ist und der indirekte Effekt von Zeitdruck über emotionale Erschöpfung auf psychsomatische Beschwerden signifikant von Null verschieden ist (dies war unsere Hypothese, die wir im vorherigen Abschnitt postuliert hatten). Für die Population würden wir also bei einer vollständigen Mediation davon ausgehen, dass die unabhängige Variable nur über den Mediator mit der abhängigen Variable zusammenhängt, also jegliche Veränderung in der unabhängigen Variable mit Veränderungen im Mediator zusammenhängt und durch diese Beziehung auch mit der abhängigen Variable kovariiert.
abbrev(X = fit_paths, begin = "R-Square", end = NULL, shift = 1, rsq = T)
Insgsamt ist die Vorhersage der emotionalen Erschöpfung und der psychosomatischen Beschwerden zwar statistisch signifikant, allerdings werden "nur" ca. r round(inspect(fit_paths, "r2")[1], 4)*100% der Variation von BOEEs erklärt sowie ca. r round(inspect(fit_paths, "r2")[2], 4)*100% der Variation von
BFs.
Im nächsten Abschnitt wollen wir unser Modell grafisch veranschaulichen. In der darauf folgenden Sektion wollen wir zusätzlich prüfen, ob der indirekte Effekt von Zeitdruck auf die psychosomatischen Beschwerden signfikant ist.
Grafische Veranschaulichung des Modells und der Ergebnisse
Das Paket semPlot bietet die Möglichkeit, Regressionen, CFAs, Pfadanalysen und Strukturgleichungsmodelle, die bspw. mit lavaan geschätzt wurden, grafisch zu veranschaulichen. Diese Grafiken sind denen, die wir in den inhaltlichen Sitzungen kennen gelernt haben, sehr ähnlich. semPaths (aus dem semPlot-Paket) ist die Funktion, welche wir hierzu nutzen wollen. Sie nimmt als Argument das geschätzte Objekt, welches in unserem Fall die Pfadanalyse enthält, entgegen; hier: fit_paths. Ohne weitere Zusatzeinstellungen sieht dieses so aus:
semPaths(fit_paths)
Der Default stellt also einfach nur das Modell grafisch dar, was sehr praktisch ist, um bspw. zu prüfen, ob alle wichtigen Beziehung im Modell enthalten sind. Gestrichelte Pfeile stehen hierbei für Restriktionen, hier wird also nichts geschätzt: in unserem Bespiel wird die Varianz von ZDs nicht geschätzt, da die Varianz der unabhängigen Variable nur implizit in die Regressionskoeffizenten einfließt, aber kein Koeffizient des Modells darstellen. Würden wir wollen, dass die Varianz von ZDs geschätzt wird, so könnten wir prinzipiell ZDs ~~ ZDs in unser Modell mit aufnehmen.
Gerichtete Pfeile sind regressive Beziehung (also Regressionsparameter, Pfadkoeffizienten oder Faktorladungen). Ungerichtete Pfeile stellen Kovarianzen oder Residualvarianzen dar. Hierbei wird immer dann von einer Kovarianz im Gegensatz zu einer Residualkovarianz gesprochen, wenn es sich um eine exogene (unabhängige) Variable handelt. Salopp gesprochen: hier kommen keine gerichteten Pfeile an! Ein weiteres wichtiges Argument, welches semPaths entgegennimmt ist what. Hiermit wird festgelegt, was genau geplottet werden soll. Wählen wir what = "model", so wird das Modell ohne Parameterschätzungen und Einfärbungen grafisch dargestellt - dies ist der Default, welchen wir bereits oben gesehen haben. Wählen wir hingegen what = "est", so werden alle geschätzten Parameter in das Modell eingezeichnet; diese werden auch farblich hinsichtlich ihrere Ausprägung kodiert.
semPaths(fit_paths, what = "est")
Probieren Sie doch selbst einmal aus, was die folgenden Zusatzeinstellungen bewirken:
semPaths(object = fit_paths, what = "model", layout = "tree2", rotation = 2, col = list(man = "skyblue"), edge.label.cex=1, sizeMan = 5)
Sie können die Argumente der Funktion semPaths nachlesen, indem sie ??semPaths in einem neuen R-Studio Fenster ausführen und dann semPlot::semPaths in der Übersicht auswählen oder sie schauen sich die Dokumentation online hier an.
semPaths(object = fit_paths, what = "est", layout = "tree2", rotation = 2, col = list(man = "skyblue"), edge.label.cex=1, sizeMan = 5)
Wenn Sie eine solche Grafik in R-Studio erzeugen (nicht hier im HTML), gibt es viele Möglichkeiten, diese abzuspeichern. Bspw. können Sie mit dev.print(device = pdf, "MeinPlot.pdf") die Grafik als PDF abspeichern und ihr den Namen "MeinPlot.pdf" geben. Die Endung ".pdf" ist hierbei obligatorisch. Eine andere Möglichkeit ist in R-Studio im Grafikfenster auf "Export" zu klicken und dann den Anweisungen zu folgen.
Berechnen und Testen des indirekten Effektes von Zeitdruck über emotionale Erschöpfung auf psychosomatische Beschwerden
Der indirekte Effekt ist der Effekt, der von der unabhängigen Variable über den Mediator auf die abhängige Variable wirkt. Der Effekt der unabhängigen Variable auf den Mediator wird häufig mit a bezeichnet. Der Effekt des Mediators wird häufig b genannt. Der verbleibende direkte Effekte der unabhängigen auf die abhängige Variable wird, sie haben es sich womöglich schon gedacht, mit c bezeichnet. Nach diesem Schema wollen wir auch die Koeffizienten in unserem Modell benennen. Schauen wir uns dazu einmal die Gleichungen an (der Vollständigkeit halber führen wir auch die Interzepts $a_0$ und $c_0$ mit):
$$BOEE_i = a_0 + aZD_i + \varepsilon_{BOEE,i},$$ $$BF_i = c_0 + bBOEE_i + cZD_i + \varepsilon_{BF,i}.$$ Nun haben wir allerdings eine Gleichung für $BOEE_i$, also können wir diese in die Gleichung von $BF_i$ einstetzen und erhalten den indirekten Effekt (IE) als Produkt der Parameter $IE:=ab$; der direkte Effekt (DE) verbleibt $DE:=c$:
$$BF_i = c_0 + b(a_0 + aZD_i + \varepsilon_{BOEE,i}) + cZD_i + \varepsilon_{BF,i} = \underbrace{(c_0 + ba_0)}{\text{Interzept}} + \underbrace{ab}{{\text{IE}}}ZD_i + \underbrace{c}{{\text{DE}}}ZD_i + \underbrace{(b \varepsilon{BOEE,i}+ \varepsilon_{BF,i})}_\text{Residuum}.$$
Der totale Effekt von Zeitdruck auf die psychosomatischen Beschwerden ergibt sich als $TE:=IE + DE = ab+c$. Da wir bisher in lavaan die Mittelwertstruktur nicht mitmodelliert hatten, haben wir $a_0$ und $c_0$ auch noch nicht untersucht. Für weitere Informationen zu Pfadanalysen, direkten, indirekten und totalen Effekten lesen Sie gerne in Eid, Gollwitzer und Schmitt (2017, pp. 952-).
In lavaan Koeffizienten zu bennen, ist ganz einfach. Sie haben es vielleicht schon im Appendix der letzten Sitzung (PsyMSc1::Sitzung_3()) gesehen: der Variable wird der Koeffizientenname gefolgt von dem Multiplikationszeichen * vorangestellt. Also wird die Beziehung zwischen BOEEs und ZDs um das Präfix a* ergänzt zu: BOEEs ~ a*ZDs, usw.:
model_paths_abc <- ' BOEEs ~ a*ZDs BFs ~ b*BOEEs + c*ZDs '
Der Output bleibt nach Schätzen des Modells identisch, allerdings werden die Namen der Koeffizienten im Output mitgeführt. So lässt sich leicht prüfen, ob die Bennungen an den richtigen Stellen gelandet sind:
fit_paths_abc <- sem(model_paths_abc, data = StressAtWork) summary(fit_paths_abc)
abbrev(fit_paths_abc, begin = "Regressions", end = "Variances")
Um nun den indirekten Effekt $ab$ und den totalen Effeket $ab + c$ mit aufzunehmen, können wir diese in das lavaan Modell inkludieren. Diese neu definierten Parameter stellen allerdings keine weiteren Modellparameter dar, es gehen also keine Freiheitsgrade verloren. Die lavaan-Syntax, die hinzukommt, ist :=; das mathematische "definiert als"-Zeichen. Links davon steht der Namen, den wir dem neuen definierten Parameter geben wollen und rechts steht die Funktion der anderen Parameter aus unserem Modell. Beispielsweise können wir den indirekten Effekt wie folgt definieren: IE := a*b; das Produkt der beiden Pfadkoeffizienten, die wir bereits benannt haben.
model_paths_IE_TE <- ' BOEEs ~ a*ZDs BFs ~ b*BOEEs + c*ZDs # Neue Parameter IE := a*b TE := IE + c '
Wenn wir nun das Modell erneut schätzen, erhalten wir einen neuen Teil im Output der Summary, welcher unsere definierten Parameter und deren Standardfehler, sowie die zugehörigen p-Werte enthält.
fit_paths_IE_TE <- sem(model_paths_IE_TE, data = StressAtWork) summary(fit_paths_IE_TE)
abbrev(fit_paths_IE_TE, begin = "Defined Parameters", shift = 1)
Achtung! Leider können wir den angezeigten Standardfehlern und dem zugehörigen p-Wert nicht unbedingt vertrauen, da einige Studien gezeigt haben, dass der indirekte Effekt asymptotisch nicht immer normalverteilt bzw. symmetrisch verteilt ist, weswegen ein einfaches Teilen des Estimates durch SE und der Vergleich mit der $z$-Verteilung (so wie wir dies eigentlich immer tun; also die einfache Parametersignifikanzentscheidung in komplexen Modellen) in diesem Fall nicht sinnvoll erscheint. Aus diesem Grund möchten wir dieser Problematik entgehen, indem wir die Bootstrap-Methode verwenden.
Bootstrapping
Bootstrapping ist das wiederholte Ziehen mit Zurücklegen aus unserer Stichprobe, solange, bis wir eine neue Stichprobe erhalten, die genauso groß ist, wie die ursprünglich Beobachtete. In dieser neuen, gezogenen Stichproben schätzen wir erneut unser Modell und notieren uns den indirekten Effekt. Dieses Vorgehen wiederholen wir sehr häufig (es hat sich irgendwie so ergeben, dass in der Statistik sehr häufig bei ungefähr und ziemlich genau 1000 liegt), sodass wir die Verteilung des indirekten Effektes und den wahren Standardfehler (also die Streuung des indirekten Effekts) annähern können. Dieses Vorgehen sollte, wenn unsere Stichprobe unabhängig gezogene Personen enthält und repräsentativ für die Gesamtpopulation ist, eine gute Schätzung der Verteilung des indirekten Effektes liefern, welcher Schlüsse auf die Gesamtpopulation zulässt. Wir können dann einfach den 2.5-ten und den 97.5-ten Prozentrang dieser Verteilung verwenden und erhalten ein 95%-Konfidenzintervall für den indirekten Effekt (dieses muss nicht immer symmetrisch sein!). Zum Glück müssen wir dies nicht mit Hand programmieren, sondern können einfach unserer Parameterschätzung mit sem die Zusatzargumente se = "boot" und bootstrap = 1000 hinzufügen. Es empfiehlt sich, diese Objekt anschließend anders zu nennen. Wir lassen unsere Phantasie freien Lauf und nennen die neue "gebootstrapte" Schätzung fit_paths_IE_TE_boot. Wenn wir dann auf das geschätze (neue) Objekt die Funktion parameterEstimates mit der Zusatzeinstellung ci = TRUE anwenden, so werden uns alle Parameter inklusive Konfidenzintervall zurückgegeben. Da es sich hierbei um einen Zufallsprozess handelt, werden die Werte bei mehrmaligem Wiederholen (leicht) unterschiedlich sein. Möchten wir das Ergebnis replizierbar machen, so können wir set.seed() verwenden, wir müssen dieser Funktion lediglich eine beliebige natürliche Zahl übergeben (wie wäre es bspw. mit 1234? Wenn Sie die gleiche R-Version haben, sollte der folgende Code zum selben Ergebnis kommen!).
set.seed(1234) fit_paths_IE_TE_boot <- sem(model_paths_IE_TE, data = StressAtWork, se = "boot", bootstrap = 1000) parameterEstimates(fit_paths_IE_TE_boot, ci = TRUE)
load('./data/fit_paths_IE_TE_boot.RData') cat("[...]") print(parameterEstimates(fit_paths_IE_TE_boot, ci = TRUE)[7:8,]) cat("[...]")
Der Output zeigt die Konfidenzintervalle (sowie weitere Informationen) für alle Parameter in unserem Modell sowie für den indirekten und den totalen Effekt. Hierbei steht ci.lower für die untere Grenze und ci.upper für die obere Grenze des Konfidenzintervalls. Die Nummern 7 und 8 am Anfang der Zeilen zeigen an, dass hier insgesamt 8 Zeilen ausgegeben werden, wir uns aber nur die 7. und die 8. ansehen. Zeile 1 bis 6 (hier nicht dargestellt) zeigen den gleichen Output für alle weiteren Parameter im Modell (wir können help("parameterEstimates") für weitere Informationen in R-Studio ausführen, nachdem wir lavaan via library(lavaan) geladen haben). Das "gebootstrapte" Konfidenzintervall des indirekten Effekts erstreckt sich von r round(parameterEstimates(fit_paths_IE_TE_boot, ci = TRUE)[7,9], 3) bis r round(parameterEstimates(fit_paths_IE_TE_boot, ci = TRUE)[7,10], 3) und schließt die 0 somit nicht ein. Dies bedeutet, dass auf dem 5%-Signifikanzniveau der indirekte Effekt in der Population nicht 0 ist.
Dem Output zufolge scheint es also in der Population einen indirekten Effekt zu geben. Der totale Effekt ist auch signifikant. Die zugehörigen Grafiken unterscheiden sich kaum von denen, die wir uns zuvor angesehen hatten. Schauen wir uns die Modellstruktur an, so werden uns allerdings die Parameternamen mit eingezeichnet, was durchaus von Nutzen sein kann. Der indireke oder der totale Effekt sind keine Modellparameter, weswegen sie auch nicht in der Grafik dargestellt werden können.
semPaths(object = fit_paths_IE_TE, what = "model", layout = "tree2", rotation = 2, col = list(man = "skyblue"), edge.label.cex=1)
Die hier durchgeführten Analysen unterliegen leider einigen Einschränkungen. Bspw. wird beim Mitteln zu Skalenwerten davon ausgegangen, dass jede Messung (jede Variable pro Skala) gleichermaßen aus der dahinterliegende latenten Variable besteht. Dies hatten wir in der letzten Sitzung im Anhang unter essentieller $\tau$-Äquivalenz kennengelernt (gleiche $\lambda$s in einem [CFA-] Messmodell). Essentielle $\tau$-Äquivalenz ist eine strenge Annahme, welche wir prüfen müssten, um den Analysen mit Skalenwerten komplett vertrauen zu können. Außerdem werden durch das Mitteln die Messfehler nicht vollständig modeliert, auch wenn die Analysen somit reliabler als Einzelitemanalysen (z.B. hätten wir auch jeweils ein Item pro Skala verwenden können) sind. Hierbei ist es nun so, dass Effekte stochastischer Regressoren konsistent unterschätzt werden, wenn diese messfehlerbehaftet sind. Wären die Variablen messfehlerfrei, würden die Effekte (die Regressionsparameter) größer ausfallen. Leider können wir nicht davon ausgehen, dass unsere beobachteten Variablen messfehlerfrei sind, wenn wir sie mit Fragebögen erheben! Aus diesem Grund ist in der multiplen Regression bspw. auch eine der Voraussetzungen die Messfehlerfreiheit der (stochastischen) Regressoren (siehe Eid, et al., 2017, Kapitel 19.13).
Wir können die Analysen auf die latente Ebene heben und Messmodelle für die latenten Variablen aufstellen, welche dann die unterschiedlichen Messgenauigkeiten pro Messung berücksichtigen und die Beziehung zwischen den latenten Variablen um die Ungenauigkeit der Einzelitems bereinigen.
Strukturgleichungsmodelle
Strukturgleichungsmodelle (SEM) kombinieren Pfadanalysen mit Messmodellen, also CFAs. Wir können mit SEM in unseren Analysen Messfehler berücksichtigen, aber dennoch gerichtete Beziehungen zwischen den latenten Variablen untersuchen. Bevor wir Messmodelle für unsere latenten Variablen aufstellen, müssen wir uns überlegen, ob dies denn für alle Variablen sinnvoll ist.
Reflexive vs. formative Messmodelle {#formvsreflMessmodell}
Wenn es um Messmodelle geht, kann zwischen reflexiven und formativen Messmodellen unterschieden werden. Reflexive Messmodelle sind die Messmodelle, die wir in der letzten Sitzung detailliert besprochen haben. Darin sind Eigenschaften von Personen latente Variable, die sich nicht direkt beobachten lassen, sich aber in Verhalten und Aussagen niederschlagen. "Konservativismus" ist eine Einstellung von Personen (latente Variable), die sich z.B. darin äußert, dass Personen einer Aussage wie "Ein noch so geschulter und kritischer Verstand kann letzten Endes doch keine echte innere Befriedigung geben." eher zustimmen (Item 36 der Machiavellismus-Konservatismus Skala; Cloetta, 2014). Die Annahme ist also, dass die latente Variable ursächlich für die Ausprägung bestimmter beobachtbarer (manifester) Variablen ist. Wie wir in Sitzung 3 gesehen haben, sieht ein entsprechendes Pfaddiagramm so aus:

Die Variable $X_1$ ist also eine abhängige Variable, deren Ausprägung von der Ausprägung der latenten Variable $\xi$ und vom Messfehler $\delta_1$ abhängt: $X_1=\lambda\xi + \delta_1$. Deswegen gehen die Pfeile im Pfaddiagramm von den latenten Variablen (UV) zu den Items (AVs).
Bei formativen Messmodellen ist es so, dass die latente Variable erst "geformt" wird. Sie ist eine Zusammensetzung aus den manifesten (beobachteten) Items, die als die (gewichtete) Summe (oder [gewichteter] Mittelwert) ihrer Items aufgefasst wird. Das zugehörige Pfaddiagramm sieht folgendermaßen aus:

Es können keine Messfehler berücksichtigt werden und die "Kompositvariable" (deshalb auch die Benennung $\mathcal{C}$, engl. composite) ist eine Linearkombination der Items: $\mathcal{C}=X_1+X_2+X_3$ oder $\mathcal{C}=\lambda_1^\mathcal{C}X_1+\lambda_2^\mathcal{C}X_2+\lambda_3^\mathcal{C}X_3$ (also auch: $\mathcal{C}=\frac{X_1+X_2+X_3}{3}$)). Somit wirken sich Unterschiede zwischen Personen in bspw. $X_1$ (und gleichen $X_2$ und $X_3$) auf Unterschiede in $\mathcal{C}$ aus. Es lassen sich also Unterschiede in der latenten Variable auf unterschiedliche Kombinationen von Ausprägungen der Beobachtungen zurückführen. Gerade in den Wirtschaftswissenschaften und der Soziologie sind solche formativen Variablen sehr verbreitet. Ein bekanntes Beispiel ist der Human Development Index, der aus verschiedenen Komponenten wie Lebenserwartung, Schulbildung und Bruttoinlandsprodukt eine Kennzahl für den Entwicklungsstand eines Landes ermittelt. Ein psychologisches Beispiel für ein formatives Messmodell ist die Variable Umgebungsbelastung, welche auch mit dem ISTA erhoben werden kann (Semmer, et al., 1999). Mit dieser Variable sollen Umwelteinflüsse erfasst werden, die die Arbeit erschweren. Dies könnten z.B. schlechte Lichtverhältnisse oder Lärmbelästigung sein, welche durch längeres Auftreten Stress zur Folge haben können. Allerdings ist es in der Regel nicht so, dass an einem Arbeitsplatz immer dann Lärm auftritt, wenn auch die Lichtverhältnisse schlecht sind. Wenn jedoch beides auftritt, so ist die Umgebungsbelastung an diesem Arbeitsplatz besonders ausgeprägt. Demnach müssen beide Aspekte berücksichtigt werden, um eine gute Schätzung für die Umgebungsbelastung eines Arbeitsplatzes zu erhalten. Würden wir von einem reflexiven Messmodell ausgehen, würde eine hohe Ausprägung auf der latenten Variable Umgebungsbelastung auch zu einer erhöhten Antworttendenz auf beiden Items (Licht- und Lärmverhältnisse) führen.
Wem dieses Konzept bekannt vorkommt, hat sich wahrscheinlich an die Unterscheidung zwischen EFA und PCA zurückerinnert: Hier war es so, dass bei der PCA die Hauptkomponenten Linearkombinationen, also gewichtete Summen, der beobachteten Items waren, während bei EFA ein Messmodell mit dahinterliegenden latenten Variablen formuliert wurde und somit Messfehler modeliert werden konnten.
Bei Zeitdurck und emotionaler Erschöpfung handelt es sich um reflexive Messmodelle. Bei psychosomatischen Beschwerden werden unterschiedliche Beschwerden wie bspw. Kopfschmerzen und Rückenschmerzen abgefragt. Demnach handelt es sich bei dieser Variable eher um ein ein formatives Messmodell. Entsprechend könnten wir die Kompositformulierung in lavaan (<~) nutzen, um eine latente Kompositvariable zu erzeugen oder wir behalten der Einfachheit halber unseren Skalenmittelwert BFs bei.
Das Modell
Das Modell in SEM unterscheidet sich im Vergleich zu CFAs: Es wird zwischen Messmodell und Strukturmodell unterschieden. Im Strukturmodell gibt es gerichtete Beziehungen, in welchen zwischen unabhängigen und abhängigen latenten Variablen unterschieden werden kann. Diese werden häufig auch exogene (unabhängige) und endogene (abhängige) latente Variablen genannt. Außerdem gibt es zwei Arten von Messmodellen: ein exogenes und ein endogenes Messmodell. Diese Messmodelle sind quasi CFAs für die exogenen und die endogenen latenten Variablen; spezifizieren also die Beziehungen zwischen den latenten und den manifesten unabhängen bzw. abhängen Variablen. Wir wollen uns die Messmodelle sowie das Strukturmodell unserer Analyse ansehen:
- Exogenes Messmodell $$\begin{pmatrix}ZD_1\ZD_2\ZD_6\end{pmatrix}=\begin{pmatrix}\tau_1^{x}\\tau_2^{x}\\tau_6^{x}\end{pmatrix}+\begin{pmatrix}\lambda_{11}^{x}=1\\lambda_{21}^{x}\\lambda_{31}^{x}\end{pmatrix}\xi_{\text{ZD}} + \begin{pmatrix}\delta_1\\delta_2\\delta_6\end{pmatrix}$$
- Endogenes Messmodell $$\begin{pmatrix}BO_1\BO_6\BO_{12}\BO_{19}\end{pmatrix}=\begin{pmatrix}\tau_1^{y}\\tau_6^{y}\\tau_{12}^{y}\\tau_{19}^{y}\end{pmatrix}+\begin{pmatrix}\lambda_{11}^{y}=1\\lambda_{21}^{y}\\lambda_{31}^{y}\\lambda_{41}^{y}\end{pmatrix}\eta_{\text{BOEE}} + \begin{pmatrix}\varepsilon_1\\varepsilon_6\\varepsilon_{12}\\varepsilon_{19}\end{pmatrix}$$
- Strukturmodell in Matrixnotation (wobei wir $BFs$ als latente Variable mitführen) $$\begin{pmatrix}\eta_\text{BOEE}\BF_s\end{pmatrix}=\begin{pmatrix}\gamma_{11}\\gamma_{21}\end{pmatrix}\xi_\text{ZD} + \begin{pmatrix}0&0\\beta_{21}&0\end{pmatrix}\begin{pmatrix}\eta_\text{BOEE}\BF_s\end{pmatrix} + \begin{pmatrix}\zeta_\text{BOEE}\\zeta_{BF_s}\end{pmatrix}$$
Für die einzelnen abhängigen latenten Variablen erhalten wir also:
$$\eta_\text{BOEE}=\gamma_{11}\xi_\text{ZD} + \zeta_\text{BOEE}$$ und
$$BFs = \gamma_{21}\xi_\text{ZD} + \beta_{21}\eta_\text{BOEE} + \zeta_{BFs}.$$
Durch Einsetzten folgt: $$BFs = \gamma_{21}\xi_\text{ZD} + \beta_{21}\gamma_{11}\xi_\text{ZD}+ \beta_{21}\zeta_\text{BOEE} + \zeta_{BFs}.$$
Wenn wir nun genauer hinschauen, erkennen wir, dass $\gamma_{11}$ dem Pfad $a$, $\beta_{21}$ dem Pfad $b$ und $\gamma_{21}$ dem Pfad $c$ aus dem Mediationspfadmodell entspricht! Entsprechend quantifiziert $\beta_{21}\gamma_{11}$ den indirekten Effekt von $\xi_\text{ZD}$ über $\eta_\text{BOEE}$ auf $BFs$. Wir wollen das Modell model_paths_IE_TE, welches so aussah:
model_paths_IE_TE <- ' BOEEs ~ a*ZDs BFs ~ b*BOEEs + c*ZDs # Neue Parameter IE := a*b TE := IE + c '
so umstellen, dass es nicht mehr die Beziehung von manifesten Skalenwerten, sondern die Beziehungen zwischen latenten Variablen untersucht. Probieren Sie unten aus, ein solches Modell aufzustellen, indem Sie Ihr Wissen um die CFA aus der letzten Sitzung mit dem kombinieren, was gerade in der Pfadaanalyse besprochen wurde. Nennen Sie die latente Variable für Zeitdruck am besten ZD (Items: zd1, zd2 und zd6) und die latente Variable emotionale Erschöpfung BOEE (Items: bo1, bo6, bo12 und bo19; Achtung: ohne "s" - diese Variablen gibt es nämlich im Datensatz - das waren die Skalenmittelwerte für Zeitdruck und emotionale Erschöpfung). Nehmen Sie als Platzhalter der psychosomatischen Beschwerden die Variable BFs in das Modell auf. Inkludieren Sie auch die Berechnung des indirekten und des totalen Effekts mit den gleichen Bezeichnungen. Sie brauchen folglich 2 Messmodelle, eine Regression für die Beziehung zwischen ZD und BOEE, sowie eine Regression für die Beziehung zwischen ZD, BOEE und BFs. Zum Schluss brauchen Sie noch die neu definierten Koeffizienten für den indirekten und den totalen Effekt (vgl. oben). Schätzen Sie anschließend das Modell mit sem und schauen Sie sich die Summary an! (In den Tips finden Sie Schrittweise mehr und mehr Vorgaben dazu, wie dieses Modell aussehen soll)
model_sem_IE_TE <- ' # Messmodelle ... # Strukturmodell ... # Neue Parameter ... ' fit_sem_IE_TE <- ... ...
model_sem_IE_TE <- ' # Messmodelle ZD =~ ... + ... ... # Strukturmodell ... # Neue Parameter ... ' fit_sem_IE_TE <- ...
model_sem_IE_TE <- ' # Messmodelle ZD =~ zd1 + ... ... # Strukturmodell ... # Neue Parameter ... ' fit_sem_IE_TE <- ...
model_sem_IE_TE <- ' # Messmodelle ZD =~ zd1 + ... ... # Strukturmodell BOEE ~ ... BFs ~ ... + ... # Neue Parameter ... ' fit_sem_IE_TE <- ...
model_sem_IE_TE <- ' # Messmodelle ZD =~ zd1 + ... BOEE =~ ... # Strukturmodell BOEE ~ ... BFs ~ ... + ... # Neue Parameter IE := a*b TE := ... ' fit_sem_IE_TE <- ...
model_sem_IE_TE <- ' # Messmodelle ZD =~ zd1 + ... BOEE =~ ... # Strukturmodell BOEE ~ a*ZD BFs ~ ... + ... # Neue Parameter IE := a*b TE := ... ' fit_sem_IE_TE <- sem(...) summary(...)
model_sem_IE_TE <- ' # Messmodelle ZD =~ zd1 + ... BOEE =~ ... # Strukturmodell BOEE ~ a*ZD BFs ~ ... + ... # Neue Parameter IE := a*b TE := ... ' fit_sem_IE_TE <- sem(model_sem_IE_TE, data = StressAtWork) summary(fit_sem_IE_TE)
model_sem_IE_TE <- ' # Messmodelle ZD =~ zd1 + zd2 + zd6 BOEE =~ bo1 + bo6 + bo12 + bo19 # Strukturmodell BOEE ~ a*ZD BFs ~ b*BOEE + c*ZD # Neue Parameter IE := a*b TE := IE + c ' fit_sem_IE_TE <- sem(model_sem_IE_TE, StressAtWork)
## Skalen bilden StressAtWork$ZDs <- rowMeans(StressAtWork[,paste0("zd",c(1, 2, 6))]) StressAtWork$BOEEs <- rowMeans(StressAtWork[,paste0("bo",c(1, 6, 12, 19))]) StressAtWork$BFs <- rowMeans(StressAtWork[,paste0("bf",1:20)]) ## Modell spezifizieren model_paths <- ' BOEEs ~ ZDs BFs ~ BOEEs + ZDs ' ## Modell fitten fit_paths <- sem(model_paths, data = StressAtWork) #### SEM: # Modell model_sem_IE_TE <- ' # Messmodelle ZD =~ zd1 + zd2 + zd6 BOEE =~ bo1 + bo6 + bo12 + bo19 # Strukturmodell BOEE ~ a*ZD BFs ~ b*BOEE + c*ZD # Neue Parameter IE := a*b TE := IE + c ' fit_sem_IE_TE <- sem(model_sem_IE_TE, StressAtWork)
Sollten Sie alles richtig formuliert haben (und die Zusatzeinstellungen in der summary als fit.measures = T, rsq = T gewählt haben), so sollten Sie folgenden Modellfit erhalten:
abbrev(fit_sem_IE_TE, begin = "Model Test User Model", end = "Parameter Estimates", fit.measures = T, rsq = T)
Falls dem so ist - herzlichen Glückwunsch, Sie haben gerade ein vollständiges Strukturgleichungsmodell formuliert und geschätzt! Jetzt bleibt nur noch die leichteste Übung: Ergebnisse korrekte interpretieren.
Dem Modelfit ist zu entnehmen, dass wir, anders als im Beispiel der Pfadanalyse, nun die Passung zwischen Modell und Daten untersuchen können: der $\chi^2$-Wert liegt bei r round(fitmeasures(fit_sem_IE_TE)[3], 3) bei $df=$ r round(fitmeasures(fit_sem_IE_TE)[4], 3) mit zugehörigem $p$-Wert von r round(fitmeasures(fit_sem_IE_TE)[5], 3). Demnach verwerfen unsere Daten das Modell nicht.
Die Fit-Indizes zeigen hier allesamt einen guten Fit des Modells an. Dies war Aufgrund des $\chi^2$-Wertes zu erwarten, da die Stichprobengröße ausreichend Power liefert (sodass das Modell sinnvoll geschätzt werden kann) und der $\chi^2$-Wert trotzdem nicht signifikant auf dem 5%-Niveau war. Es gibt also keinen Grund an der $H_0$ (auf Modellpassung zwischen Daten und Modell) zu zweifeln. Denn nur in solchen Modellen, in denen tatsächlich die $H_1$-Hypothese gilt, wächst der $\chi^2$-Wert mit der Stichprobengröße (dies steht im Widerspruch zu einigen Textbüchern, welche propagieren, dass der $\chi^2$-Wert immer mit der Stichprobengröße wächst!). Es empfiehlt sich sehr über die Beziehung zwischen Stichprobengröße, $\chi^2$-Wert und den Fit-Indizes noch einmal im Appendix A nachzulesen!
Für die Messmodelle ergibt sich Folgendes:
abbrev(fit_sem_IE_TE, begin = "Latent Variables", end = "Regressions")
Diesen entnehmen wir, dass die Variablen auf der latenten Dimension ähnlich stark diskriminieren (ähnlich große Faktorladungen). Wir sehen außerdem, dass die ersten Faktorladungen jeweils auf 1 gesetzt sind und hier entsprechend keine Signifikanzentscheidung vonnöten ist (wir haben den Wert hier "ohne Unsicherheit" vorgegeben). Um die Reliabilität beurteilen zu können, schauen wir uns die erklärten Varianzanateile an:
abbrev(fit_sem_IE_TE, begin = "R-Square", end = "Defined Parameters", rsq = T)
Die beobachteten Variablen zeigen Reliabilitäten zwischen r round(min(inspect(fit_sem_IE_TE, "r2")[1:7]), 3) und r round(max(inspect(fit_sem_IE_TE, "r2")[1:7]), 3).
Nun wollen wir die Ergebnisse mit den Ergebnissen der Pfadanalyse vergleichen. Dazu schauen wir uns die gerichteten Beziehungen zwischen den latenten Variablen an:
abbrev(fit_sem_IE_TE, begin = "Regressions", end = "Variances")
Hier werden uns erneut die Label "(a)", "(b)" und "(c)" angezeigt. Der direkte Effekt (c) von Zeitdruck auf psychosomatische Beschwerden scheint, im Gegensatz zu den Ergebnissen der Pfadanalyse im Abschnitt zuvor, nicht signifikant von 0 verschieden zu sein. Die beiden anderen Pfade sind statistisch signifikant:
Auch für diese Analysen ist der $R^2$-Output interessant:
abbrev(fit_sem_IE_TE, begin = "R-Square", end = "Defined Parameters", rsq = T)
Im Bezug auf die latenten Regressionen enthält der R-Square Output die Anteile erklärter Varianzen von BOEE und BFs. Demnach können r round(inspect(fit_sem_IE_TE, "r2")[8]*100, 2)% der Variation der psychosomatischen Beschwerden durch die emotionale Erschöpfung und Zeitdruck (obwohl die Beziehung nicht signifikant ist) erklärt werden. r round(inspect(fit_sem_IE_TE, "r2")[9]*100, 2)% der Variation der emotionale Erschöpfung werden durch Zeitdruck erklärt. Somit kann etwas mehr Variation erklärt werden als im Pfadanalysefall (dort: r round(inspect(fit_paths_IE_TE, "r2")[2]*100, 2)% der Variation der psychosomatischen Beschwerden durch die emotionale Erschöpfung und Zeitdruck sowie r round(inspect(fit_paths_IE_TE, "r2")[1]*100, 2)% der Variation der emotionale Erschöpfung durch Zeitdruck).
Berechnen und Testen des indirekten Effektes von Zeitdruck über emotionale Erschöpfung auf psychosomatische Beschwerden im SEM
Der indirekte und der totale Effekt liegen bei:
abbrev(fit_sem_IE_TE, begin = "Defined Parameters", end = NULL, shift = 1)
Der indirekte Effekt aus dem Pfadanalysemodell lag bei r round(parameterEstimates(fit_paths_IE_TE)[7,5], 3) und der totale bei r round(parameterEstimates(fit_paths_IE_TE)[8,5], 3). Somit liegen auch diese beiden Werte (deskriptiv gesehen) etwas höher, wenn wir latente Modellierung nutzen! Um den indirekten Effekt auf Signifikanz zu prüfen, benutzten wir wieder die Bootstrap-Methode (set.seed soll die Analyse replizierbar machen; schauen Sie dazu noch einmal im Abschnitt zur Pfadanalyse nach):
set.seed(12345) fit_sem_IE_TE_boot <- sem(model_sem_IE_TE, data = StressAtWork, se = "boot", bootstrap = 1000) parameterEstimates(fit_sem_IE_TE_boot, ci = TRUE)
load('./data/fit_sem_IE_TE_boot.RData') print(parameterEstimates(fit_sem_IE_TE_boot, ci = TRUE)[21:22,])
Die Nummern 21 und 22 am Anfang der Zeilen zeigen an, dass hier insgesamt 22 Zeilen ausgegeben werden, wir uns aber nur die 21. und die 22. ansehen. Auch hier ist zu sehen, dass der indirekte Effekt signifikant von 0 abweicht. Da der direkte Effekt von Zeitdruck auf die psychosomatischen Beschwerden nicht signifikant ist, handelt es sich in diesem Zusammenhang um eine vollständige Mediation. Die vollständige lineare Beziehung zwischen Zeitdruck und psychosomatischen Beschwerden "fließt" über die emotionale Erschöpfung. Die Frage ist nun, warum es hier zu Unterschieden zwischen der Pfadanalyse und SEM kommt. Naiverweise würde wir zunächst erwarten, dass alle Effekte eher signifikant werden, wenn für Messfehler kontrolliert wird. So hatten wir zuvor mit messfehlerbehaften Regressoren in einer Regressionsanalyse argumentiert. Demnach würden wir eher erwarten, dass der direkte Effekt mit SEM signfikant ist. Allerdings haben wir in der Pfadanalyse die Daten unter der Annahme der essentiellen $\tau$-Äquivalenz zusammengefasst: wir haben einfach Mittelwerte ohne bestimmte Gewichtung verwendet. Im SEM haben wir die Faktorladungen frei Schätzen lassen - wir haben also statt eines gleichgewichteten formativen Konstrukts ein reflexives Konstrukt aus den Beziehungen in den Daten schätzen lassen. Zusammenfassend gehen wir also von einer vollständigen Mediation aus.
Grafische Repräsentation
Wir können diese Modelle auch wieder grafisch darstellen. Probieren Sie doch einmal aus, was in diesem Zusammenhang durch die folgendenen weiteren Einstellungen passiert:
semPaths(object = fit_sem_IE_TE, what = "model", layout = "tree2", rotation = 2, curve = T, col = list(man = "skyblue", lat = "yellow"), curvePivot = T, edge.label.cex=1.2, sizeMan = 5, sizeLat = 8)
semPaths(object = fit_sem_IE_TE, what = "est", layout = "tree2", rotation = 2, curve = T, col = list(man = "skyblue", lat = "yellow"), curvePivot = T, edge.label.cex=1.2, sizeMan = 5, sizeLat = 8)
curve = T und curvePivot = T haben in diesem Modell keinen Effekt, da es hierbei um die Kovarianzen geht und wird nur der Vollständigkeit halber aufgeführt. Der Effekt dieser beiden kann im Appendix A der ersten Grafik des $H_0$-Modells entnommen werden: diese Einstellungen bewirken die nicht vollständig runde Kurve der Fehlerkovarianz (durch curvePivot = T), welche vor allem dann die Übersichtlichkeit erhöht, wenn mehrere Kovarianzen vorhanden sind. Mit curve = F würden alle Fehlerkovarianzen und Kovarianzen (wenn vorhanden) als gerade Linien eingezeichnet werden.
BFs wird hier als Viereck dargestellt, da wir hier Werte gemittelt haben und diesen Skalenwert als direkt beobachtbare Variable in das Modell aufgenommen haben.
Multi Sample Analysis {#MSA}
In einer Multi-Sample-Analysis wird in mehreren Gruppen gleichzeitig ein Strukturgleichungsmodell geschätzt. Wir könnten uns bspw. fragen, ob die gleichen Beziehungen, so wie wir sie gerade beobachtet haben, gleichermaßen für Männer und Frauen gelten. Im Datensatz StressAtWork ist die Variable sex enthalten. Hier sind Frauen mit 1 und Männer mit 2 kodiert. Wenn wir diese Variable als Gruppierung verwenden, können wir die Invarianz der Parameter über das Geschlecht untersuchen. Um die Gruppierung in das Modell mit aufzunehmen, können wir in sem einfach dem Argument group den Namen der Gruppierungsvariable übergeben (hierbei sind die "Gänsefüßchen" wichtig!).
fit_sem_IE_TE_MSA <- sem(model_sem_IE_TE, data = StressAtWork, group = "sex") summary(fit_sem_IE_TE_MSA)
Wir sehen, dass im Output nun für jede Gruppe das Modell einzeln geschätzt wurde. Alle Parameter werden sowohl für Frauen als auch für Männer geschätzt. Wir entnehmen,
abbrev(X = fit_sem_IE_TE_MSA, begin = "Number of observations per group", end = "Model Test User Model")
dass insgesamt r sum(StressAtWork$sex == 1) der Probanden Frauen und r sum(StressAtWork$sex == 2) Männer waren. Auch erhalten wir einen globalen sowie einen substichprobenspezifischen Modellfitwert:
abbrev(X = fit_sem_IE_TE_MSA, begin = "Model Test User Model", end = "Parameter Estimates")
Der $\chi^2$-Wert für das gesamte Modell liegt bei r round(fitmeasures(fit_sem_IE_TE_MSA)[3], 3) bei $df=$ r round(fitmeasures(fit_sem_IE_TE_MSA)[4], 3) mit zugehörigem $p$-Wert von r round(fitmeasures(fit_sem_IE_TE_MSA)[5], 3). Demnach verwerfen unsere Daten das Modell nicht. Die Freiheitsgrade sind doppelt so hoch, wie im Ein-Stichprobenfall, da wir alle Parameter für beide Stichproben schätzen müssen. Die $\chi^2$-Werte der beiden Stichproben waren 21.400 für die Frauen und 14.403 für die Männer. Der $\chi^2$-Wert für das gesamte Modell ist also einfach die Summe der subpopulationsspezifischen $\chi^2$-Werte ($\chi^2_{g=1}$ und $\chi^2_{g=2}$, wobei $g=1$ und $g=2$ für die erste und zweite Gruppe steht):
$$\chi^2=\chi^2_{g=1}+\chi^2_{g=2}.$$
In der gesamten Summary fällt uns auf, dass nur in Gruppe 1 die Benennungen der Pfadkoeffizienten mit $a, b$ und $c$ vorkommen und in Gruppe 2 keine Benennung mehr auftaucht.
abbrev(X = fit_sem_IE_TE_MSA, begin = "Group 1", end = "Intercepts") abbrev(X = fit_sem_IE_TE_MSA, begin = "Group 2", end = "Intercepts")
Dies liegt daran, dass Benennungen für Parameter über Gruppen hinweg als Vektor übergeben werden müssen. Somit gehört auch der indirekte Effekt, den wir der Summary entnehmen können, nur zur Gruppe 1, also den Frauen:
abbrev(X = fit_sem_IE_TE_MSA, begin = "Defined Parameters", end = NULL, shift = 1)
Wir müssten also BOEE ~ c(a1, a2)*ZD schreiben, um den Effekt der unabhängigen Variable auf den Mediator in den Gruppen jeweils a1 und a2 zu nennen.
Außerdem fällt uns auf, dass sowohl die Faktorladungen als auch die Pfadkoeffizienten sich kaum über die Gruppen hinweg unterscheiden. Wir wollen den indirekten Effekt auch für die Männer berechnen und erweitern unser Modell entsprechend:
model_sem_IE_TE_MSA_abc <- ' # Messmodelle ZD =~ zd1 + zd2 + zd6 BOEE =~ bo1 + bo6 + bo12 + bo19 # Strukturmodell BOEE ~ c(a1, a2)*ZD BFs ~ c(b1, b2)*BOEE + c(c1,c2)*ZD # Neue Parameter IE1 := a1*b1 TE1 := IE1 + c1 IE2 := a2*b2 TE2 := IE2 + c2 ' fit_sem_IE_TE_MSA_abc <- sem(model_sem_IE_TE_MSA_abc, StressAtWork, group = "sex") summary(fit_sem_IE_TE_MSA_abc)
Nun sind alle Pfadkoeffizienten benannt:
abbrev(X = fit_sem_IE_TE_MSA_abc, begin = "Group 1", end = "Intercepts") abbrev(X = fit_sem_IE_TE_MSA_abc, begin = "Group 2", end = "Intercepts")
Bis auf die hinzukommenden indirekten und totalen Effekte:
abbrev(X = fit_sem_IE_TE_MSA_abc, begin = "Defined Parameters", end = NULL, shift = 1)
hat sich nichts am Output geändert. Wir haben ja auch nur Labels vergeben und neu definierte Parameter hinzugefügt, die allerdings, wie zuvor schon erwähnt, die Modellstruktur (und somit auch die $df$) nicht tangieren.
Die totalen und die indirekten Effekte müssten in einer wissenschaftlichen Untersuchung erneut mithilfe von Bootstrapping inferenzstatistisch geprüft werden. Diesen Schritt lassen wir an dieser Stelle weg.
Invarianzstufen
Mit Invarianz meinen wir die Gleichheit von Parametern über Gruppen hinweg, also bspw., dass es keine Unterschiede über das Geschlecht hinweg gibt. Welche Stufen der Invarianz es gibt, was diese bedeuten und wie wir diese spezifizieren, gucken wir uns im Folgenden an.
So wie wir die indirekten Effekte bestimmt und die Koeffizienten für beide Gruppen benannt haben, lassen sich auch Invarianzen händisch prüfen. Wenn zwei Koeffizienten das selbe Label tragen, werden diese Parameter in den Gruppen auf den gleichen Wert gesetzt. Wir könnten nun für die jeweiligen Invarianzstufen die Parameter händisch gleichsetzen. Dieses ganze Prozedere erscheint recht aufwendig. Allerdings kann so in jedem Schritt überprüft werden, dass die Parameter richtig restringiert wurden. Auch lassen sich so auch leicht partielle Invarianz einbauen, in welchen bspw. nicht alle Faktorladungen über die Gruppen hinweg gleich sind. Außerdem könnten Invarianzen nur für bestimmte Variablen angenommen werden. Diesen kompletten, händischen Prozess sehen Sie in Appendix B. Glücklicherweise enthält das lavaan-Paket aber Möglichkeiten, Invarianzen global zu definieren. Dazu müssen wir lediglich in der Schätzung unserer Modelle in sem das Zusatzargument group.equal spezifizieren. Für partielle Invarianzen gibt es zusätzlich group.partial. Bevor wir mit den Analysen beginnen, sehen Sie in der folgenden Tabelle noch einmal eine Übersicht über die Invarianzstufen. Eine detaillierte Wiederholung dessen, was auch in den inhaltlichen Sitzungen zu den Invarianzstufen behandelt wurde, finden Sie im Appendix D. Die beiden Spalten "Annahme" und "Implikation" sind kumulativ: Invarianzstufen, die weiter unten stehen, enthalten immer auch alle vorherigen Annahmen und erlauben auch immer alle vorherigen Aussagen. Die jeweiligen Einträge einer Zeile sind lediglich für diese Stufe zusätzlich.
Invarianzstufe | Annahme | Implikation ---- | ------- | -------- konfigural | gleiche Modellstruktur | gleiche Konstruktdefinition metrisch (schwach) | gleiche Faktorladungen | latente Variablen haben gleiche Bedeutung; Beziehungen zwischen latenten Variablen vergleichbar skalar (stark) | gleiche Interzepte | mittlere Gruppenunterschiede in manifesten Variablen auf Unterschiede in latenten Mittelwerten zurückführbar; latente Mittelwerte vergleichbar strikt | gleiche Residualvarianzen | Varianzunterschiede in manifesten Variablen auf Varianzunterschiede in latenten Varianzen zurückführbar
Probieren wir dies doch gleich einmal aus (das Modell sollte hierzu keine Parameterbenennungen haben, da diese die gloablen Invarianzeinstellungen in sem überschreiben könnten):
model_sem <- ' # Messmodelle ZD =~ zd1 + zd2 + zd6 BOEE =~ bo1 + bo6 + bo12 + bo19 # Strukturmodell BOEE ~ ZD BFs ~ BOEE + ZD '
Um BFs hier wie eine latente Variable zu behandeln, müssen wir bestimmen, dass das Interzept und die Residualvarianz nicht mit den manifesten Variablen zusammen gleichgesetzt werden, sondern erst mit den latenten Variablen über die Gruppen restringiert werden (bei der Testung der vollständigen Invarianz). Dazu müssen wir zusätzlich die partielle Invarianzeinstellung verwenden: group.partial = c("BFs ~ 1", "BFs ~~BFs"). Hiermit wird bestimmt, welche Koeffizienten nicht von den Invarianzeinstellungen betroffen sein sollen. Auch wenn diese Einstellungen erst bei der skalaren/starken Invarianz ("BFs~1") und bei der strikten Invarianz ("BFs~~BFs") zum tragen kommen, stellen wir diese auch beim konfigural-invarianten und beim metrisch-invarianten (schwach-invarianten) Modell mit ein. Fangen wir mit dem Fitten des konfigural-invarianten Modells an.
Konfigurale Invarianz
Bei der konfiguralen Invarianz geht es darum, dass in beiden Gruppen die gleichen Modelle aufgestellt werden. Gilt diese Annahme bereits nicht, so macht es keinen Sinn, das Modell weiter einzuschränken und Parameter über die Gruppen zu restringieren. Glücklicherweise passt das Modell zu den Daten, in welchem das Modell für das Geschlecht jeweils geschätzt wurde. Hier schauen wir uns dies noch einmal zur Wiederholung und zum Umbenennen des geschätzten Modells an und spezifizieren mit group.equal = c(""), dass keine Parameter über die Gruppen als identisch angenommen werden sollen:
fit_sem_sex_konfigural <- sem(model_sem, data = StressAtWork, group = "sex", group.equal = c(""), group.partial = c("BFs ~ 1", "BFs ~~*BFs")) summary(fit_sem_sex_konfigural, fit.measures = T)
Dem Modell-Fit Teil der Summary entnehmen wir, dass das Modell gut zu den Daten passt:
abbrev(X = fit_sem_sex_konfigural, begin = "Model Test User Model", end = "Parameter Estimates", fit.measures = T)
Der $\chi^2$-Wert ist nicht signifikant und auch die Fit-Indizes CFI, TLI, RMSEA und SRMR sind unauffällig und deuten auf guten Modell-Fit hin. Dies bedeutet, dass wir frohen Mutes das Modell einschränken können, um zu prüfen, welche Invarianz über das Geschlecht hinweg gilt.
Metrische Invarianz
Unter metrischer oder schwacher Invarianz verstehen wir, dass die Faktorladungen ($\lambda$, bzw. $\Lambda$) über die Gruppen hinweg gleich sind. Somit ist der Anteil jedes Items, der auf die latenten Variablen zurückzuführen ist, über die Gruppen hinweg gleichen. Dies ist wichtig, um zu prüfen, ob die Konstrukte über die beiden Gruppen hinweg die gleiche Bedeutung haben. Wir erreichen dies, indem wir group.equal = c("loadings") spezifizieren.
fit_sem_sex_metrisch <- sem(model_sem, data = StressAtWork, group = "sex", group.equal = c("loadings"), group.partial = c("BFs ~ 1", "BFs ~~BFs")) summary(fit_sem_sex_metrisch, fit.measures = T)
Wir entnehmen,
abbrev(X = fit_sem_sex_metrisch, begin = "Model Test User Model", end = "Parameter Estimates", fit.measures = T)
dass das Modell immer noch gut zu den Daten passt. Die Frage ist nur, ob das metrisch-invariante Modell nicht doch vielleicht signifikant schlechter zu den Daten passt als das konfigural-invariante Modell. Bevor wir dieser Frage nachgehen, schauen wir uns noch schnell an, wie Parameter hier per Default benannt werden:
abbrev(X = fit_sem_sex_metrisch, begin = "Group 1", end = "Intercepts") abbrev(X = fit_sem_sex_metrisch, begin = "Group 2", end = "Intercepts")
Wir erkennen, dass "einfach" nur die Parameter durchnummeriert werden, wobei Parameter, die auf 1 restringiert sind, mitgezählt werden, aber nicht ihr eigenes Label erhalten. So heißt $\lambda_{21}^x$, der Ladungskoeffizient von zd2 hier .p2., wobei das p für Parameter steht und die Punkte andeuten, dass es sich hierbei um ein intern vergebenes (also ein durch die Funktion selbst vergebenes) Label handelt. Wollen wir nun wissen, ob sich die Modelle statistisch signifikant von einander underscheiden, können wir wieder den Likelihood-Ratio-Test ($\chi^2$-Differenzentest) heranziehen.
lavTestLRT(fit_sem_sex_metrisch, fit_sem_sex_konfigural)
res_metrisch <- lavTestLRT(fit_sem_sex_metrisch, fit_sem_sex_konfigural) print(res_metrisch)
Die $\chi^2$-Differenz liegt bei r round(res_metrisch[[5]][2], 4) bei $\Delta df=$ r res_metrisch[[6]][2] mit dem zugehörigen $p$-Wert von r round(res_metrisch[[7]][2], 4). Somit können wir weiter von metrischer Invarianz ausgehen. Dies bedeutet, dass sich Unterschiede zwischen Frauen auf der latenten Variable in gleicher Weise in den beobachtbaren Variablen niederschlagen, wie sie es bei Männern tun.
Skalare Invarianz
Als nächsten wollen wir prüfen, ob zusätzlich zu den Faktorladungen auch die Interzepte ($\tau$) über die Gruppen hinweg gleich sind (insgesamt also $\lambda$s und $\tau$s gleich über die Gruppen hinweg). Dazu passen wir erneut unser Modell an. Hierbei ist zu beachten, dass wir nicht das Interzept von BFs über die Gruppen hinweg gleichsetzten, da sich die Interzepte auf die manifesten Variablen beziehen, wir BFs hier allerdings wie eine latente Variable behandeln wollen. Dazu haben wir die group.partial = c("BFs ~ 1", "BFs ~~BFs") Einstellungen verwendet. Eine Besonderheit der skalaren Invarianz ist, dass wir sobald wir die Interzepte über die Gruppen hinweg gleichsetzten, die Freiheitsgrade haben, mit welchen wir die latenten Interzepte von ZD und BOEE schätzen können. Dies ist dann eine Art Effektkodierung, wobei der Mittelwert der einen Gruppe auf 0 gesetzt und in der anderen Gruppe dann die Abweichung zu dieser Gruppe mitgeführt wird. Andernfalls würden wir fälschlicherweise Invarianz der latenten Mittelwerte annehmen, was wir hier noch gar nicht prüfen wollen! Wir schauen uns dies im Output an.
fit_sem_sex_skalar <- sem(model_sem, data = StressAtWork, group = "sex", group.equal = c("loadings", "intercepts"), group.partial = c("BFs ~ 1", "BFs ~~BFs")) summary(fit_sem_sex_skalar, fit.measures = T)
abbrev(X = fit_sem_sex_skalar, begin = "Group 1", end = "Variances") abbrev(X = fit_sem_sex_skalar, begin = "Group 2", end = "Variances")
Wir erkennen, dass nun lavaan sogar nur Zahlen zwischen zwei Punkten als Labels vergibt. Diese Nummer enspricht weiterhin der Parameternummer. Hier greift nun tatsächlich die Einstellung "BFs~1" in group.partial. BFs hat zwei unterschiedlichte Interzepte. Bei ZD und BOEE fällt auf, dass diese in Gruppe 1 auf 0 gesetzt sind (ohne Unsicherheit) und in Gruppe 2 hier eine Effektkodierung durchgeführt wurde: hier wurden die Interzepte geschätzt. Unterscheidet sich dieser Interzept nun von 0, so unterscheiden sich die Gruppe in ihren Interzepten ([möglicherweise bedingten] Mittelwerten). Dazu später mehr!
Nun wollen wir untersuchen, ob das metrisch- und das skalar-invariante Modell sich signifikant in der Modellbeschreibung unterscheiden.
lavTestLRT(fit_sem_sex_skalar, fit_sem_sex_metrisch)
res_skalar <- lavTestLRT(fit_sem_sex_skalar, fit_sem_sex_metrisch) print(res_skalar)
Die $\chi^2$-Differenz ist erneut klein (r round(res_skalar[[5]][2], 3)) und der p-Wert zeigt auch hier an, dass es sich um keine signifikante Verschlechterung des Modells handelt p = r round(res_skalar[[7]][2], 3). Die Null-Hypothese, dass die Interzepte über die Faktorladungen hinaus über das Geschlecht hinweg gleich sind, wird somit nicht verworfen. Dies bedeutet nun, dass Unterschiede im Mittelwert der Items zwischen den beiden Gruppen tatsächlich auch auf Unterschiede der Mittelwerte der latenten Variablen zurückzuführen sind. Das heißt, dass es erst ab dieser Invarianzstufe zulässig ist, Mittelwerte zwischen den Gruppen zu vergleichen.
Strikte Invarianz
Unter strikter Invarianz verstehen wir, dass zusätzlich zu den Faktorladungen und den Interzepten auch die Residualvarianzen ($\theta$) gleich sind (insgesamt also $\lambda$s, $\tau$s und $\theta$s gleich über die Gruppen hinweg). Wir passen entsprechend das Modell an:
fit_sem_sex_strikt <- sem(model_sem, data = StressAtWork, group = "sex", group.equal = c("loadings", "intercepts", "residuals"), group.partial = c("BFs ~ 1", "BFs ~~BFs"))
Hier greift nun tatsächlich die Einstellung "BFs~~BFs" in group.partial. Wir vergleichen das skalar- und das strikt-invariante Modell hinsichtlich der Modellbeschreibung.
lavTestLRT(fit_sem_sex_strikt, fit_sem_sex_skalar)
res_strikt <- lavTestLRT(fit_sem_sex_strikt, fit_sem_sex_skalar) print(res_strikt)
Die $\chi^2$-Differenz ist erneut klein (r round(res_strikt[[5]][2], 3)) und der p-Wert zeigt auch hier an, dass es sich um keine signifikante Verschlechterung des Modells handelt p = r round(res_strikt[[7]][2], 3). Die Null-Hypothese, dass die Fehlervarianzen über die Faktorladungen und Interzepte hinaus über das Geschlecht hinweg gleich sind, wird somit nicht verworfen.
Dies bedeutet nun, dass Unterschiede in der Varianz der manifesten Variablen tatsächlich auf Unterschiede in den Varianzen der latenten Variablen zurückzuführen sind. In anderen Worten, wenn wir z.B. beobachten, dass Männer in den beobachtbaren Verhaltensweisen homogener sind als Frauen, können wir bei dieser Varianzstufe davon ausgehen, dass dies daher kommt, dass Männer im Konstrukt ähnlicher sind und nicht nur daher, dass sie z.B. aufgrund der Formulierung der Fragen genauer gemessen werden konnten.
Vollständige Invarianz
Unter vollständiger Invarianz verstehen wir das Gleichsetzten aller Strukturparameter. Hier werden nun alle Varianzen, Residualvarianzen, ungerichtete und gerichtete Effekte des Strukturmodells über die Gruppen hinweg gleichgesetzt (insgesamt also $\lambda$s, $\tau$s, $\theta$s, $\gamma$s, $\beta$s, $\kappa$s, $\phi$s und $\psi$s gleich über die Gruppen hinweg). Wir passen entsprechend das Modell an. Außerdem müssen wir nun das Interzept und die Residualvarianz von BFs invariant zwischen den Gruppen setzen, was wir erreichen, indem wir die group.partial Option rausnehmen.
fit_sem_sex_voll <- sem(model_sem, data = StressAtWork, group = "sex", group.equal = c("loadings", "intercepts", "residuals", "means", "lv.variances", "lv.covariances", "regressions"))
Wenn wir nun den Modellvergleich zwischen dem strikt invarianten und dem vollständig invarianten Modell durchführen,
lavTestLRT(fit_sem_sex_voll, fit_sem_sex_strikt)
res_voll <- lavTestLRT(fit_sem_sex_voll, fit_sem_sex_strikt) print(res_voll)
erhalten wir diesmal eine große $\chi^2$-Differenz (r round(res_voll[[5]][2], 3)) und der p-Wert zeigt an, dass es sich um eine signifikante Verschlechterung des Modells handelt p = r round(res_voll[[7]][2], 3) $< 0.05$, wenn wir die Restriktion der vollständigen Invarianz in das Modell aufnehmen. Die Null-Hypothese, dass alle Parameter im Modell über das Geschlecht hinweg gleich sind, wird somit verworfen. Dies bedeutet also, dass es Geschlechtsunterschiede in den Beziehungen zwischen den latenten Variablen gibt. Wir schauen uns dies an, indem wir uns den Output der Summary des strikt-invarianten Modells ansehen, da sich hier Pfadkoeffizienten, sowie die Mittelwerte und Varianzen der latenten Variablen (bzw. von BFs, was wir als latente Variable mitführen) noch unterscheiden:
summary(fit_sem_sex_strikt)
abbrev(X = fit_sem_sex_strikt, begin = "Group 1", end = "Variances") abbrev(X = fit_sem_sex_strikt, begin = "Group 2", end = "Variances")
Dem jeweiligen Unterpunkt Regressions in beiden Gruppen entnehmen wir, dass die Pfadkoeffizienten recht ähnlich groß zu sein scheinen. Die Standardfehler der Koeffizienten in beiden Gruppen überlappen sich stark, wenn wir jeweils Estimate $\pm$ Std.Err für einen Koeffizienten rechnen (zur Erinnerung: ein 95% Konfidenzintervall würden wir mit $Est \pm 1.96SE$ erhalten, was noch viel größer wäre und sich diese also "noch stärker" überlappen würde). Schauen wir uns jeweils die Interzepte in den Unterpunkten Intercepts in den beiden Gruppen an, erkennen wir an der Effektkodierung für ZD und BOEE, dass die latenten Interzepte ($\kappa$) in der einen Gruppe auf 0 gesetzt und in der anderen Gruppe frei geschätzt wurden. Das Interzept in Gruppe 2 (den Männern) von Zeitdruck ist signifikant von 0 verschieden, das von emotionaler Erschöpfung nicht. Dies bedeutet, dass sich Männer und Frauen in ihrem latenten Mittelwert von Zeitdruck unterscheiden, da der Mittelwert von Zeitdruck in der Gruppe der Frauen auf 0 gesetzt war und der Mittelwert von Zeitdruck der Männer signifikant von 0 abweicht (Est = r round(parameterEstimates(fit_sem_sex_strikt)[59,7], 3), $p < .05$). Das negative Vorzeichen verrät uns, dass Männer im Durchschnitt weniger Zeitdruck erleben. Auch wenn wir Konfidenzintervalle um die Interzeptschätzung von BFs legen, erhalten wir einen signifikanten Unterschied: die untere Grenze des Konfidenzintervalls in Gruppe 1 liegt bei $2.486-1.960.05 \approx 2.38$ und und die obere Grenze in Gruppe 2 liegt bei $2.206+1.960.069 \approx 2.35$; hier haben wir konservativer "gerundet", um den $\beta$-Fehler zu minimieren; - die Konfidenzintervalle überlappen sich nicht! Diese signifikanten Unterschiede könnte der Grund gewesen sein, dass die vollständige Invarianz verworfen wurde. Um dies genauer zu prüfen, müssten wir sukzessive alle Parameter über die Gruppen gleichsetzen und schauen, für welchen Parameter diese Gleichsetzung zu einer signifikanten Verschlechterung des Modells führt.
Zusammenfassend können wir also nur von strikter Invarianz des Modells über das Geschlecht ausgehen.
Wer davon noch nich genug hat, ist herzlich eingeladen im Appendix C sich die mit der Invarianzidee verwandten Gleichsetzung der Faktorladungen innerhalb einer latenten Variable anzusehen: den Test auf essentielle $\tau$-Äquivalenz. Für weiterführende Literatur, die zum Teil für diesen Abschnitt aufgearbeitet wurde, siehe bspw. Gregorich (2006) - dies ist keine Prüfungsliteratur.
Appendix A {#AppendixA}
Modell-Fit, Stichprobengröße und Fehlspezifikation
Der Likelihood-Ratio-Test ($\chi^2$-Differenzentest) vergleicht die Likelihoods zweier Modelle und somit implizit eigentlich die Kovarianzmatrizen (und Mittelwerte). In Lehrbüchern steht häufig der $\chi^2$-Wert ist stichprobenabhängig und wächst mit der Stichprobengröße, was ebenfalls als Grund für die Fit-Indizes genannt wird. Das ist allerdings nur teilweise richtig, denn der $\chi^2$-Wert ist nur für Modelle stichprobenabhängig, in welchen die $H_0$-Hypothese nicht gilt. In einigen Lehrbüchern steht zudem die Formel für den $\chi^2$-Wert wie folgt: $$F(\hat{\Sigma}M,S) = \log(\hat{\Sigma}_M)-\log(S)+\text{Spur}\left[S\hat{\Sigma}_M^{-1}\right] - p,$$ wobei $\hat{\Sigma}_M$ die modellimplizierte Kovarianzmatrix und $S$ die Kovarianzmatrix der Daten ist und $p$ die Anzahl an beobachteten Variablen. $\text{Spur}$ bezeichnet hierbei die Summe der Diagonalelemente des jeweiligen Objekts (der resultierenden quadratischen Matrix). Die Null-Hypothese besagt: $$H_0:S=\Sigma_M$$ Der $\chi^2$-Wert ergibt sie wie folgt: $$\chi^2:=(n-1)F(\hat{\Sigma}_M,S)$$ Somit wirkt es so, dass der $\chi^2$-Wert zwangsläufig mit der Stichprobengröße wachsen muss. Allerdings ist für wachsendes ($n\to\infty$) $F(\hat{\Sigma}_M,S)\to0$, solange die Null-Hypothese gilt. Dies liegt daran, dass $F(\hat{\Sigma}_M,S)$ gerade die Differenz zwischen den beiden Matrizen quantifiziert und diese Differenz unter der Null-Hypothese im Mittel gegen 0 konvergiert. Diese Differenz geht für steigende Stichprobengröße gegen den Wert 0, wird also kleiner mit steigender Stichprobengröße. Um eine Verteilung als Referenz verwenden zu können (hier: die kritischen Werte der $\chi^2$-Verteilung), ist eine Reskalierung vonnöten. Aus diesem Grund wird $F(\hat{\Sigma}_M,S)$ mit $n-1$ multipliziert und die bekannte $\chi^2$-Verteilung entsteht. Gilt nun eine Alternativ-Hypothese: $$H_1:S\neq\Sigma_M$$ dann konvergiert $F(\hat{\Sigma}_M,S)$ im Mittel nicht mehr gegen Null; es gilt also ($n\to\infty$) $F(\hat{\Sigma}_M,S)\nrightarrow0$, sondern $F(\hat{\Sigma}_M,S)\to d$, wobei $d>0$ gerade die wahre Differenz zwischen den beiden Modellen quantifiziert. Das bedeutet gleichzeitig, dass für den zugehörige mittlere $\chi^2$-Wert unter $H_1$ gilt: $\chi^2{H_1}\to dn \to \infty$, der $\chi^2$-Wert also mit der Stichprobengröße wächst (da $dn$ gerade proportional zu $n$ wächst)! Wir wollen uns dies an folgendem Modell klar machen:
## Example of a Full LISREL model path diagram with the same number of ## exgenous and endogenous variables: # Lambda matrices: LoadingsY <- matrix(c(1,0, 1,0, 0,1, 0,1),4,2, byrow = T) LoadingsX <- as.matrix(c(1,1,1)) # Phi and Psi matrices: Phi <- diag(1, 1, 1) Psi <- diag(2) # Beta matrix: Beta <- matrix(0, 2, 2) Beta[2, 1] <- 1 # Theta matrices: ManVarX <- diag(1, nrow(LoadingsX), nrow(LoadingsX)) ManVarX[2,1] <- 1; ManVarX[1,2] <- 1 ManVarY <- diag(1, nrow(LoadingsY), nrow(LoadingsY)) # Gamma matrix: Gamma <- as.matrix(c(1,1)) # Tau matrices: tauX <- rep(0, nrow(LoadingsX)) tauY <- rep(0, nrow(LoadingsY)) # Alpha and Kappa matrices: LatInts <- rep(0, 2) # Combine model: mod <- lisrelModel(LY = LoadingsY, PS = Psi, BE = Beta, TE = ManVarY, LX = LoadingsX, PH = Phi, GA = Gamma, TD = ManVarX, TY = tauY, TX = tauX, AL = c(0), KA = LatInts) # Plot path diagram: semPaths(mod, as.expression = c("nodes", "edges"), sizeMan = 5, sizeInt = 3, sizeLat = 7, label.prop = 1, layout = "tree2", edge.label.cex = 1)
Als Populationsmodell wählen wir das Folgende:
pop_model_H0 <- ' # Messmodelle Xi1 =~ x1 + 0.7*x2 + 0.6*x3 Eta1 =~ y1 + 0.8*y2 Eta2 =~ y3 + 0.9*y4 # Strukturmodell Eta1 ~ 0.5*Xi1 Eta2 ~ 0.54*Xi1 + 0.4*Eta1 # Fehlerkovarianzen x1 ~~ 0.4*x2 ' set.seed(123456) data <- simulateData(model = pop_model_H0, meanstructure = F, sample.nobs = 200)
Die Werte, die in diesem Modell stehen, symbolisieren die wahren Populationsparameter. Bspw. bedeutet Xi1 =~ x1 + 0.7*x2 + 0.6*x3, dass in der Population gilt: $\lambda_{11}=1,\lambda_{21}=.7$ und $\lambda_{31}=.6$ (wobei $\lambda_{11}=1$ auch der Skalierer ist!). Oder Eta2 ~ 0.54*Xi1 + 0.4*Eta1 steht für: $\eta_2=0.54\xi_1+0.4\eta_1+\zeta_2$ in der Population. x1 ~~ 0.4*x2 symbolisert eine Fehlerkovarianz von 0.4, also $\theta_{21}=.4$.
Wenn ein Modell in dieser Form vorliegt, so kann die simulateData Funktion in lavaan verwendet werden, um diese Modell zu simulieren. Wir übergeben der Funktion dazu das Modell model = pop_model_H0, spezifizieren, dass alle Mittelwerte im Mittel 0 sind meanstructure = F und legen die Stichprobengröße fest sample.nobs = 200. In data liegen nun die simulierten manifesten Variablen (die latenten Variablen werden nicht abgespeichert). Hierbei entscheiden die Kürzel, die wir vergeben (z.B. x1 oder y2) über die Namen in data:
head(data)
print(head(data))
In data liegen nun simulierte Daten mit $n=200$. Wir verwenden das $H_0$ Modell auch, um die Daten zu analysieren (dies ist das Modell von oben ohne jegliche Zahlen, also in dem Format, welches wir bereits kennen!):
model_H0 <- ' # Messmodelle Xi1 =~ x1 + x2 + x3 Eta1 =~ y1 + y2 Eta2 =~ y3 + y4 # Strukturmodell Eta1 ~ Xi1 Eta2 ~ Xi1 + Eta1 # Fehlerkovarianzen x1 ~~ x2 '
Das Pfaddiagramm sieht so aus (hier wurden die Zusatzeinstellungen curve = T, curvePivot = T im semPlot verwenden)
fit_H0 <- sem(model = model_H0, data = data) semPaths(fit_H0, curve = T, curvePivot = T)
Schätzen wir nun das Modell und gucken uns den den $\chi^2$-Wert an.
fit_H0 <- sem(model = model_H0, data = data) summary(fit_H0)
lavaan::summary(fit_H0)
Besonders von Interesse sind der $\chi^2$-Wert, die $df$ und der zugehörige $p$-Wert, die wir auch so erhalten:
fitmeasures(fit_H0, c("chisq", 'df', "pvalue"))
Außerdem wollen wir zwei fehlspezifizierte Modell betrachten. Unter model_H1_kov speichern wir ein Modell, welches, bis auf die fehlende Fehlerkovarianz, äquivalent zu model_H0 ist.
model_H1_kov <- ' # Messmodelle Xi1 =~ x1 + x2 + x3 Eta1 =~ y1 + y2 Eta2 =~ y3 + y4 # Strukturmodell Eta1 ~ Xi1 Eta2 ~ Xi1 + Eta1 '
semPaths(sem(model_H1_kov, data))
Unter model_H1_Strukspeichern wir ein Modell, welches erneut äquivalent zu model_H0ist, bis auf die fehlende gerichtete Beziehung zwischen $\xi_1$ und $\eta_2$.
model_H1_Struk <- ' # Messmodelle Xi1 =~ x1 + x2 + x3 Eta1 =~ y1 + y2 Eta2 =~ y3 + y4 # Strukturmodell Eta1 ~ Xi1 Eta2 ~ Eta1 # Fehlerkovarianzen x1 ~~ x2 '
semPaths(sem(model_H1_Struk, data))
Hierbei ist die Fehlerkovarianz wegzulassen ein "kleiner" Fehler, während eine vollständige Mediation anzunehmen hier zu einem deutlichen Fehler führen sollte, da in die Fehlervarianz nur zwei Variablen involviert sind, während die gerichtete Beziehung zwischen den beiden latenten Variablen $\xi_1$ und $\eta_2$ mindestens alle manifesten Variabel, die Messungen von $\xi_1$ und $\eta_2$ sind, betrifft. Wir gucken uns den Modellfit für alle drei Modelle an:
fit_H1_kov <- sem(model_H1_kov, data) fit_H1_Struk <- sem(model_H1_Struk, data) fitmeasures(fit_H0, c("chisq", 'df', "pvalue")) fitmeasures(fit_H1_kov, c("chisq", 'df', "pvalue")) fitmeasures(fit_H1_Struk, c("chisq", 'df', "pvalue"))
fit_H1_kov <- sem(model_H1_kov, data) fit_H1_Struk <- sem(model_H1_Struk, data) cat("H0:") print(round(fitmeasures(fit_H0, c("chisq", 'df', "pvalue")), 3)) cat("H1: Fehlerkovarianz") print(round(fitmeasures(fit_H1_kov, c("chisq", 'df', "pvalue")), 3)) cat("H1: Vollständige Mediation") print(round(fitmeasures(fit_H1_Struk, c("chisq", 'df', "pvalue")), 3))
Nun wiederholen wir das ganze für eine größere Stichprobengröße von $n=1000$.
set.seed(123456) data <- simulateData(model = pop_model_H0, meanstructure = F, sample.nobs = 1000) fit_H0 <- sem(model = model_H0, data = data) fit_H1_kov <- sem(model_H1_kov, data) fit_H1_Struk <- sem(model_H1_Struk, data)
fitmeasures(fit_H0, c("chisq", 'df', "pvalue")) fitmeasures(fit_H1_kov, c("chisq", 'df', "pvalue")) fitmeasures(fit_H1_Struk, c("chisq", 'df', "pvalue"))
cat("H0:") print(round(fitmeasures(fit_H0, c("chisq", 'df', "pvalue")), 3)) cat("H1: Fehlerkovarianz") print(round(fitmeasures(fit_H1_kov, c("chisq", 'df', "pvalue")), 3)) cat("H1: Vollständige Mediation") print(round(fitmeasures(fit_H1_Struk, c("chisq", 'df', "pvalue")), 3))
Wir sehen, dass das Weglassen der gerichteten Beziehung zu einem größeren mittleren Fehler führt, also zu einem größeren mittleren $\chi^2$-Wert. Gilt die Null-Hypothese, so sollte der mittlere $\chi^2$-Wert bei der Anzahl der $df$ liegen. Nun wollen wir uns die mittleren $\chi^2$-Werte ansehen für verschiedene $n$. Da diese Simulation länger dauern würde, schauen wir uns nur die Ergebnisse an:

Wir sehen deutlich, dass in beiden $H_1$-Bedingungen der mittlere $\chi^2$-Wert mit der Stichprobengröße wächst. Nur in der $H_0$-Bedingung pendelt sich der mittlere $\chi^2$-Wert gerade bei den $df$ ein. Die gestrichelte Linie repräsentiert den $\chi^2_\text{krit}(df=11)$, somit ist ersichtlich, dass beide $H_1$-Modelle ab einer gewissen Stichprobengröße verworfen werden. Nun ist es aber so, dass in der Wissenschaft Daten häufig nicht perfekt vorliegen, sondern kleine Fehlspezifikationen (also Abweichungen von der Theorie, die aber an sich nicht bedeutsam sind) vorhanden sind. Aus diesem Grund wurden Fit-Indizes entwickelt, welche kleine Fehlspezifikationen relativieren sollen. Ansonsten würde das Verhalten dieses Tests die Wissenschaft dazu bringen kleinere Stichproben zu untersuchen, was allerdings das Aufdecken von Effekten erschwert. Um diesem Dilemma aus dem Weg zu gehen, wird auf die Fit-Indizes zurückgegriffen.
Beispielhaft gucken wir uns nun das Verhalten des $CFI$ und des $RMSEA$ an. Die Definition der Fit-Indizes ist: $$CFI:= 1- \frac{\max(\chi^2_t-df_t,0)}{\max(\chi^2_t-df_t,\chi^2_i-df_i,0)},$$ wobei die Subskripts $t$ und $i$ für das $target$-Modell, also unser Modell und das $independence$-Modell stehen, welches keine Beziehung zwischen den manifesten Variablen annimmt (das am schlechtesten passende Modell). Einen Ausdruck wie $\max(\chi^2_t-df_t,0)$ bzw. $\max(\chi^2_t-df_t,\chi^2_i-df_i,0)$ oder einfacher $\max(a,0)$ bzw. $\max(a,b,0)$ lesen wir so: hier wird das Maximum zwischen 2 bzw. 3 Ausdrücken bestimmt und damit weitergerechnet; dadurch, dass einer der 2 bzw. 3 Ausdrücke gerade die 0 ist, bedeutet dies, dass dieses Maximum immer größer oder gleich 0 sein wird ($\ge0$). Der $CFI$ ist ein Vergleich zwischen dem schlechtesten und dem betrachteten Modell. Der mittlere $CFI$ unter der $H_0$-Hypothese sollte bei 1 liegen für große $n$, da für große $n$ der $\chi^2$-Wert im Mittel bei den $df$ liegt und somit $\chi^2_t-df_t=0$, also der Bruch im Mittel bei 0 liegt (somit wird von der 1 im Mittel nichts abgezogen unter der $H_0$). Dies erkennen wir in der Grafik daran, dass im $H_0$-Modell der mittlere $CFI$-Wert gegen 1 geht (dies bedeutet gleichzeitig, dass kleine $CFI$s gerade für einen schlechten Fit sprechen!):

Der $RMSEA$ $$RMSEA:= \sqrt{\max\left(\frac{F(S,\hat{\Sigma}_M)}{df}-\frac{1}{n-1}, 0\right)}.$$ ist die mittlere Abweichung pro Freiheitsgrad kontrolliert für die Stichprobengröße. Der mittlere $RMSEA$ unter der $H_0$-Hypothese sollte bei 0 liegen für große $n$, da für große $n$, $F(S,\hat{\Sigma}_M)$ nahe 0 liegt und damit $\frac{F(S,\hat{\Sigma}_M)}{df}-\frac{1}{n-1} < 0$ also negativ ist. Das Maximum wiederum zwischen einer negativen Zahl und 0 liegt gerade bei 0. In der Grafik erkennen wir dies daran, dass der mittlere $RMSEA$-Wert des $H_0$-Modells gegen 0 geht (dies bedeutet gleichzeitig, dass große $RMSEA$s gerade für einen schlechten Fit sprechen!):

Der $CFI$ sowie der $RMSEA$ pendeln sich für die $H_1$-Modelle gerade bei den "wahren" Abweichungen des Modells unabhängig von der Stichprobengröße ein. Somit ist ersichtlich, dass dies nicht die tatsächlichen Modelle sind, welche den Daten zugrunde liegen, aber zumindest wird quantifiziert, wie stark diese Modelle vom wahren Modell abweichen. Die gestrichelten Linien geben jeweils die Grenze an, ab welchem Wert nicht mehr von einem "guten" Fit gesprochen werden sollte: $CFI < .97$ und $RMSEA > .05$ (Schermelleh-Engel, Moosbrugger, & Müller, 2003). Die Abweichungen von 0 bzw. 1 beim $RMSEA$ sowie bei $CFI$ sind allerdings nur für eines der beiden $H_1$ Modelle "extrem". Nur in diesem würde von keinem guten Fit mehr gesprochen werden: nämlich beim Modell, in welchem fälschlicherweise eine vollständige Mediation angenommen wird (keine gerichtete Beziehung zwischen $\xi_1$ und $\eta_2$).
Wann genau die Fit-Indizes für eine deutliche Abweichung zwischen Daten und Modell sprechen, hängt von vielen Dingen ab. Im Artikel von Schermelleh et al. (2003) wurden Cut-Kriterien für spezifische Modelle entwickelt ($CFI < .97$ und $RMSEA > .05$), welche nicht notwendigerweise auf alle anderen Modelle mit unterschiedlicher Komplexität, Stichprobengröße und Anzahl manifester Variablen, etc. übertragbar sind. Diesem Umstand geschuldet wurde das R-Paket ezCutoffs entwickelt, welches simulationsbasierte Cut-Kriterien speziell angepasst an das vorliegende Modell berechnet. Der ezCutoffs-Funktion müssten wir dazu einfach unser angenommenes Modell sowie die Daten übergeben. Schauen wir uns dem Output zu unserem model_sem Modell, in welchem die Mediation von Zeitdruck über emotionale Erschöpfung auf Burnout modelliert wurde, an:
library(ezCutoffs) ezCutoffs(model = model_sem, data = StressAtWork)
cat("Data Generation |==================================================| 100% elapsed = 11s ~ 0s Model Fitting |==================================================| 100% elapsed = 8s ~ 0s Empirical fit Cutoff (alpha = 0.05) chisq 18.444008456 29.49324699 cfi 0.999650351 0.97982435 tli 0.999456102 0.96861566 rmsea 0.008993101 0.04575465 srmr 0.027500985 0.04021870")
Emprircal fit ist hier gerade der Modelfit, den wir auch in unseren Daten beobachtet haben. Der $\chi^2$-Wert von 18.444 kommt uns auch sehr bekannt vor! In der Spalte Cutoff (alpha = 0.05) steht der zugehörige Cut-Off-Wert, ab welchem von keinem guten Fit mehr zu sprechen wäre. Hierbei ist zu beachten, dass für $TLI$ und $CFI$ kleine Werte (also kleiner als der Cut-Off-Wert) für einen schlechten Fit sprechen, während für $RMSEA$ und $SRMR$ große Werte (also größer als der Cut-Off-Wert) für einen schlechten Fit sprechen.
Appendix B {#AppendixB}
MSA zu Fuß
Wir wollen uns die Invarianztestung auch noch einmal zu Fuß ansehen. Um das ganze abzukürzen, schauen wir uns immer mit fitMeasures nur den $\chi^2$-Wert, die $df$ sowie den $p$-Wert an und vergleichen diese mit den bereits geschätzten Modell aus dem Abschnitt zur MSA.
Konfigurale Invarianz
Wir beginnen mit der Spezifikation des konfigural invarianten Modells. Hier muss nichts gleichgesetzt werden. Wir kopieren also einfach das Modell model_sem:
model_sem <- ' # Messmodelle ZD =~ zd1 + zd2 + zd6 BOEE =~ bo1 + bo6 + bo12 + bo19 # Strukturmodell BOEE ~ ZD BFs ~ BOEE + ZD '
und schätzen dies anschließend. Wir verwenden das Anhängsel 2, um zu zeigen, welches die händisch gleichgesetzte Variante ist:
fit_sem_sex_konfigural <- sem(model_sem, data = StressAtWork, group = "sex", group.equal = c(""), group.partial = c("BFs ~ 1", "BFs ~~*BFs")) fit_sem_sex_konfigural2 <- sem(model_sem, data = StressAtWork, group = "sex") # chi^2, df, p-Wert fitmeasures(fit_sem_sex_konfigural, c("chisq", 'df', "pvalue")) fitmeasures(fit_sem_sex_konfigural2, c("chisq", 'df', "pvalue"))
cat("group.equal:") print(round(fitmeasures(fit_sem_sex_konfigural, c("chisq", 'df', "pvalue")), 3)) cat("zu Fuß/händisch:") print(round(fitmeasures(fit_sem_sex_konfigural2, c("chisq", 'df', "pvalue")), 3))
Wir erkennen keine Unterschiede, was nicht verwunderlich ist. Hier wurde noch nichts gleichgesetzt.
Metrische Invarianz
Wir müssen nun ein neues Modell spezifizieren, in welchem wir allen Faktorladungen ($\lambda$s) jeweils über die Gruppen hinweg das gleiche Label geben. Hierbei verwenden wir l als Label für die Faktorladungen und nummerieren alle Faktorladungen nacheinander durch (wie genau hier vorgegangen wird, ist immer dem Anwender überlassen. Es muss lediglich darauf geachtet werden, nicht mehrfach das gleiche Label für unterschiedliche Parameter zu verwenden, wenn dies nicht explizit gewünscht ist).
model_sem_metrisch <- ' # Messmodelle ZD =~ zd1 + c(l1, l1)*zd2 + c(l2, l2)*zd6 BOEE =~ bo1 + c(l3,l3)*bo6 + c(l4, l4)*bo12 + c(l5, l5)*bo19 # Strukturmodell BOEE ~ ZD BFs ~ BOEE + ZD'
fit_sem_sex_metrisch <- sem(model_sem, data = StressAtWork, group = "sex", group.equal = c("loadings"), group.partial = c("BFs ~ 1", "BFs ~~BFs")) fit_sem_sex_metrisch2 <- sem(model_sem_metrisch, data = StressAtWork, group = "sex") # chi^2, df, p-Wert fitmeasures(fit_sem_sex_metrisch, c("chisq", 'df', "pvalue")) fitmeasures(fit_sem_sex_metrisch2, c("chisq", 'df', "pvalue"))
cat("group.equal:") print(round(fitmeasures(fit_sem_sex_metrisch, c("chisq", 'df', "pvalue")), 3)) cat("zu Fuß/händisch:") print(round(fitmeasures(fit_sem_sex_metrisch2, c("chisq", 'df', "pvalue")), 3))
Erneut erkennen wir keine Unterschiede zwischen den Modellen. Beide Vorgehensweisen kommen zum selben Ergenis!
Skalare Invarianz
Erneut müssen wir ein neues Modell spezifizieren, in welchem wir jeweils allen Faktorladungen und Interzepten ($\lambda$s und $\tau$s) über die Gruppen hinweg das gleiche Label geben. Hierbei verwenden wir tau als Label für die Interzepte und nummerieren alle Interzepte der manifesten Variablen nacheinander durch. Wir fügen außerdem die Effektkodierung ein via ZD ~ c(0, NA)*1 und BOEE ~c(0, NA)*1, womit der latente Mittelwert in einer Gruppe auf 0 gesetzt und in der zweiten frei geschätzt wird und erweitern das Modell model_sem_metrisch zu:
model_sem_skalar <- ' # Messmodelle ZD =~ zd1 + c(l1, l1)*zd2 + c(l2, l2)*zd6 BOEE =~ bo1 + c(l3,l3)*bo6 + c(l4, l4)*bo12 + c(l5, l5)*bo19 zd1 ~ c(tau1, tau1)*1 zd2 ~ c(tau2, tau2)*1 zd6 ~ c(tau3, tau3)*1 bo1 ~ c(tau4, tau4)*1 bo6 ~ c(tau5, tau5)*1 bo12 ~ c(tau6, tau6)*1 bo19 ~ c(tau7, tau7)*1 # Strukturmodell BOEE ~ ZD BFs ~ BOEE + ZD BOEE ~ c(0, NA)*1 ZD ~ c(0, NA)*1 '
fit_sem_sex_skalar <- sem(model_sem, data = StressAtWork, group = "sex", group.equal = c("loadings", "intercepts"), group.partial = c("BFs ~ 1", "BFs ~~BFs")) fit_sem_sex_skalar2 <- sem(model_sem_skalar, data = StressAtWork, group = "sex") # chi^2, df, p-Wert fitmeasures(fit_sem_sex_skalar, c("chisq", 'df', "pvalue")) fitmeasures(fit_sem_sex_skalar2, c("chisq", 'df', "pvalue"))
cat("group.equal:") print(round(fitmeasures(fit_sem_sex_skalar, c("chisq", 'df', "pvalue")), 3)) cat("zu Fuß/händisch:") print(round(fitmeasures(fit_sem_sex_skalar2, c("chisq", 'df', "pvalue")), 3))
Erneut erkennen wir keine Unterschiede zwischen den Modellen. Beide Vorgehensweisen kommen zum selben Ergenis!
Strikte Invarianz
Wieder müssen wir ein neues Modell spezifizieren, in welchem wir jeweils allen Faktorladungen, Interzepten und Fehlervarianzen ($\lambda$s, $\tau$s und $\theta$s) über die Gruppen hinweg das gleiche Label geben. Hierbei verwenden wir t als Label für die Fehlervarianzen und nummerieren alle Fehlervarianzen der manifesten Variablen nacheinander durch. Wir erweitern das Modell model_sem_skalar.
model_sem_strikt <- ' # Messmodelle ZD =~ zd1 + c(l1, l1)*zd2 + c(l2, l2)*zd6 BOEE =~ bo1 + c(l3,l3)*bo6 + c(l4, l4)*bo12 + c(l5, l5)*bo19 zd1 ~ c(tau1, tau1)*1 zd2 ~ c(tau2, tau2)*1 zd6 ~ c(tau3, tau3)*1 bo1 ~ c(tau4, tau4)*1 bo6 ~ c(tau5, tau5)*1 bo12 ~ c(tau6, tau6)*1 bo19 ~ c(tau7, tau7)*1 zd1 ~~ c(t1, t1)*zd1 zd2 ~~ c(t2, t2)*zd2 zd6 ~~ c(t3, t3)*zd6 bo1 ~~ c(t4, t4)*bo1 bo6 ~~ c(t5, t5)*bo6 bo12 ~~ c(t6, t6)*bo12 bo19 ~~ c(t7, t7)*bo19 # Strukturmodell BOEE ~ ZD BFs ~ BOEE + ZD BOEE ~ c(0, NA)*1 ZD ~ c(0, NA)*1 '
fit_sem_sex_strikt <- sem(model_sem, data = StressAtWork, group = "sex", group.equal = c("loadings", "intercepts", "residuals"), group.partial = c("BFs ~ 1", "BFs ~~BFs")) fit_sem_sex_strikt2 <- sem(model_sem_strikt, data = StressAtWork, group = "sex") # chi^2, df, p-Wert fitmeasures(fit_sem_sex_strikt, c("chisq", 'df', "pvalue")) fitmeasures(fit_sem_sex_strikt2, c("chisq", 'df', "pvalue"))
cat("group.equal:") print(round(fitmeasures(fit_sem_sex_strikt, c("chisq", 'df', "pvalue")), 3)) cat("zu Fuß/händisch:") print(round(fitmeasures(fit_sem_sex_strikt2, c("chisq", 'df', "pvalue")), 3))
Erneut erkennen wir keine Unterschiede zwischen den Modellen. Beide Vorgehensweisen kommen zum selben Ergenis!
Vollständige Invarianz
Zum letzten Mal müssen wir ein neues Modell spezifizieren, in welchem wir allen Parametern des Modells (insgesamt also $\lambda$s, $\tau$s, $\theta$s, $\gamma$s, $\beta$s, $\kappa$s, $\phi$s und $\psi$s) über die Gruppen hinweg das gleiche Label geben. Wir müssen die Effektkodierung der latenten Mittelwert wieder herausnehmen und setzten die Mittelwerte in beiden Gruppen auf 0. Als Label verwenden wir wieder die Notation, die wir schon aus den Mediationanalysen kennen (a, b und c). Für die latenten Residualvarianzen führen wir psi als Label ein (hierbei zählen wir weiterhin BFs zu den latenten Variablen). Für die latente Varianz von ZD verwenden wir phi als Label. Für den Mittelwert von BFs verwenden wir ausnahmsweise kappa, da wir BFs, wie zuvor erwähnt, weiter als latente Variable zählen wollen.
model_sem_voll <- ' # Messmodelle ZD =~ zd1 + c(l1, l1)*zd2 + c(l2, l2)*zd6 BOEE =~ bo1 + c(l3,l3)*bo6 + c(l4, l4)*bo12 + c(l5, l5)*bo19 zd1 ~ c(tau1, tau1)*1 zd2 ~ c(tau2, tau2)*1 zd6 ~ c(tau3, tau3)*1 bo1 ~ c(tau4, tau4)*1 bo6 ~ c(tau5, tau5)*1 bo12 ~ c(tau6, tau6)*1 bo19 ~ c(tau7, tau7)*1 zd1 ~~ c(t1, t1)*zd1 zd2 ~~ c(t2, t2)*zd2 zd6 ~~ c(t3, t3)*zd6 bo1 ~~ c(t4, t4)*bo1 bo6 ~~ c(t5, t5)*bo6 bo12 ~~ c(t6, t6)*bo12 bo19 ~~ c(t7, t7)*bo19 # Strukturmodell BOEE ~ c(a, a)*ZD BFs ~ c(b, b)*BOEE + c(c, c)*ZD BOEE ~ c(0, 0)*1 ZD ~ c(0, 0)*1 BFs ~ c(kappa, kappa)*1 ZD ~~ c(phi, phi)*ZD BOEE ~~ c(psi1, psi1)*BOEE BFs ~~ c(psi2, psi2)*BFs '
fit_sem_sex_voll <- sem(model_sem, data = StressAtWork, group = "sex", group.equal = c("loadings", "intercepts", "residuals", "means", "lv.variances", "lv.covariances", "regressions")) fit_sem_sex_voll2 <- sem(model_sem_voll, data = StressAtWork, group = "sex") # chi^2, df, p-Wert fitmeasures(fit_sem_sex_voll, c("chisq", 'df', "pvalue")) fitmeasures(fit_sem_sex_voll2, c("chisq", 'df', "pvalue"))
cat("group.equal:") print(round(fitmeasures(fit_sem_sex_voll, c("chisq", 'df', "pvalue")), 3)) cat("zu Fuß/händisch:") print(round(fitmeasures(fit_sem_sex_voll2, c("chisq", 'df', "pvalue")), 3))
Erneut erkennen wir keine Unterschiede zwischen den Modellen. Beide Vorgehensweisen kommen zum selben Ergenis! Zuvor hatten wir gesehen, dass ein Modellvergleich zwischen der strikten und der vollständigen Invarianz zu Gunsten der strikten Invarianz ausfällt. Somit wird die vollständige Invarianz verworfen und wir entscheiden uns final für die strikte Invarianz über das Geschlecht.
Appendix C {#AppendixC}
Essentielle $\tau$-Äquivalenz
Das Pfadanalysemodell auf Basis von Skalenmittelwerten hatte als Voraussetzung, dass alle Messunger der selben latenten Variable essentiell $\tau$-äquivalent sind, also alle $\lambda$s gleich sind pro latenter Variable. Dies können wir in unserem Modell prüfen, indem wir den Faktorladungen Namen geben, analog zur Bennenung der gerichteten Beziehungen zwischen den latenten Variablen, die wir zur Berechnung des indirekten etc. Effekts gebraucht haben. Das letzte Modell sah so aus:
model_sem_IE_TE <- ' # Messmodelle ZD =~ zd1 + zd2 + zd6 BOEE =~ bo1 + bo6 + bo12 + bo19 # Strukturmodell BOEE ~ a*ZD BFs ~ b*BOEE + c*ZD # Neue Parameter IE := a*b TE := IE + c '
Wenn wir weiterhin die Defaulteinstellungen der Funktion sem nutzen wollen (1. Faktorladung = 1, etc.), dann müssen wir bei der Bennenung der Faktorladungen etwas aufpassen. Wenn nun alle Faktorladungen gleich sein sollen, so können wir einfach den Präfix 1* vor alle Faktorladungen schreiben. Dies kommt zum äquivalenten Ergebnis wie das Schreiben von bspw. l1* vor alle Messungen der selben latenten Variable (z.B. Zeitdruck): alle Faktorladungen würden auch so per Default auf 1 gesetzt und würden das Label l1 tragen. Das Vergeben von dem Label l1 ist der allgemeinere Ansatz, weswegen wir diesen hier verwenden wollen. Achtung, wenn wir hier die gleichen Labels über die latenten Variablen (auch l1) verwendet hätten, so wären die Faktorladungen gleich mit denen des Zeitdruck, was wir allerdings nicht wollen. Die essentielle $\tau$-Äquivalenzannahme gilt nur pro latenter Variable.
model_sem_IE_TE_tau <- ' # Messmodelle ZD =~ zd1 + zd2 + zd6 BOEE =~ bo1 + bo6 + bo12 + bo19 # Strukturmodell BOEE ~ a*ZD BFs ~ b*BOEE + c*ZD # Neue Parameter IE := a*b TE := IE + c ' fit_sem_IE_TE_tau <- sem(model_sem_IE_TE_tau, StressAtWork) summary(fit_sem_IE_TE_tau, fit.measures = T)
model_sem_IE_TE_tau <- ' # Messmodelle ZD =~ l1*zd1 + l1*zd2 + ...*zd6 BOEE =~ l2*bo1 + ...*bo6 + bo12 + bo19 # Strukturmodell BOEE ~ a*ZD BFs ~ b*BOEE + c*ZD # Neue Parameter IE := a*b TE := IE + c ' fit_sem_IE_TE_tau <- sem(model_sem_IE_TE_tau, StressAtWork) summary(fit_sem_IE_TE_tau, fit.measures = T)
model_sem_IE_TE_tau <- ' # Messmodelle ZD =~ l1*zd1 + l1*zd2 + l1*zd6 BOEE =~ l2*bo1 + l2*bo6 + l2*bo12 + l2*bo19 # Strukturmodell BOEE ~ a*ZD BFs ~ b*BOEE + c*ZD # Neue Parameter IE := a*b TE := IE + c ' fit_sem_IE_TE_tau <- sem(model_sem_IE_TE_tau, StressAtWork)
## Skalen bilden StressAtWork$ZDs <- rowMeans(StressAtWork[,paste0("zd",c(1, 2, 6))]) StressAtWork$BOEEs <- rowMeans(StressAtWork[,paste0("bo",c(1, 6, 12, 19))]) StressAtWork$BFs <- rowMeans(StressAtWork[,paste0("bf",1:20)]) ## Modell spezifizieren model_paths <- ' BOEEs ~ ZDs BFs ~ BOEEs + ZDs ' ## Modell fitten fit_paths <- sem(model_paths, data = StressAtWork) #### SEM: # Modell model_sem_IE_TE <- ' # Messmodelle ZD =~ zd1 + zd2 + zd6 BOEE =~ bo1 + bo6 + bo12 + bo19 # Strukturmodell BOEE ~ a*ZD BFs ~ b*BOEE + c*ZD # Neue Parameter IE := a*b TE := IE + c ' fit_sem_IE_TE <- sem(model_sem_IE_TE, StressAtWork) ## Modell für essentielle tau-Äquvivalenz model_sem_IE_TE_tau <- ' # Messmodelle ZD =~ l1*zd1 + l1*zd2 + l1*zd6 BOEE =~ l2*bo1 + l2*bo6 + l2*bo12 + l2*bo19 # Strukturmodell BOEE ~ a*ZD BFs ~ b*BOEE + c*ZD # Neue Parameter IE := a*b TE := IE + c ' fit_sem_IE_TE_tau <- sem(model_sem_IE_TE_tau, StressAtWork)
Sollten Sie alles richtig formuliert haben (und die Zusatzeinstellungen in der summary als fit.measures = T, rsq = T gewählt haben), so sollten Sie folgenden Modellfit erhalten:
abbrev(fit_sem_IE_TE_tau, begin = "Model Test User Model", end = "Parameter Estimates", fit.measures = T, rsq = T)
Sowie folgenden Ergebnisse für die Messmodelle:
abbrev(fit_sem_IE_TE_tau, begin = "Latent Variables", end = "Regressions")
Wir erkennen durch die gleiche Bennenung, dass es sich hier um restringierte Koeffizienten handelt. Auch erkennen wir, dass die Faktorladungen von ZD und BOEE alle gleich heißen und auf 1 gesetzt wurden.
Dem Modellfit ist zu entnehmen, dass das Modell immer noch zu den Daten passt: der $\chi^2$-Wert liegt bei r round(fitmeasures(fit_sem_IE_TE_tau)[3], 3) bei $df=$ r round(fitmeasures(fit_sem_IE_TE_tau)[4], 3) mit zugehörigem $p$-Wert von r round(fitmeasures(fit_sem_IE_TE_tau)[5], 3). Demnach verwerfen unsere Daten das Modell nicht. Außerdem sind die $df$ genau um r round(fitmeasures(fit_sem_IE_TE_tau)[4] - fitmeasures(fit_sem_IE_TE)[4], 3) größer als im unrestringierten Modell (ohne die Annahme der essentiellen $\tau$-Äquivalenz pro latenter Variable). Wir können diese Differrenz leicht prüfen: es mussten 2 Faktorladungen weniger für ZD geschätzt werden und 3 weniger für BOEE - aus diesem Grund unterscheiden sich die $df$ gerade um 5! Da das Modell nicht durch die Daten verworfen wird, müssen wir uns auch nicht unbedingt die Fit-Indizes ansehen. Diese sind hier nämlich auch unauffällig. Ob sie überhaupt im Bezug zu diesem Modell interpretiert werden sollten, wurde im Appendix A näher beleuchtet.
Auch wenn die Daten unser Modell nicht verwerfen, können wir die beiden Modelle mit und ohne essentieller $\tau$-Äquivalenzannahme inferenzstatistisch miteinander vergleichen. Dazu verwenden wir wieder die Funktion lavTestLRT und führen also einen Likelihood-Ratio-Test ($\chi^2$-Differenzentest) durch. Probieren Sie den Modellvergleichstest selbst aus:
## Skalen bilden StressAtWork$ZDs <- rowMeans(StressAtWork[,paste0("zd",c(1, 2, 6))]) StressAtWork$BOEEs <- rowMeans(StressAtWork[,paste0("bo",c(1, 6, 12, 19))]) StressAtWork$BFs <- rowMeans(StressAtWork[,paste0("bf",1:20)]) ## Modell spezifizieren model_paths <- ' BOEEs ~ ZDs BFs ~ BOEEs + ZDs ' ## Modell fitten fit_paths <- sem(model_paths, data = StressAtWork) #### SEM: # Modell model_sem_IE_TE <- ' # Messmodelle ZD =~ zd1 + zd2 + zd6 BOEE =~ bo1 + bo6 + bo12 + bo19 # Strukturmodell BOEE ~ a*ZD BFs ~ b*BOEE + c*ZD # Neue Parameter IE := a*b TE := IE + c ' fit_sem_IE_TE <- sem(model_sem_IE_TE, StressAtWork) ## Modell für essentielle tau-Äquvivalenz model_sem_IE_TE_tau <- ' # Messmodelle ZD =~ (l1)*zd1 + (l1)*zd2 + (l1)*zd6 BOEE =~ (l2)*bo1 + (l2)*bo6 + (l2)*bo12 + (l2)*bo19 # Strukturmodell BOEE ~ a*ZD BFs ~ b*BOEE + c*ZD # Neue Parameter IE := a*b TE := IE + c ' fit_sem_IE_TE_tau <- sem(model_sem_IE_TE_tau, StressAtWork) # Begin grading: res <- lavTestLRT(fit_sem_IE_TE_tau, fit_sem_IE_TE) # 2 Faktoren passen besser zu den Daten! sol2 <- lavTestLRT(fit_sem_IE_TE, fit_sem_IE_TE_tau) # 2 Faktoren passen besser zu den Daten! grade_result( fail_if(~ (!identical(.result, res) & !identical(.result, sol2)), 'Der Befehl lavTestLRT muss in diesem Zusammenhang zwei Argumente entgegennehmen: die zwei Objekte der ML-Schätzung unserer SEM-Modelle, welche wir zuvor erstellt haben! Das eine Modell ist jenes mit der Annahme der essentiellen tau-Äquivalenz, das andere ist das unrestringierte Modell. Die Modelle hießen `fit_sem_IE_TE` und `fit_sem_IE_TE_tau`.'), pass_if(~ identical(.result, res) | identical(.result, sol2)), correct = 'Sehr gut! Dies ist der Modellvergleich des Modells mit der Annahme der essentiellen tau-Äquivalenz, das andere ist das unrestringierte Modell!', incorrect = 'Leider falsch.', glue_correct = '{.correct}', glue_incorrect = '{.incorrect} {.message}')
resLRT_tau <- lavTestLRT(fit_sem_IE_TE_tau, fit_sem_IE_TE) # 2 Faktoren passen besser zu den Daten!
Dem Modellvergleich ist zu entnehmen, dass das $\tau$-Äquivalenz-Modell signifikant schlechter zu den Daten passt. Der $p$-Wert liegt bei r round(resLRT_tau[[7]][2], 3) und ist somit $<.05$. Auch die Differenz der $df$ lässt sich diesem Output entnehmen. Dies zeigt erneut, dass die Annahme der essentiellen $\tau$-Äquivalenz, auf welcher das Pfadanalysemodell untersucht wurde, auf dem 5% Signifikanzniveau nicht haltbar ist.
Auf die gleiche Art und Weise können wir auch Invarianz über die Zeit oder über Gruppen hinweg untersuchen. Über die Zeit würden wir zwei latente Variablen formulieren, welche einmal $t_1$ und einmal $t_2$ symbolisiert. Die Annahme, dass zu beiden Zeitpunkte die Messmodelle gleich sind, ist tatsächlich in vielen Situation sinnvoll.
Appendix D {#AppendixD}
Invarianzstufen
Die Invarianzstufen sind nach Einschränkungen der Modellparameter sortiert und werden auch (fast) immer in dieser Reihenfolge sukzessive getestet: konfigurale, metrische (schwache), skalare (starke), strikte und vollständige Invarianz. Wir gehen so vor, wie dies per Default im R-Paket lavaan durchgeführt wird. Wir gehen hierzu davon aus, dass die Skalierung für die Varianzen auf den ersten Faktorladungen ($\lambda=1$) liegt und dass die Skalierung für die Mittelwerte (Interzepte) auf dem latenten Mittelwert liegt ($\kappa=0$).
Die erste und am wenigsten restriktive Invarianzstufe ist die konfigurale Invarianz. Unter konfiguraler Invarianz verstehen wir die Gleichheit der Modelle in den Gruppen - oder salopp gesprochen: gilt konfigurale Invarianz, dann gilt in beiden Gruppen die selbe Faktorstruktur (bzw. die gleiche Konstruktdefinition). Die folgende Grafik soll dies für ein einfaches CFA Modell veranschaulichen, welches auch die oben erwähnten Skalierer enthält. In Schwarz dargestellt sind Invarianzen über die Gruppen hinweg (natürlich ist das Modell, das wir hier behandeln, wesentlich komplexer, aber das angegeben Modell stellt die wichtigsten Aspekte der Gleichsetzung dar!):

Ob diese Invarianzstufe hält, können wir durch die Modellpassung des Multigruppenmodells aus dem letzten Abschnitt prüfen. Weil wir das Modell aufgrund der sehr guten Passung nicht verwerfen müssen, gibt uns das den Hinweis, dass zumindest die Faktorstruktur in beiden Gruppen gleich ist (als konfigurale Invarianz hält). Wenn wir z.B. durch Fehlpassung feststellen würden, dass in einer der beiden Gruppen eine zusätzliche Residualkorrelation oder eine Querladung aufgenommen werden muss, müssten wir die Annahme der konfiguralen Messinvarianz verwerfen.
Um die weiteren Invarianzstufen und deren Implikationen besser zu verstehen, schauen wir uns noch einmal die modellimplizierte Varianz und die modellimplizierte Erwartung (den modell implizierten Mittelwert) einer manifesten Variable $X$ an. Wir verwenden $^{(g)}$ um Unterschiede zwischen Gruppen darzustellen, wobei dies als Platzhalter für bspw. die Gruppennummer fungiert. Lassen wir dies weg, so gehen wir davon aus, dass der Parameter gleich ist über die Gruppen hinweg. Alle anderen Indizes lassen wir weg, um das ganze übersichtlicher zu gestalten.
Eine einfache modellimplizierte Gleichung einer Messung $X$ ist:
$$X^{(g)}=\tau^{(g)}+\lambda^{(g)}\xi^{(g)}+\delta^{(g)}.$$
$\tau$ ist das Interzept und $\lambda$ ist die Faktorladung. Die Varianz von $X$ ist folglich:
$$\mathbb{V}ar\left[X^{(g)}\right]=\left(\lambda^{(g)}\right)^2\phi^{(g)}+\theta^{(g)}.$$
$\phi$ ist die Varianz der latenten Variable, $\xi$, und $\theta$ ist die Residualvarianz, also die Varianz von $\delta$. Wenn Sie sich nun fragen, wieso wir $\lambda$ hier quadrieren müssen, dann überlegen Sie sich folgendes Beispiel anhand der empirischen Stichprobenvarianz von einer Variable $Y$. Diese berechnen wir so:
$$\hat{\mathbb{V}ar}[Y] = \frac{1}{n}\sum_{i=1}^n(Y_i-\bar{Y})^2,$$
wobei $\bar{Y}$ der Mittelwert von $Y$ ist. Wenn wir nun alle Einträge von $Y$ mit einer Konstanten multiplizieren, also für jede Person $i$ das Produkt $aY_i$ berechnen (z.B. $a=10$) und die Varianz bestimmen (der Mittelwert von $aY$ ist einfach $a\bar{Y}$), dann ergibt sich:
$$\hat{\mathbb{V}ar}[aY] = \frac{1}{n}\sum_{i=1}^n(aY_i-a\bar{Y})^2=\frac{1}{n}\sum_{i=1}^n\big(a(Y_i-a\bar{Y})\big)^2=\frac{1}{n}\sum_{i=1}^na^2(Y_i-\bar{Y})^2=a^2\frac{1}{n}\sum_{i=1}^n(Y_i-\bar{Y})^2=a^2\hat{\mathbb{V}ar}[Y],$$
Da $a$ in der Klammer steht, die quadriert wird, muss natürlich $a$ quadriert werden. Da auch $a^2$ eine Konstante ist, kann sie vor die Summe gezogen werden. Daraus wird dann ersichtlich, dass die Varianz des Produktes einer Variablen mit einer Konstanten gleich der Konstanten zum Quadrat multipliziert mit der Varianz der Variablen ist. Der Mittelwert von $X$ ergibt sich als: $$\mathbb{E}\left[X^{(g)}\right]=\tau^{(g)}+\lambda^{(g)}\kappa^{(g)},$$ wobei $\kappa$ das Interzept (den Mittelwert) von $\xi$ darstellt. Da $X$ von so vielen Parametern abhängt, ist ohne weitere Annahmen (von Invarianzen der Parameter über die Gruppen hinweg) nicht zu sagen, ob Unterschiede zwischen Gruppen bspw. im Mittelwert von $X$ auf Unterschiede in der latenten Variable oder auf Unterschiede in der Messung zurückzuführen sind. Ähnlich sieht es für die Varianz aus.
Die nächste Invarianzstufe ist die metrische oder schwache Invarianz. Nehmen wir metrische Invarianz an, so gehen wir davon aus, dass die Faktorladungen ($\lambda$) über die Gruppen hinweg gleich sind. Dies impliziert, dass die latenten Variablen über die Gruppen hinweg die gleiche Bedeutung haben. Die latenten Variablen lassen sich also inhaltlich gleich interpretieren und die Beziehungen zwischen ihnen sind vergleichbar. Dies ist sehr wichtig, wenn wir bspw. Fragebögen oder Tests über Gruppen (z.B. Länder oder das Geschlecht) hinweg vergleichen wollen. In der Grafik sind nun alle $\lambda$s schwarz (anstatt blau oder rot):

Die Varianz von $X$ vereinfacht sich zu (Weglassen von $^{(g)}$ symbolisiert Gleichheit über die Gruppen hinweg): $$\mathbb{V}ar\left[X^{(g)}\right]=\lambda^2\phi^{(g)}+\theta^{(g)}.$$
Hierbei ist es so, dass $\lambda^2\phi^{(g)}$ gerade die Systematik oder den wahren Anteil in der Varianz von $X$ beschreibt. Falls metrische Invarianz gilt, so lassen sich also Unterschiede in den wahren Anteilen der Varianzen der Beobachtungen auf Unterschiede in den latenten Varianzen zurückführen (Achtung: dies ist nicht das Gleiche wie Gleichheit der Reliabilitäten, was anteilig die Systematik in $X$ an der gesamten Varianz von $X$ beschreibt - hierfür müssten ebenfalls $\theta$ gleich sein). Dies bedeutet, wenn wir metrische Invarianz nicht verwerfen (also weiter davon ausgehen, dass metrische Invarianz gilt), dass wir durch weitere Modellrestriktionen die latenten Varianzen auf Gleichheit prüfen können. Dies ist nicht zulässig, wenn metrische Invarianz nicht gilt, also die Faktorladungen sich über die Gruppen hinweg unterscheiden. Spezifizieren wir group.equal = c("loadings"), so wird das metrisch-invariante Modell geschätzt.
Unter skalarer oder starker Invarianz verstehen wir die nächste Invarianzstufe, welche zusätzlich zu den Faktorladungen auch noch die Interzepte ($\tau$) als invariant über die Gruppen hinweg annimmt. Der Mittelwert von $X$ vereinfacht sich zu:
$$\mathbb{E}\left[X^{(g)}\right]=\tau+\lambda\kappa^{(g)},$$
wobei nun ersichtlich ist, dass nur noch $\kappa$ über die Gruppen hinweg variiert (wegen $^{(g)}$). Dies bedeutet also, dass unter Annahme der skalaren Invarianz Unterschiede in den Mittelwerten der Beobachtungen auf Unterschiede in den Mittelwerten der latenten Variablen zurückzuführen sind. Um dies zu prüfen, würden überlicherweise über die skalare Invarianz hinaus auch noch $\kappa$ über die Gruppen hinweg gleichgesetzt werden. Der Likelihood-Ratio-Test ($\chi^2$-Differenzentest) zwischen dem skalar-invarianten Modell und dem zusätzlich $\kappa$-invarianten Modell entscheidet dann darüber, ob die latenten Mittelwerte sich über die Gruppen hinweg unterscheiden. Wir schätzen das skalar-invariante Modell durch group.equal = c("loadings", "intercepts"). Da auch für die Mittelwertsstruktur Skalierer gesetzt werden müssen und diese üblicherweise auf den latenten Mittelwerten liegen ($\kappa=0$), muss dies bei der Invarianztestung berücksichtigt werden. Wenn wir die Skalierer auf den latenten Mittelwerten lassen und die Interzept der manifesten Variablen gleichsetzen, so nehmen wir damit auch implizit an, dass die latenten Mittelwerte über die Gruppen hinweg gleich sind. Um diesem Problem aus dem Weg zu gehen, stellt die sem-Funktion klugerweise ein, dass nur in der ersten Gruppe die latenten Mittelwerte/Interzepte ($\kappa$) auf 0 gesetzt werden, alle übrigen werden frei geschätzt. Dies haben wir uns an entsprechender Stelle im Output unserer Modellschäzung angesehen! In der Grafik sind nun alle $\lambda$s und $\tau$s schwarz (anstatt blau oder rot). Außerdem ist die Effektkodierung der $\kappa$s zu sehen: diese bedeutet, dass der erste latente Mittelwert auf 0 restringiert wird, während der zweite frei geschätzt wird und somit die Abweichungen (den Effekt) quantifiziert (beide sind in der Gruppenfarbe dargestellt):

Setzen wir zusätzlich zu Faktorladungen und Interzepten auch noch die Residualvarianzen ($\theta$) gleich über die Gruppen, so sprechen wir von strikter Invarianz. Gilt diese Invarianzbedingung, so lassen sich Unterschiede in den beobachteten Varianzen ausschließlich auf Unterschiede in den latenten Varianzen (also die Varianzen der latenten Variablen) zurückführen. Dies lässt sich der Varianzformel entnehmen: $$\mathbb{V}ar\left[X^{(g)}\right]=\lambda^2\phi^{(g)}+\theta.$$ Nur noch $\phi$ trägt auf der rechten Seite ein $^{(g)}$. Somit sind nur noch Unterschiede in $\phi$ verantwortlich für Unterschiede in $\mathbb{V}ar\left[X\right]$. In der Grafik sind nun alle $\lambda$s, $\tau$s und $\theta$s schwarz (anstatt blau oder rot):

group.equal = c("loadings", "intercepts", "residuals").
Gehen wir von vollständiger Invarianz aus, so sind alle Parameter über die Gruppen hinweg gleich. Es gibt also keinerlei Unterschiede mehr zwischen den Gruppen. Die $\phi$s und auch die $\kappa$s müssen wieder auf den gleichen Wert über die Gruppen hinweg gesetzt werden. Die Variablen selbst ($X$, $\xi$ und $\delta$) bleiben farbig, da wir nur Parameter über die Gruppen hinweg gleichsetzen. Die Grafik der vollständigen Invarianz sieht folgendermaßen aus:

Das vollständig-invariante Modell erhalten wir mit group.equal = c("loadings", "intercepts", "residuals", "means", "lv.variances", "lv.covariances", "regressions"). Hierbei restringiert "means" die latenten Mittelwerte ($\kappa$), "lv.variances" die latenten (Residual-)Varianzen ($\phi$s und $\psi$s exogen und endogen) und "regressions" die Strukturpfadkoeffizienten ($\gamma$s und $\beta$s). Hätten wir Residualkovarianzen bei den manifesten oder latenten Variablen gehabt, so müssten wir diese auch mit "residual.covariances" sowie mit "lv.covariances" restringieren.
Die Invarianzstufen lassen sich nach der Anzahl der zu schätzenden Parameter oder den $df$ sortieren. Im konfigural-invarianten Modell müssen am meisten Parameter geschätzt werden, es besitzt also die wenigsten $df$, während im vollständig-invarianten Modell die wenigsten Parameter geschätzt werden müssen, weswegen dieses auch die meisten $df$ hat. Die anderen Modelle liegen wie folgt dazwischen (je weiter links, desto weniger restriktiv ist das Modell, desto mehr Parameter sind zu schätzen und desto weniger $df$ gibt es): $$\textbf{konfigural}\quad<\quad\textbf{metrisch/schwach}\quad<\quad\textbf{skalar/stark}\quad<\quad\textbf{strikt}\quad<\quad\textbf{vollständig}.$$
Likelihood-Ratio-Tests ($\chi^2$-Differenzentests) werden herangezogen, um zu prüfen, ob die Invarianzeinschränkungen das Modell signifikant hinsichtlich der Passung zu den Daten verschlechtern.
Literatur
Eid, M., Gollwitzer, M., & Schmitt, M. (2017). Statistik und Forschungsmethoden (5. Auflage, 1. Auflage: 2010). Weinheim: Beltz.
Gregorich, S. E. (2006). Do self-report instruments allow meaningful comparisons across diverse population groups? Testing measurement invariance using the confirmatory factor analysis framework. Medical Care, 44(11), 78-94.
Schermelleh-Engel, K., Moosbrugger, H., & Müller, H. (2003). Evaluation the fit of structural equation models: tests of significance and descriptive goodness-of-fit measures. Methods of Psychological Research Online, 8(2), 23-74.
Inhaltliche Literatur
Büssing, A., & Perrar, K.-M. (1992). Die Messung von Burnout. Untersuchung einer deutschen Fassung des Maslach Burnout Inventory (MBI-D) [The measurement of Burnout. The study of a German version of the Maslach Burnout Inventory (MBI-D)]. Diagnostica, 38, 328 – 353.
Maslach, C., & Jackson, S.E. (1986). Maslach Burnout Inventory (Vol. 2). Palo Alto, CA: Consulting Psychologists Press.
Mohr, G. (1986). Die Erfassung psychischer Befindensbeeinträchti- gungen bei Arbeitern [Assessment of impaired psychological well-being in industrial workers]. Frankfurt am Main, Fermany: Lang.
Semmer, N. K., Zapf, D., & Dunckel, H. (1999). Instrument zur Stressbezogenen Tätigkeitsanalyse (ISTA) [Instrument for stress-oriented task analysis (ISTA)]. In H. Dunkel (Ed.), Handbuch psychologischer Arbeitsanalyseverfahren (pp. 179 – 204). Zürich, Switzerland: vdf Hochschulverlag an der ETH.
- Blau hinterlegte Autorenangaben führen Sie direkt zur universitätsinternen Ressource.
Add the following code to your website.
For more information on customizing the embed code, read Embedding Snippets.
